利用者:YasuakiH/note
英語版 Molecular modelling の2021-02-18T10:39:06(UTC)版から翻訳のため転記
分子モデリング-Molecular_modelling.txt

分子モデリング(英: molecular modelling)は、分子の挙動をモデル化または模倣するために用いられる、理論的および計算上のさまざまな手法を指す[1]。これらの手法は、計算化学、医薬品設計、計算生物学、材料科学の分野で、小さな化学系から大きな生体分子や物質の集合体まで、さまざまな分子系の研究に用いられている。最も簡単な計算は手作業でできるが、合理的サイズの系の分子モデリングを行うには、必然的にコンピュータが必要になる。分子モデリング手法における共通の特徴は、分子系を原子レベルで記述することである。これには、原子を最小の個々の単位として扱う方法(分子力学的アプローチ)、または陽子や中性子をそのクォーク、反クォークとグルーオン、および電子をその光子とともに明示的にモデル化する方法(量子化学的アプローチ)などがある。
分子力学
分子力学は、分子モデリングの一つの側面であり、モデルの背後にある物理的基礎を記述するために古典力学(ニュートン力学)を使用する。分子モデルでは一般的に原子(原子核と電子を合わせたもの)を関連する質量を持つ点電荷として記述する。隣接する原子間の相互作用は、ばねのような相互作用(化学結合を表す)とファンデルワールス力によって表現される。後者の表現には、レナード-ジョーンズ・ポテンシャルが一般的に使用される。静電相互作用はクーロンの法則に基づいて計算される。原子には直交座標または内部座標で座標が割り当てられ、動的なシミュレーションでは速度を割り当てることもできる。原子の速度は、巨視的な量である系の温度に関連している。この集合的な数式はポテンシャル関数と呼ばれ、系の内部エネルギー(U)、つまり位置エネルギーと運動エネルギーの合計に等しい熱力学的な量に関連している。位置エネルギーを最小化する手法はエネルギー最小化法(最急降下法や共役勾配法など)と呼ばれ、時間の伝播を伴う系の挙動をモデル化する手法は分子動力学法と呼ばれる。
ポテンシャル関数(potential function)と呼ばれるこの関数は、分子ポテンシャルエネルギーを、結合長、結合角、およびねじれ角の平衡値からの偏差を表すエネルギー項と、ファンデルワールスや静電的相互作用を表す非結合原子対項の合計として計算する。平衡結合長、結合角、部分電荷量、力定数、およびファンデルワールスパラメータからなる一連のパラメータは総称して力場(英: force field)と呼ばれる。分子力学のさまざまな実装では、ポテンシャル関数にさまざまな数式とパラメータが使用される[2]。現在使用されている一般的な力場は、化学理論、実験的参照データ、および高レベル量子計算を用いて開発されている。エネルギー最小化(energy minimization)と呼ばれるこの方法は、すべての原子のエネルギー勾配がゼロになる位置、つまり局所的なエネルギー最小値を見つけるために使用される。低エネルギー状態はより安定しており、化学的および生物学的プロセスにおいて大きな役割を果たしているため、一般に研究されている。
分子動力学
分子動力学シミュレーションは、系の挙動を時間の関数として計算する。これには、ニュートンの運動法則、主に第2法則 を解くことが含まれる。ニュートンの運動法則を、さまざまな積分アルゴリズムを用いて積分することで、空間と時間の中での原子の軌跡を得ることができる。原子にかかる力は、位置エネルギー関数の負の勾配として定義される。エネルギー最小化法は、類似した系の状態間を比較するための静止画を得るのに有効であるが、分子動力学法は、温度効果を内在させた動的プロセスに関する情報を提供する。
分子モデルの変数
溶媒
分子のモデリングは、真空中、または水などの溶媒の存在下で行うことができる。真空中の系のシミュレーションは気相シミュレーション(gas-phase simulation)と呼ばれ、溶媒分子の存在を含むシミュレーションは明示的溶媒シミュレーション(explicit solvent simulation)と呼ばれる。また、経験的な数式を用いて溶媒の影響を推定するタイプのシミュレーションもあり、これは非明示的溶媒和シミュレーション(implicit solvation simulation)と呼ばれている。
座標表現
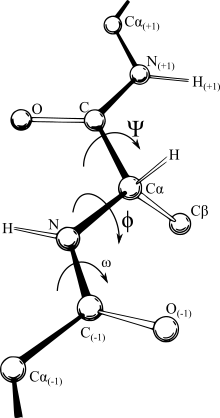
ほとんどの力場は距離に依存するため、これらの直交座標の表現が最も便利である。しかし、特定の原子間で発生する結合は比較的堅固な性質で、本質的には分子の意味を定義しているため、内部座標系は最も論理的な表現となっている。一部の分野では、IC表現(図に示すように、結合長、結合間の角度、結合のねじれ角)を、Z-行列またはねじれ角表現(torsion angle representation)と呼んでいる。残念ながら、デカルト空間での連続運動は、内部座標での不連続な角度分岐を必要とすることが多く、内部座標表現での力場を扱うことは比較的困難である。逆にデカルト空間での原子の単純な変位は、相互結合による禁則のために、直線の軌跡にならないことがある。したがって、計算最適化プログラムは、反復処理中に表現間を行き来することがよくある。これは、ポテンシャル自体の計算時間を支配し、長鎖分子では累積的な数値の不正確さをもたらす可能性がある。すべての変換アルゴリズムは、数学的には同じ結果を生成するものの、速度と数値精度が異なってくる[3]。現在、最速かつ最も正確なねじれの直交座標変換は、Natural Extension Reference Frame(NERF)法である[3]。
応用
今日、分子モデリングの手法は、無機系、生物系、高分子系の構造、動力学、表面特性、熱力学を研究するため日常的に使用されている。分子モデリングを用いて研究されている生物学的活動の種類には、タンパク質の折りたたみ、酵素の触媒作用、タンパク質の安定性、生体分子の機能に関連するコンフォメーション変化、およびタンパク質、DNA、膜複合体の分子認識などがある[4]。
参照項目
- ケモインフォマティクス
- 力場の実装の比較
- 核酸シミュレーションソフトの比較
- 分子力学モデリング用ソフトの比較
- 密度汎関数理論
- 分子グラフィックスシステムの一覧
- タンパク質構造予測ソフトウェアの一覧
- モンテカルロ法による分子モデリングソフトウェアの一覧
- ナノ構造モデリングのソフトウェアの一覧
- 分子設計ソフトウェア
- 分子動力学法
- 分子工学
- 分子グラフィックス
- 分子モデル
- GPUによる分子モデリング
- 分子エディタ
- モンテカルロ法
- 量子化学および固体物理計算ソフトの一覧
- 半経験的分子軌道法
- 構造バイオインフォマティクス
- タンパク質構造予測
脚注
- ^ Leach, Andrew R. (2009). Molecular modelling : principles and applications. Pearson Prentice Hall. ISBN 978-0-582-38210-7. OCLC 635267533
- ^ “Simulations of inorganic-bioorganic interfaces to discover new materials: insights, comparisons to experiment, challenges, and opportunities”. Chemical Society Reviews 45 (2): 412–48. (January 2016). doi:10.1039/C5CS00890E. PMID 26750724.
- ^ a b “Practical conversion from torsion space to Cartesian space for in silico protein synthesis”. Journal of Computational Chemistry 26 (10): 1063–8. (July 2005). doi:10.1002/jcc.20237. PMID 15898109.
- ^ “CHARMM-GUI Input Generator for NAMD, GROMACS, AMBER, OpenMM, and CHARMM/OpenMM Simulations Using the CHARMM36 Additive Force Field”. Journal of Chemical Theory and Computation 12 (1): 405–13. (January 2016). doi:10.1021/acs.jctc.5b00935. PMC 4712441. PMID 26631602.
推薦文献
- Computer simulation of liquids. Oxford University Press. (1989). ISBN 0-19-855645-4
- Understanding Molecular Simulation: From Algorithms to Applications. (1996). ISBN 0-12-267370-0
- The Art of Molecular Dynamics Simulation. (2004). ISBN 0-521-82568-7
- Molecular Simulation of Fluids: Theory, Algorithms and Object-Orientation. (2002). ISBN 0-444-51082-6
- Computational Chemistry and Molecular Modeling Principles and Applications. Springer-Verlag GmbH. (2008). ISBN 978-3-540-77302-3



