HIV-1プロテアーゼ
| HIV-1 Protease (Retropepsin) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 識別子 | |||||||||
| EC番号 | 3.4.23.16 | ||||||||
| CAS登録番号 | 144114-21-6 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| 遺伝子オントロジー | AmiGO / QuickGO | ||||||||
| |||||||||
HIV-1プロテアーゼ(英: HIV-1 protease、HIV-1 PR)は、レトロウイルス型アスパラギン酸プロテアーゼ(レトロペプシン)の1つであり、レトロウイルスにおいてペプチド結合の加水分解に関与している酵素である。HIV-1プロテアーゼは、AIDSの原因となるレトロウイルスであるHIVの生活環に必要不可欠である[1][2]。このプロテアーゼは新たに合成されたポリプロテイン(すなわちGagとGag-Pol[3])を9か所で切断し、宿主細胞外で感染性を有するウイルス形態である、ビリオンの構成要素となるタンパク質を形成する[4]。HIV-1プロテアーゼが有効に機能していない場合、HIVビリオンは感染性を持たない[5][6]。
構造
[編集]
成熟型HIV-1プロテアーゼは約22 kDaのホモ二量体として存在し、各サブユニットは99アミノ酸から構成される。同一な2つのサブユニットの間に1つの活性部位が位置し、そこにはアスパラギン酸プロテアーゼに共通した、特徴的なAsp-Thr-Gly型触媒三残基が存在している[8]。HIV-1プロテアーゼは二量体の状態でのみ機能し、成熟型プロテアーゼでは各単量体に由来する2つのAsp25が互いに協働的に触媒残基として機能する[9]。さらに、HIV-1プロテアーゼには2つのフラップ構造が存在し、この部位は酵素が基質を結合した際に最大で7 Å移動する[10]。
生合成
[編集]
前駆体
[編集]Gag-Polポリプロテインには、HIV-1プロテアーゼを含む複数の未成熟タンパク質が含まれている[9]。プロテアーゼのN末端側にはフレームシフト領域に位置するp6pol、C末端側には逆転写酵素が存在している[11]。
この前駆体タンパク質が機能的なタンパク質となるためには、プロテアーゼ単量体が他のプロテアーゼ単量体と結合し、それぞれ触媒三残基のAsp25を提供して機能的な活性部位を形成しなければならない[9]。
合成機構
[編集]HIVのRNAは、逆転写酵素、インテグラーゼ、成熟型HIV-1プロテアーゼを伴って細胞へ侵入する。逆転写酵素はウイルスRNAをDNAへ変換し、インテグラーゼはウイルスのゲノム情報を宿主のDNAへ組み込む。ウイルスDNAは核内で潜伏状態となるか、もしくは宿主の装置によってmRNAへの転写とGag-Polポリプロテインへの翻訳が行われ、その後、成熟型HIV-1プロテアーゼによって個々の機能的タンパク質(HIV-1プロテアーゼを含む)へと切断される[9]。
HIV-1プロテアーゼ前駆体は、自己プロセシングと呼ばれる機構によってGag-Polポリプロテインからの切断を促進し、自身の産生を触媒する。HIV-1プロテアーゼの自己プロセシングは次の2段階によって特徴づけられる。(1) フォールディングしたプロテアーゼ領域が二量体を形成し、N末端側のp6pol-プロテアーゼ間切断部位での切断はこの分子内で行われる。この切断によって新生プロテアーゼ-逆転写酵素中間体の酵素活性が高まる。(2) C末端側のプロテアーゼ-逆転写酵素間切断部位での切断はこの中間体分子間で行われ、成熟型プロテアーゼ二量体が形成される[12][13]。この二量体化過程によって、各単量体由来の2つのAsp25触媒残基を有する、完全に機能的な活性部位が形成される[14]。
機能
[編集]HIV-1プロテアーゼは二重の機能を有する。HIV-1プロテアーゼ前駆体は、自己プロセシングによって自身の成熟型酵素への変換の触媒を担う[15]。そして成熟型プロテアーゼはGag-Polポリプロテイン内の9か所のペプチド結合を特異的に加水分解し、成熟型の機能的タンパク質を形成する。こうして切断されるタンパク質には逆転写酵素、インテグラーゼ、RNaseHが含まれており、ウイルスの複製に必要である[4]。
機構
[編集]HIV-1プロテアーゼはアスパラギン酸プロテアーゼであり、二量体型HIV-1プロテアーゼはアスパラギン酸残基を介して加水分解を行う。HIV-1プロテアーゼの活性部位の2つのAsp25残基は、微小環境によるpKaの差異によって、一方は脱プロトン化され、もう一方はプロトン化されている[16]。
一般的なアスパラギン酸プロテアーゼの機構として、酵素の活性部位に基質が正しく結合すると、脱プロトン化されたAsp25が塩基触媒として作用し、入ってきた水分子が脱プロトン化されてより良い求核剤となる。こうして形成された水酸化物イオンはペプチド結合のカルボニル炭素を攻撃し、当初プロトン化されていたAsp25によって安定化された一過性のオキシアニオン中間体が形成される。オキシアニオンは二重結合を再形成し、ペプチド結合が切断される一方、当初脱プロトン化されていたAsp25は酸触媒としてプロトンをアミノ基に供与してアミノ基をより良い脱離基とし、再び元の脱プロトン化状態へと戻る[2][17]。

HIV-1プロテアーゼの多くの特性が非ウイルス型のアスパラギン酸プロテアーゼと共通したものである一方で、協調的な加水分解の触媒を行うこと、すなわち触媒時に求核剤となる水分子とプロトン化Asp25が同時にペプチド結合を攻撃することを示すエビデンスが得られている[17][18]。
薬剤標的として
[編集]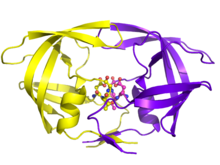
HIV-1プロテアーゼはHIVの複製において重要な役割を果たしているため、治療薬の主要な標的となっている。HIV-1プロテアーゼ阻害剤は基質の四面体型中間体を模倣して活性部位に特異的に結合し、酵素を「スタック」させることで機能する。活性型プロテアーゼを欠くウイルス粒子は、組み立てと出芽の後、感染性を有するビリオンへ成熟することができない。いくつかのプロテアーゼ阻害薬がHIVに対する治療に認可されている[19]。
現在FDAによって承認されているHIV-1プロテアーゼ阻害薬は、インジナビル、ネルフィナビル、ロピナビル、アンプレナビル、ホスアンプレナビル、アタザナビル、チプラナビル、ダルナビルである。これらの阻害薬はそれぞれ分子構成が異なり、そのため活性部位の遮断などの作用機序も異なる。また、他の阻害薬の血中濃度を高めることを目的として用いられるものや(リトナビル)、ウイルスが他の阻害薬に耐性を示す特定の状況でのみ使用されるもの(チプラナビル)もある[4][20]。
進化と耐性
[編集]レトロウイルスは変異率が高く、触媒三残基配列も含まれるmutationally sensitive regionと呼ばれる領域では特に高い。HIV-1プロテアーゼのわずかなアミノ酸の変化で阻害薬の効果はかなり弱まり、複製阻害薬による選択圧下ではこの酵素の活性部位は迅速に変化する[21][22]。
一般的に薬剤耐性の増大と関係しているのは、major mutationとsecondary mutationと呼ばれる2種類の変異である。Major mutationはプロテアーゼの活性部位の変異を伴い、選択的阻害薬の結合を防ぐ。Secondary mutationはmajor mutationが生じた後に生じる酵素の周縁部の分子的変化であり、耐性獲得後の触媒活性の増大をもたらしていると考えられている[3]。こうした薬剤耐性の獲得を最低限に抑えるために、1種類の薬剤を用いるのではなく、HIVの複製サイクルに重要ないくつかの面を阻害する薬剤の同時投与が行われる。プロテアーゼ以外に、逆転写酵素、ウイルスの接着、膜融合、cDNAの組み込み、ビリオンの組み立て過程などが薬剤の標的となっている[23][24]。
出典
[編集]- ^ “The structure and function of the aspartic proteinases”. Annual Review of Biophysics and Biophysical Chemistry 19 (1): 189–215. (1990). doi:10.1146/annurev.bb.19.060190.001201. PMID 2194475.
- ^ a b “HIV-1 protease: mechanism and drug discovery”. Organic & Biomolecular Chemistry 1 (1): 5–14. (January 2003). doi:10.1039/b208248a. PMID 12929379.
- ^ a b “The role of select subtype polymorphisms on HIV-1 protease conformational sampling and dynamics”. The Journal of Biological Chemistry 289 (24): 17203–14. (June 2014). doi:10.1074/jbc.M114.571836. PMC 4059161. PMID 24742668.
- ^ a b c “HIV protease inhibitors: a review of molecular selectivity and toxicity” (英語). HIV/AIDS: Research and Palliative Care 7: 95–104. (April 2015). doi:10.2147/hiv.s79956. PMC 4396582. PMID 25897264.
- ^ “Activity of purified biosynthetic proteinase of human immunodeficiency virus on natural substrates and synthetic peptides”. Proceedings of the National Academy of Sciences of the United States of America 86 (3): 807–11. (February 1989). Bibcode: 1989PNAS...86..807K. doi:10.1073/pnas.86.3.807. PMC 286566. PMID 2644644.
- ^ “Active human immunodeficiency virus protease is required for viral infectivity”. Proceedings of the National Academy of Sciences of the United States of America 85 (13): 4686–90. (July 1988). Bibcode: 1988PNAS...85.4686K. doi:10.1073/pnas.85.13.4686. PMC 280500. PMID 3290901.
- ^ “HIV-1 protease molecular dynamics of a wild-type and of the V82F/I84V mutant: possible contributions to drug resistance and a potential new target site for drugs”. Protein Science 13 (4): 1108–23. (April 2004). doi:10.1110/ps.03468904. PMC 2280056. PMID 15044738. オリジナルの2008-12-16時点におけるアーカイブ。.
- ^ “Folding regulates autoprocessing of HIV-1 protease precursor”. The Journal of Biological Chemistry 280 (12): 11369–78. (March 2005). doi:10.1074/jbc.M412603200. PMID 15632156.
- ^ a b c d “Initial cleavage of the human immunodeficiency virus type 1 GagPol precursor by its activated protease occurs by an intramolecular mechanism”. Journal of Virology 78 (16): 8477–85. (August 2004). doi:10.1128/JVI.78.16.8477-8485.2004. PMC 479095. PMID 15280456.
- ^ “Structure of complex of synthetic HIV-1 protease with a substrate-based inhibitor at 2.3 A resolution”. Science 246 (4934): 1149–52. (December 1989). doi:10.1126/science.2686029. PMID 2686029.
- ^ “Autoprocessing of HIV-1 protease is tightly coupled to protein folding”. Nature Structural Biology 6 (9): 868–75. (September 1999). doi:10.1038/12327. PMID 10467100.
- ^ “Kinetics and mechanism of autoprocessing of human immunodeficiency virus type 1 protease from an analog of the Gag-Pol polyprotein”. Proceedings of the National Academy of Sciences of the United States of America 91 (17): 7970–4. (August 1994). Bibcode: 1994PNAS...91.7970L. doi:10.1073/pnas.91.17.7970. PMC 44526. PMID 8058744.
- ^ “A transient precursor of the HIV-1 protease. Isolation, characterization, and kinetics of maturation”. The Journal of Biological Chemistry 271 (8): 4477–81. (February 1996). doi:10.1074/jbc.271.8.4477. PMID 8626801.
- ^ “HIV-1 protease function and structure studies with the simplicial neighborhood analysis of protein packing method”. Proteins 73 (3): 742–53. (November 2008). doi:10.1002/prot.22094. PMC 2765824. PMID 18498108.
- ^ “Understanding HIV-1 protease autoprocessing for novel therapeutic development”. Future Medicinal Chemistry 5 (11): 1215–29. (July 2013). doi:10.4155/fmc.13.89. PMC 3826259. PMID 23859204.
- ^ “Ionization states of the catalytic residues in HIV-1 protease”. Nature Structural Biology 3 (11): 946–50. (November 1996). doi:10.1038/nsb1196-946. PMID 8901873.
- ^ a b “A combined quantum/classical molecular dynamics study of the catalytic mechanism of HIV protease”. Journal of Molecular Biology 261 (3): 454–69. (August 1996). doi:10.1006/jmbi.1996.0476. PMID 8780786.
- ^ “Structure at 2.5-A resolution of chemically synthesized human immunodeficiency virus type 1 protease complexed with a hydroxyethylene-based inhibitor”. Biochemistry 30 (6): 1600–9. (February 1991). doi:10.1021/bi00220a023. PMID 1993177.
- ^ Rang and Dale's pharmacology (6th ed.). Philadelphia, Pa., U.S.A.: Churchill Livingstone/Elsevier. (2007). ISBN 9780808923541
- ^ “Influence of drug transport proteins on the pharmacokinetics and drug interactions of HIV protease inhibitors”. Journal of Pharmaceutical Sciences 100 (9): 3636–54. (September 2011). doi:10.1002/jps.22655. PMC 3750718. PMID 21698598.
- ^ “Selection of high-level resistance to human immunodeficiency virus type 1 protease inhibitors”. Antimicrobial Agents and Chemotherapy 47 (2): 759–69. (February 2003). doi:10.1128/AAC.47.2.759-769.2003. PMC 151730. PMID 12543689.
- ^ “Complete mutagenesis of the HIV-1 protease”. Nature 340 (6232): 397–400. (August 1989). Bibcode: 1989Natur.340..397L. doi:10.1038/340397a0. PMID 2666861.
- ^ “New targets for inhibitors of HIV-1 replication”. Nature Reviews. Molecular Cell Biology 1 (1): 40–9. (October 2000). doi:10.1038/35036060. PMID 11413488.
- ^ “The design of drugs for HIV and HCV”. Nature Reviews. Drug Discovery 6 (12): 1001–18. (December 2007). doi:10.1038/nrd2424. PMID 18049474.
関連項目
[編集]外部リンク
[編集]- ペプチダーゼとインヒビターに関するMEROPSオンラインデータベース: A02.001
- Proteopedia HIV-1_protease - インタラクティブ3DによるHIV-1プロテアーゼ構造の可視化
- Proteopedia Flaps_Morph_for_HIV_Protease - X線結晶構造に基づくフラップ開閉のアニメーション
- HIV-1 Protease - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス


