アンチトロンビン
アンチトロンビン(英: antithrombin、略称: AT)は血液凝固系のいくつかの酵素を不活性化するタンパク質であり、ヒトではSERPINC1遺伝子にコードされる。アンチトロンビンは肝臓で産生される糖タンパク質で、432アミノ酸からなる。3つのジスルフィド結合を含み、4か所のグリコシル化部位が存在する。α-アンチトロンビンは血漿中で優勢な形態のアンチトロンビンで、4つのグリコシル化部位のそれぞれにオリゴ糖が付加されている。より少ない形態であるβ-アンチトロンビンでは、1ヶ所のグリコシル化部位が常に修飾されていない状態である[5]。アンチトロンビンの活性は抗凝固薬であるヘパリンによって何倍にも増大する。ヘパリンはアンチトロンビンの第IIa因子(トロンビン)や第Xa因子への結合を強化する[6]。
命名
[編集]アンチトロンビンはアンチトロンビンIII(AT III)とも呼ばれる。アンチトロンビンIからIVの名称は1950年代に行われたSeegers、JohnsonとFellによる初期の研究に由来するものである[7]。
アンチトロンビンI(AT I)は、トロンビンがフィブリノゲンを活性化した後のフィブリンへの吸着を指す。アンチトロンビンII(AT II)は血漿中のコファクターを指し、ヘパリンとともにトロンビンとフィブリノゲンの相互作用に干渉する。アンチトロンビンIII(AT III)は血漿中でトロンビンを不活性化する物質を指す。アンチトロンビンIV(AT IV)は血液凝固の直後に活性化されたアンチトロンビンを指す[8]。アンチトロンビンIII(とおそらくアンチトロンビンI)のみが医学的に重要である。一般的にはアンチトロンビンIIIが「アンチトロンビン」と呼ばれ、そのためこの記事で議論されているのはアンチトロンビンIIIである。
構造
[編集]
アンチトロンビンの血漿中での半減期は約3日である[9]。ヒト血漿中の正常なアンチトロンビン濃度は約0.12 mg/mlであり、モル濃度では2.3 μMに相当する[10]。アンチトロンビンはヒト以外でも多数の生物種の血漿から単離されている[11]。タンパク質とcDNAの配列から推測すると、ウシ、ヒツジ、ウサギ、マウスのアンチトロンビンはすべて433アミノ酸からなり、ヒトのアンチトロンビンよりも1アミノ酸分だけ長い。この余剰なアミノ酸は6番のアミノ酸であると考えられている。ウシ、ヒツジ、ウサギ、マウスそしてヒトのアンチトロンビンのアミノ酸配列の同一性は84%から89%の間である[12]。6つのアミノ酸が3つのジスルフィド結合(Cys8-Cys128, Cys21-Cys95, and Cys248-Cys430)を形成している。4つのN-グリコシル化部位が存在する。ヒトのアンチトロンビンでは96番、135番、155番、192番のアスパラギン(Asn)であり、他の種にも同様の部位が存在する。ヒトのアンチトロンビンの優勢な形態であるα-アンチトロンビンでは、すべてのグリコシル化部位が共有結合的に結合したオリゴ糖側鎖によって占められており、その結果この形態のアンチトロンビンの分子量は58,200となる[5]。より少ない形態(約10%)であるβ-アンチトロンビンではAsn135のグリコシル化は行われていない[13]。
正常なヒトアンチトロンビンと同様の性質を有する組換え型アンチトロンビンもバキュロウイルス感染昆虫細胞や哺乳類培養細胞株を用いて生産されている[14][15][16][17]。一般的にこうした組換えアンチトロンビンは正常なアンチトロンビンとは異なるグリコシル化パターンを有しており、典型的にはアンチトロンビンの構造研究に利用される。蛋白質構造データバンクにはアンチトロンビンの構造が多数登録されており、この記事でもさまざまなグリコシル化パターンの構造が示されている。
機能
[編集]

アンチトロンビンはセルピン(セリンプロテアーゼインヒビター)であり、α1-アンチキモトリプシン、α2-アンチプラスミン、ヘパリンコファクターIIなど他の血漿プロテアーゼインヒビターと構造的に類似している。
アンチトロンビンの生理的標的となるプロテアーゼは、主に活性化された第II因子(IIa、トロンビン)、そして血液凝固の内因性経路の因子、すなわち第X因子(Xa)、第IX因子(IXa)、第XI因子(XIa)、第XII因子(XIIa)であり、外因性経路の第VII因子(VIIa)も標的となる[20]。また、トリプシンや補体系の古典的経路に関与するC1複合体のC1sサブユニットなど、血液凝固に関与していない他のセリンプロテアーゼも不活性化する[12][21]。
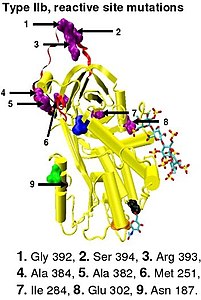
プロテアーゼの不活性化は、アンチトロンビンがプロテアーゼと1:1の複合体を形成し、通常の基質がプロテアーゼの活性部位にアクセスできなくなることによって引き起こされる[12]。アンチトロンビン-プロテアーゼ複合体の形成には、プロテアーゼとアンチトロンビン内の特定の反応性ペプチド結合との相互作用が関与している。ヒトのアンチトロンビンでは、この結合はアルギニン393番(Arg393)とセリン394番(Ser394)の間の結合である[12]。
このアンチトロンビンの反応性結合へ攻撃した結果、プロテアーゼは不活性なアンチトロンビン-プロテアーゼ複合体にトラップされた状態となると考えられている。プロテアーゼが通常の基質内の同様の結合を攻撃した際には基質タンパク質は迅速に切断されるが、アンチトロンビンの反応性結合への攻撃の開始によってアンチトロンビンは活性化され、プロテアーゼはタンパク質切断の中間段階にトラップされる。トロンビンがアンチトロンビン内の反応性結合の切断を完了することは可能であり、それによって不活性なアンチトロンビン-トロンビン複合体は解離するが、この反応が起こるためには3日以上の時間が必要となる[22]。しかしながら、P3-P4とP1'-P2'間の結合はそれぞれ好中球エラスターゼと細菌酵素サーモリシンによって迅速に切断され、その結果アンチトロンビンはトロンビンの活性を阻害することができなくなる[23]。アンチトロンビンによるプロテアーゼ阻害活性はヘパリンの結合によって大幅に強化され、好中球エラスターゼによる不活性化も同様に強化される[23]。
アンチトロンビンとヘパリン
[編集]アンチトロンビンは生理的な標的酵素であるトロンビン、第Xa因子、第IXa因子をそれぞれ速度定数(結合速度定数)7–11 x 103 M−1 s−1、2.5 x 103 M−1 s−1、1 x 10 M−1 s−1で不活性化する[5][24]。アンチトロンビンによるトロンビン不活性化の速度はヘパリンの存在下で1.5–4 x 107 M−1 s−1まで加速され、反応は2000倍から4000倍加速される[25][26][27][28]。ヘパリン存在下での第Xa因子の阻害の加速は500倍から1000倍であり、速度定数の最大値はトロンビン阻害の1/10である[25][28]。アンチトロンビンによる第IXa因子の阻害の速度はヘパリンと生理的レベルのカルシウムの存在下で約100万倍加速される[24]。
アンチトロンビンはヘパリンポリマーに含まれる特定の五糖配列(GlcNAc/NS(6S)-GlcA-GlcNS(3S,6S)-IdoA(2S)-GlcNS(6S))に結合する。この五糖配列への結合に伴い、プロテアーゼ活性の阻害は2つの異なる機構によって増大する[29]。1つの機構では、ヘパリンはアンチトロンビンの反応部位ループ(RSL)のコンフォメーション変化に依存して第IXa因子と第Xa因子の阻害を促進し、そのためアロステリックな機構である[30]。もう1つの機構では、アンチトロンビン、トロンビン、ヘパリンの三者複合体の形成に依存してトロンビンの阻害が促進される[30]。
アロステリック活性化
[編集]
第IXa因子と第Xa因子に対する阻害の増強には、ヘパリンの五糖配列が必要となる。五糖の結合に応答してアンチトロンビンに生じるコンフォメーション変化については詳細な記載がなされている[18][31][32]。
ヘパリンが結合していない場合、反応部位ループのN末端領域に位置するP14位とP15位のアミノ酸はタンパク質の本体(具体的にはβシートAの上部)に埋め込まれている。この特徴はヘパリンコファクターII、α1-アンチキモトリプシン、MENTなど他のセルピンと共通している。
第IXa因子と第Xa因子の阻害と最も関係の深いコンフォメーション変化は、このP14位とP15位のアミノ酸が関与するものである。これらのアミノ酸が位置する反応部位ループのN末端領域はヒンジ領域と呼ばれている。ヘパリンの結合に応答したヒンジ領域内のコンフォメーション変化によってP14とP15はタンパク質の本体から排除されること、またこのコンフォメーション変化を防ぐことで第IXa因子と第Xa因子の阻害の増強が起こらなくなることが示されている[30]。ヒンジ領域のコンフォメーション変化による反応部位ループの柔軟性の増加は、第IXa因子と第Xa因子の阻害の増強に影響を与える重要な因子であると考えられている。五糖が存在しない場合、P14位とP15位のアミノ酸が排除された活性型コンフォメーションをとるアンチトロンビンの割合は0.25%と計算されている[30]。
非アロステリック活性化
[編集]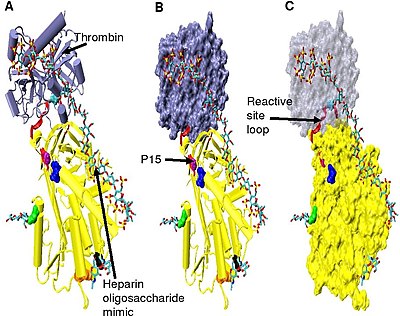
トロンビンの阻害の増強にはヘパリンの五糖に加えてさらに13単位の糖が必要である[33]。これは、アンチトロンビンとトロンビンに同一のヘパリン鎖が結合する必要があるためであると考えられている。
アンチトロンビン-トロンビン-ヘパリン三者複合体構造中では、他の非活性化型構造やヘパリン活性化型構造と比較して、反応部位ループのC末端部分(P'側)が伸長したコンフォメーションとなっている[34]。アンチトロンビンのP'領域は他のセルピンのP'領域と比較して例外的に長く、非活性化型構造やヘパリン活性化型構造では強固な水素結合によるβターンを形成している。P'領域の伸長は、βターンの形成に関与しているすべての水素結合が破壊されることで生じる[34]。
アンチトロンビン-トロンビン-ヘパリン三者複合体構造中ではヒンジ領域のP9位からP14位のアミノ酸の電子密度は観察されておらず、モデルを置くことができていない。コンフォメーションの柔軟性のためであると考えられている。このコンフォメーションの柔軟性は、P14位とP15位が埋め込まれたコンフォメーションと排除されたコンフォメーションとの間の平衡が複合体中に存在する可能性を示唆している。P15のグリシンはβシートAに埋め込まれている[34]。
活性に対するグリコシル化の影響
[編集]α-アンチトロンビンとβ-アンチトロンビンはヘパリンに対する親和性が異なる[35]。両者の解離定数は五糖に対しては3倍、ヘパリン全長に対しては10倍以上β-アンチトロンビンの方が高い親和性を持つ[36]。β-アンチトロンビンの高い親和性は、ヘパリンの初期の結合に伴って生じるタンパク質内のコンフォメーション変化が速いためであると考えられている。α-アンチトロンビンに存在するAsn135のグリコシル化はヘパリンの結合を妨げることはないが、その結果生じるコンフォメーション変化を阻害すると考えられている[35]。
β-アンチトロンビンはα-アンチトロンビンの5–10%のレベルでしか存在しないが、そのヘパリンに対する高い親和性のため、組織の損傷後の血栓形成の制御にはα-アンチトロンビンよりも重要であると考えられている。事実、大動脈損傷後のトロンビンの阻害はβ-アンチトロンビンのみに起因している[37]。
疾患における役割
[編集]アンチトロンビンが正常な血液凝固の調節に重要な役割を果たしていることは、先天性や後天性のアンチトロンビン欠乏症との相関によって明らかにされており、欠乏症の患者は血栓性疾患を発症するリスクが高くなる[38]。一般的に、アンチトロンビン欠乏症は患者が再発性静脈血栓症と肺塞栓症を発症した際に明らかとなる。
後天性アンチトロンビン欠乏症
[編集]後天性アンチトロンビン欠乏症は3つの異なる機構によって生じる。1つ目の機構は、タンパク尿性のネフローゼ症候群と関係した腎不全と共に生じる排泄の増加である。2つ目の機構は、肝不全や肝硬変または早産による未成熟な肝臓でみられる産生の減少である。3つ目の機構は消費の加速によるものであり、重大な外傷性損傷の結果であることが最も多いが、大手術や人工心肺装置などの介入の結果生じる可能性もある[39]。
先天性アンチトロンビン欠乏症
[編集]先天性アンチトロンビン欠乏症の発生率は通常の集団では2000人から5000人に1人と推計されており、先天性アンチトロンビン欠乏症の家系は1965年に最初に記載されている[40][41]。その後、機能的・免疫化学的な解析によって先天性アンチトロンビン欠乏症はタイプIとタイプIIに分類された[42]。血液凝固プロテアーゼを効果的に阻害するためには、アンチトロンビンの活性を適切なレベル(少なくとも正常な機能的レベルの70%)に維持することが必要不可欠である[43]。一般的にはタイプIまたはタイプIIのアンチトロンビン欠乏症の結果、アンチトロンビンの機能レベルは正常値の50%未満に低下している[44]。
タイプIアンチトロンビン欠乏症
[編集]タイプIアンチトロンビン欠乏症は、血中のアンチトロンビン活性とアンチトロンビン濃度の双方の低下によって特徴づけられる。ヘパリンに対する親和性に基づいて、タイプI欠乏症はさらにIa、Ibの2つのサブグループに分類される。サブグループIaの患者のアンチトロンビンはヘパリンに対する親和性は正常であるが、サブグループIbでは親和性が低下している[45]。その後の機能的解析により、サブグループIbの症例ではヘパリンに対する親和性の低下だけでなく、反応部位、ヘパリン結合部位やアンチトロンビンの血中濃度に影響を与える複数の多面的な異常が存在することが発見された。国際血栓止血学会(International Society on Thrombosis and Haemostasis)の学術標準化委員会(Scientific and Standardization Committee)による改訂分類システムでは、サブグループIbの症例はタイプII PE(pleiotropic effect)に分類されている[46]。
タイプI欠乏症の大部分の症例はアンチトロンビン遺伝子内の点変異、欠失、または小さな挿入によるものである。こうした遺伝的変異はさまざまな機構でタイプI欠乏症を引き起こす。
- 生合成の完了後に適切に血中へ放出されない、または血中に短期間しか存在しない不安定なアンチトロンビンが産生される(例: コドン106–108の6塩基対欠失など)[47]。
- アンチトロンビン遺伝子のmRNAのプロセシングが影響を受ける。
- 小さな挿入または欠失によってフレームシフトが起こり、本来の終止コドンよりも上流で翻訳が終結する。
- 点変異によってアルギニンのコドンが終止コドンに置き換えられ、上流で翻訳が終結する(例: コドン129のCGA→TGA変異)[48]。
タイプIIアンチトロンビン欠乏症
[編集]タイプIIアンチトロンビン欠乏症は、血中のアンチトロンビンのレベルは正常であるが、アンチトロンビンの活性が低下していることで特徴づけられる。当初は、アンチトロンビンのどの機能的活性が影響を受けるかによって、タイプII欠乏症を3つのサブグループ(IIa、IIb、IIc)に分類することが提唱されていた[45]。
- サブグループIIa - トロンビン、第Xa因子の不活性化の低下、ヘパリン親和性の低下。
- サブグループIIb - トロンビン不活性化の低下、ヘパリン親和性は正常。
- サブグループIIc - トロンビン、第Xa因子の不活性化は正常、ヘパリン親和性は低下。
改訂された分類システムでは、タイプII欠乏症は上述したタイプII PEに加え、変異によって反応部位が影響を受けるタイプII RS(reactive site)、ヘパリン欠乏部位が影響を受けるタイプII HBS(heparin binding site)の3つのサブグループへと分類されている[46]。Plasma Coagulation Inhibitors Subcommitteeのメンバーによって収集が行われているアンチトロンビン変異データベースでは、現在ではタイプIIaの症例はタイプII PE、タイプIIbはタイプII RS、タイプIIcはタイプII HBSに分類されている[49]。
トポニム
[編集]現在では、アンチトロンビンの遺伝的変異の特定は比較的容易である。しかしながら、現代的同定技術が利用されるようになる以前は、アンチトロンビンの変異にはトポニム、すなわち欠乏症患者の居住する町や都市の名称が変異の名称として利用されていた[50]。その後、現代的な変異同定によってアンチトロンビンの多くのトポニムが実際には同一の遺伝的変異によるものであることが示された。例えばアンチトロンビン-Toyama(富山)は-Kumamoto(熊本)、-Amien、-Tours、-Paris-1、-Paris-2、-Alger、-Padua-2、-Barcelonaと同一の変異である[49]。
医学における利用
[編集]アンチトロンビンはタンパク質製剤として利用されており、ヒト血漿からの精製や[51]、組換え生産が行われている。例えば、ATrynは遺伝子組換えヤギの乳中に産生される[52][53]。
アンチトロンビンは、先天的アンチトロンビン欠乏症患者の手術や出産前後の抗凝固薬としてFDAの承認を受けている[51][53]。
アンチトロンビンは敗血症において播種性血管内凝固症候群などを低減させる研究が行われているが、重症敗血症患者に対する有効性は確認されていない[54]。
切断型と潜在型のアンチトロンビン
[編集]
反応部位の切断はトロンビンをトラップさせるが、切断された反応部位ループは結合したプロテアーゼとともに移動し、ループはβシートAの中央部に新たに6つ目のストランドを形成する。この反応部位ループの動きは切断がなくとも誘導されることがあり、その結果構造はPAI-1の生理的な潜在型(latent)コンフォメーションと同一となる[55]。そのため、アンチトロンビンの反応部位ループが切断されていない状態でタンパク質の本体に取り込まれたコンフォメーションは、潜在型アンチトロンビン(latent antithrombin、L-antithrombin)と呼ばれる。PAI-1とは対照的に、アンチトロンビンのネイティブコンフォメーションから潜在型コンフォメーションへの変換は不可逆的である。
ネイティブ型のアンチトロンビンは加熱のみ、またはクエン酸存在下での加熱によって潜在型アンチトロンビンへと変換される[56][57]。しかし極度の加熱がなくとも、37°C(体温)で血中を循環するアンチトロンビンは24時間で10%が潜在型アンチトロンビンへと変換される[58][59]。
ネイティブ型のアンチトロンビンの立体構造は1994年に決定された[31][32]。予想外なことに、ヘテロ二量体として結晶化したタンパク質の一方の分子はネイティブ型、もう一方の分子は潜在型の構造であった。形成された潜在型アンチトロンビンは直ちにネイティブ型アンチトロンビン分子と結合してヘテロ二量体を形成し、潜在型アンチトロンビンが分析的に検出可能となるのは潜在型アンチトロンビンの濃度が全アンチトロンビンの50%を超えてからである[59]。潜在型アンチトロンビンは標的の血液凝固プロテアーゼに対して不活性であるだけでなく、活性のあるネイティブ型アンチトロンビンとの二量体化もネイティブ型分子の不活性化につながる。α-アンチトロンビンとは対照的に、ヘパリン活性化β-アンチトロンビンと潜在型アンチトロンビンとの二量体化は安定なため、潜在型アンチトロンビンの形成またはその後の二量体形成によるアンチトロンビン活性の喪失の生理的影響は大きなものとなる[59]。
ネイティブ型から潜在型への変換の中間体となる形態のアンチトロンビンも単離されており、プレ潜在型アンチトロンビン(prelatent antithrombin)と呼ばれている[60]。
抗血管新生作用
[編集]血管新生は既存の血管から新たな血管が成長する生理的過程である。正常な生理的条件下では血管新生は緊密に調節されており、促進因子と抑制因子とのバランスによって制御されている。腫瘍の成長は血管新生に依存しており、腫瘍の成長時には腫瘍細胞は産生する血管新生抑制因子の量を減少させるとともに、血管新生促進因子を持続的に産生することが必要となる[61]。切断型や潜在型のアンチトロンビンは血管新生と腫瘍成長を強力に阻害することが動物モデルで示されている[62]。
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000117601 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000026715 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ a b c Bjork, I; Olson, JE (1997). Antithrombin, A bloody important serpin (in Chemistry and Biology of Serpins). Plenum Press. pp. 17–33. ISBN 978-0-306-45698-5
- ^ Finley, Alan; Greenberg, Charles (2013-06-01). “Review article: heparin sensitivity and resistance: management during cardiopulmonary bypass”. Anesthesia and Analgesia 116 (6): 1210–1222. doi:10.1213/ANE.0b013e31827e4e62. ISSN 1526-7598. PMID 23408671.
- ^ “An antithrombin reaction to prothrombin activation”. Am. J. Physiol. 176 (1): 97–103. (1954). doi:10.1152/ajplegacy.1953.176.1.97. PMID 13124503.
- ^ “Identity of plasma-activated factor X inhibitor with antithrombin 3 and heparin cofactor”. J. Biol. Chem. 246 (11): 3712–3719. (1971). PMID 4102937.
- ^ “Metabolism of antithrombin III (heparin cofactor) in man: Effects of venous thrombosis of heparin administration”. Eur. J. Clin. Invest. 7 (1): 27–35. (1977). doi:10.1111/j.1365-2362.1977.tb01566.x. PMID 65284.
- ^ “Molar antithrombin concentration in normal human plasma”. Haemostasis 13 (6): 363–368. (1983). doi:10.1159/000214823. PMID 6667903.
- ^ Jordan RE (1983). “Antithrombin in vertebrate species: Conservation of the heparin-dependent anticoagulant mechanism”. Arch. Biochem. Biophys. 227 (2): 587–595. doi:10.1016/0003-9861(83)90488-5. PMID 6607710.
- ^ a b c d “Regulation of thrombin activity by antithrombin and heparin”. Sem. Thromb. Hemost. 20 (4): 373–409. (1994). doi:10.1055/s-2007-1001928. PMID 7899869.
- ^ “Physiological variant of antithrombin-III lacks carbohydrate side-chain at Asn 135”. FEBS Lett 219 (2): 431–436. (1987). doi:10.1016/0014-5793(87)80266-1. PMID 3609301.
- ^ “Expression of functionally active human antithrombin III”. Proceedings of the National Academy of Sciences of the United States of America 84 (11): 3886–3890. (1987). doi:10.1073/pnas.84.11.3886. PMC 304981. PMID 3473488.
- ^ “Characterization of recombinant human antithrombin III synthesized in Chinese hamster ovary cells”. J. Biol. Chem. 264 (35): 21153–21159. (1989). PMID 2592368.
- ^ “Expression of biologically active human antithrombin III by recombinant baculovirus in Spodoptera frugiperda cells”. J. Biol. Chem. 266 (6): 3995–4001. (1991). PMID 1995647.
- ^ “Elimination of glycosylation heterogeneity affecting heparin affinity of recombinant human antithrombin III by expression of a beta-like variant in baculovirus-infected insect cells”. Biochem. J. 310 (Pt 1): 323–330. (1995). doi:10.1042/bj3100323. PMC 1135891. PMID 7646463.
- ^ a b “Conformational changes in serpins: II. The mechanism of activation of antithrombin by heparin”. J. Mol. Biol. 301 (5): 1287–1305. (2000). doi:10.1006/jmbi.2000.3982. PMID 10966821.
- ^ “On the size of the active site in proteases. I. Papain”. Biochem. Biophys. Res. Commun. 27 (2): 157–162. (1967). doi:10.1016/S0006-291X(67)80055-X. PMID 6035483.
- ^ “Substitution of valine for leucine 305 in factor VIIa increases the intrinsic enzymatic activity”. J. Biol. Chem. 276 (31): 29195–29199. (2001). doi:10.1074/jbc.M102187200. PMID 11389142.
- ^ “Inhibition of the activated Cls subunit of the first component of complement by antithrombin III in the presence of heparin”. Thromb. Res. 9 (3): 217–222. (1976). doi:10.1016/0049-3848(76)90210-3. PMID 982345.
- ^ “Slow, spontaneous dissociation of the antithrombin-thrombin complex produces a proteolytically modified form of the inhibitor”. FEBS Lett 119 (2): 241–244. (1980). doi:10.1016/0014-5793(80)80262-6. PMID 7428936.
- ^ a b “Probing serpin reactive-loop conformations by proteolytic cleavage”. Biochem. J. 314 (2): 647–653. (1996). doi:10.1042/bj3140647. PMC 1217096. PMID 8670081.
- ^ a b “Heparin and calcium ions dramatically enhance antithrombin reactivity with factor IXa by generating new interaction exosites”. Biochemistry 42 (27): 8143–8152. (2003). doi:10.1021/bi034363y. PMID 12846563.
- ^ a b “The kinetics of hemostatic enzyme-antithrombin interactions in the presence of low molecular weight heparin”. J. Biol. Chem. 255 (21): 10081–10090. (1980). PMID 6448846.
- ^ Griffith MJ (1982). “Kinetics of the heparin-enhanced antithrombin III/thrombin reaction. Evidence for a template model for the mechanism of action of heparin”. J. Biol. Chem. 257 (13): 7360–7365. PMID 7085630.
- ^ “Predominant contribution of surface approximation to the mechanism of heparin acceleration of the antithrombin-thrombin reaction. Elucidation from salt concentration effects”. J. Biol. Chem. 266 (10): 6353–6354. (1991). PMID 2007588.
- ^ a b “Role of the antithrombin-binding pentasaccharide in heparin acceleration of antithrombin-proteinase reactions. Resolution of the antithrombin conformational change contribution to heparin rate enhancement”. J. Biol. Chem. 267 (18): 12528–12538. (1992). PMID 1618758.
- ^ “Crystal structure of monomeric native antithrombin reveals a novel reactive center loop conformation”. J. Biol. Chem. 281 (46): 35478–35486. (2006). doi:10.1074/jbc.M607204200. PMC 2679979. PMID 16973611.
- ^ a b c d “Allosteric activation of antithrombin critically depends upon hinge region extension”. J. Biol. Chem. 279 (45): 47288–47297. (2004). doi:10.1074/jbc.M408961200. PMID 15326167.
- ^ a b “The intact and cleaved human antithrombin III complex as a model for serpin-proteinase interactions”. Nature Structural & Molecular Biology 1 (1): 48–54. (1994). doi:10.1038/nsb0194-48. PMID 7656006.
- ^ a b “Biological implications of a 3 A structure of dimeric antithrombin”. Structure 2 (4): 257–270. (1994). doi:10.1016/S0969-2126(00)00028-9. PMID 8087553.
- ^ “Synthesis of Thrombin inhibiting Heparin mimetics without side effects”. Nature 398 (6726): 417–422. (1999). doi:10.1038/18877. PMID 10201371.
- ^ a b c “Structure of the antithrombin-thrombin-heparin ternary complex reveals the antithrombotic mechanism of heparin”. Nature Structural & Molecular Biology 11 (9): 857–862. (2004). doi:10.1038/nsmb811. PMID 15311269.
- ^ a b “Structure of beta-antithrombin and the effect of glycosylation on antithrombin's heparin affinity and activity”. J. Mol. Biol. 326 (3): 823–833. (2003). doi:10.1016/S0022-2836(02)01382-7. PMID 12581643.
- ^ “The oligosaccharide side chain on Asn-135 of alpha-antithrombin, absent in beta-antithrombin, decreases the heparin affinity of the inhibitor by affecting the heparin-induced conformational change”. Biochemistry 36 (22): 6682–6691. (1997). doi:10.1021/bi9702492. PMID 9184148.
- ^ “Thrombin inhibition by antithrombin III on the subendothelium is explained by the isoform AT beta”. Arterioscler. Thromb. Vasc. Biol. 16 (10): 1292–1297. (1996). doi:10.1161/01.ATV.16.10.1292. PMID 8857927.
- ^ “Antithrombin and its inherited deficiency states”. Semin. Hematol. 34 (3): 188–204. (1997). PMID 9241705.
- ^ “Hereditary and acquired antithrombin deficiency: epidemiology, pathogenesis and treatment options”. Drugs 67 (10): 1429–1440. (2007). doi:10.2165/00003495-200767100-00005. PMID 17600391.
- ^ “Molecular genetics of antithrombin deficiency”. Blood Rev. 10 (2): 59–74. (1996). doi:10.1016/S0268-960X(96)90034-X. PMID 8813337.
- ^ Egeberg O (1965). “Inherited antithrombin deficiency causing thrombophilia”. Thromb. Diath. Haemorrh. 13: 516–530. doi:10.1055/s-0038-1656297. PMID 14347873.
- ^ “Heterogeneity of the "classical" antithrombin III deficiency”. Thromb. Haemost. 43 (2): 133–136. (1980). doi:10.1055/s-0038-1650034. PMID 7455972.
- ^ “Pleiotropic effects of antithrombin strand 1C substitution mutations”. J. Clin. Invest. 90 (6): 2422–2433. (1992). doi:10.1172/JCI116133. PMC 443398. PMID 1469094.
- ^ “Antithrombin III: summary of first database update”. Nucleic Acids Res. 22 (17): 3556–3559. (1994). PMC 308318. PMID 7937056.
- ^ a b Sas G (1984). “Hereditary antithrombin III deficiency: biochemical aspects”. Haematologica 17 (1): 81–86. PMID 6724355.
- ^ a b “Antithrombin III mutation database: first update. For the Thrombin and its Inhibitors Subcommittee of the Scientific and Standardization Committee of the International Society on Thrombosis and Haemostasis”. Thromb. Haemost. 70 (2): 361–369. (1993). doi:10.1055/s-0038-1649581. PMID 8236149.
- ^ “A recurrent deletion in the antithrombin gene, AT106-108(-6 bp), identified by DNA heteroduplex detection”. Genomics 16 (1): 298–299. (1993). doi:10.1006/geno.1993.1184. PMID 8486379.
- ^ “A common point mutation producing type 1A antithrombin III deficiency: AT129 CGA to TGA (Arg to Stop)”. Thromb. Res. 64 (5): 621–625. (1991). doi:10.1016/S0049-3848(05)80011-8. PMID 1808766.
- ^ a b Imperial College London, Faculty of Medicine, Antithrombin Mutation Database. Retrieved on 2008-08-16.
- ^ “Molecular basis of inherited human antithrombin deficiency”. Blood 80 (9): 2159–2171. (1992). PMID 1421387.
- ^ a b “Thrombate III label”. 2012年11月15日時点のオリジナルよりアーカイブ。2013年2月23日閲覧。
- ^ Research, Center for Biologics Evaluation and (2019-04-24). “ATryn” (英語). FDA.
- ^ a b Antithrombin (Recombinant) US Package Insert ATryn for Injection February 3, 2009
- ^ Allingstrup, Mikkel; Wetterslev, Jørn; Ravn, Frederikke B.; Møller, Ann Merete; Afshari, Arash (9 February 2016). “Antithrombin III for critically ill patients: a systematic review with meta-analysis and trial sequential analysis”. Intensive Care Medicine 42 (4): 505–520. doi:10.1007/s00134-016-4225-7. PMC 2137061. PMID 26862016.
- ^ “Structural basis of latency in plasminogen activator inhibitor-1”. Nature 355 (6357): 270–273. (1992). doi:10.1038/355270a0. PMID 1731226.
- ^ “Commercial antithrombin concentrate contains inactive L-forms of antithrombin”. Thromb. Haemost. 77 (2): 323–328. (1997). doi:10.1055/s-0038-1655962. PMID 9157590.
- ^ “Preparative induction and characterization of L-antithrombin: a structural homologue of latent plasminogen activator inhibitor-1”. Biochemistry 36 (42): 13133–13142. (1997). doi:10.1021/bi970664u. PMID 9335576.
- ^ “The conformational basis of thrombosis”. Thromb. Haemost. 86 (1): 14–22. (2001). doi:10.1055/s-0037-1616196. PMID 11487000.
- ^ a b c “Formation of the antithrombin heterodimer in vivo and the onset of thrombosis”. Blood 94 (10): 3388–3396. (1999). PMID 10552948.
- ^ “A novel anti-angiogenic form of antithrombin with retained proteinase binding ability and heparin affinity”. J. Biol. Chem. 276 (15): 11996–12002. (2001). doi:10.1074/jbc.M010170200. PMID 11278631.
- ^ O'Reilly MS (2007). “Antiangiogenic antithrombin”. Semin. Thromb. Hemost. 33 (7): 660–666. doi:10.1055/s-2007-991533. PMID 18000792.
- ^ “Antiangiogenic activity of the cleaved conformation of the serpin antithrombin”. Science 285 (5435): 1926–1928. (1999). doi:10.1126/science.285.5435.1926. PMID 10489375.
関連文献
[編集]- Panzer-Heinig, Sabine (2009). Antithrombin (III) - Establishing Pediatric Reference Values, Relevance for DIC 1992 versus 2007 (Thesis). Medizinische Fakultät Charité - Universitätsmedizin Berlin.
外部リンク
[編集]- ペプチダーゼとその阻害因子に関するMEROPSオンラインデータベース: I04.018
- Antithrombin III - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- Human SERPINC1 genome location and SERPINC1 gene details page in the UCSC Genome Browser.