利用者:YasuakiH/note
英語版 Protein structure の2021-02-22T04:47:18UTC)版を翻訳のため転記
タンパク質構造-Protein structure.txt
英語版の記事の修正
・ifferent protein→ ピリオド追加
タンパク質構造(protein structure)は、アミノ酸鎖分子内での原子の3次元的な配置のことである。タンパク質は、モノマー(単量体)であるアミノ酸の配列から形成されたポリマー(重合体)で、具体的にはポリペプチドである。単一のアミノ酸モノマーを、ポリマーの繰り返し単位を示した「残基」と呼ぶこともある。タンパク質は、アミノ酸が縮合反応を起こすことによって形成される。この反応は、アミノ酸がペプチド結合で互いに結合して、その反応ごとに1つの水分子が失われる。30アミノ酸以下の鎖は、慣習的に(タンパク質ではなく)ペプチドと呼ぶことが多い[1]。タンパク質は、その生物学的機能を発揮するために、水素結合、イオン結合、ファンデルワールス力、および疎水性パッキングなどのいくつかの非共有結合によって、1つまたは複数の特定の空間的コンフォメーション(立体配座)に折りたたまれる。タンパク質の機能を分子レベルで理解するためには、その立体構造を決定する必要がよくある。これは、構造生物学という科学分野のトピックであり、タンパク質の構造は、X線結晶構造解析、NMR分光法、低温電子顕微鏡(クライオ電子顕微鏡)、二重偏光干渉法などの技術を用いて決定される。
タンパク質の構造は、数十個から数千個のアミノ酸で構成されている[2]。物理的な大きさによって、タンパク質は1~100 nmのナノ粒子に分類される。非常に大きなタンパク質複合体がタンパク質サブユニットから形成されることがある。たとえば、何千個ものアクチン分子が集まってマイクロフィラメントを形成する。
同じタンパク質の異なる構造を異なるコンフォメーション(立体配座)と呼び、それらの間の遷移をコンフォメーション変化と呼ぶ。タンパク質では通常、その生物学的機能を果たす際に可逆的なコンフォメーション変化が起こる。
タンパク質の構造のレベル

タンパク質の構造には4つのレベルがある。
一次構造
タンパク質の一次構造は、ポリペプチド鎖の中のアミノ酸の配列を指す。この一次構造は、タンパク質生合成の過程で作られるペプチド結合によって保持されている。ポリペプチド鎖の両端を、それぞれの末端にある遊離基の性質に基づいて、カルボキシル末端(C末端)とアミノ末端(N末端)と呼ぶ。残基の数え方は、常にアミノ基がペプチド結合に関与していないN-末端(NH2基)から始まる。タンパク質の一次構造は、そのタンパク質に対応する遺伝子によって決定される。DNAの特定のヌクレオチドがmRNAに転写されて、それをリボソームが読み取ることで翻訳と呼ばれるプロセスが行われる。インスリンのアミノ酸配列は、フレデリック・サンガーによって発見され、タンパク質が決まったアミノ酸配列を持っていることが証明された[3][4]。タンパク質の配列は、そのタンパク質に固有であり、タンパク質の構造と機能を定義する。タンパク質の配列は、エドマン分解やタンデム質量分析法などの方法で知ることができる。ただし、多くの場合、遺伝情報を用いて遺伝子の配列から直接読み取られる。タンパク質について議論するときは「アミノ酸残基」という言葉を使うことが強く推奨されており、その理由は、ペプチド結合が形成される時に水分子が失われ、タンパク質がアミノ酸残基で構成されることによる。リン酸化やグリコシル化などの翻訳後修飾も通常は一次構造の一部とみなされるが、遺伝子から読み取ることはできない。たとえば、インスリンは51個のアミノ酸を含む2本の鎖の構成されている。一方の鎖には31個のアミノ酸があり、もう一方の鎖には20個のアミノ酸がある。
二次構造

二次構造とは、実際のポリペプチド骨格鎖上の極めて規則的な局所的部分構造を指す。二次構造には大きく分けて、α-ヘリックスとβ-ストランドまたはβ-シートの2種類があり、1951年にライナス・ポーリングらによって提案された[5]。これらの二次構造は、主鎖ペプチド基間の水素結合のパターンによって定義される。これらは規則的な形状をしており、ラマチャンドランプロット上の二面角ψとφの特定の値に制約されている。α-ヘリックスとβ-シートは、どちらも、ペプチド骨格内のすべての水素結合ドナーとアクセプターを飽和させる方法である。タンパク質の一部は順序正しく並んでいるが、規則的な構造は形成されていない。それらを、一定の立体構造を持たない展開したポリペプチド鎖であるランダムコイルと混同すべきでない。いくつかの連続した二次構造が「超二次ユニット」を形成することがある[6]。
三次構造
三次構造とは、1つのタンパク質分子(1本のポリペプチド鎖)が作る三次元構造を指す。これは、1つまたは複数のドメインを含むことがある。α-ヘリックスとβ-プリーツシートは折りたたまれてコンパクトな球状構造になる。折りたたみは、非特異的な疎水性相互作用や、疎水性残基が水と反発して潜り込むことによって推進されるが、タンパク質ドメインの各部分が、塩橋、水素結合、側鎖やジスルフィド結合の緊密なパッキングなどの特異的な三次的相互作用によって所定の位置に固定されている場合のみ、その構造は安定化する。細胞質タンパク質では、ジスルフィド結合は極めて少なく、これは細胞質基質(細胞内液)は一般に還元的な環境であることによる。
四次構造
四次構造は、1つの機能単位(多量体)として機能する2つ以上の個別のポリペプチド鎖(サブユニット)が集合した三次元構造のことである。こうしてできた多量体は、三次構造と同じ非共有結合やジスルフィド結合によって安定化する。四次構造の組織化には多くの可能性がある[7]。2つ以上のポリペプチドの複合体(すなわち複数のサブユニット)はマルチマーと呼ばれる。具体的には、サブユニットが2個の場合はダイマー(二量体)、3個の場合はトリマー(三量体)、4個の場合はテトラマー(四量体)、5個の場合はペンタマー(五量体)と呼ばれる。サブユニットは、二量体における2回軸のように、対称操作によって相互に関連付けられることがよくある。同一のサブユニットで構成される多量体には「ホモ」(homo-)という接頭語をつけ、異なるサブユニットからなる多量体には「ヘテロ」(hetero-)という接頭語をつけて呼ぶ。たとえば、ヘモグロビンの2本のα鎖と2本のβ鎖のようなヘテロテトラマー(heterotetramer)が挙げられる。
タンパク質構造のドメイン、モチーフ、フォールド

タンパク質はいくつかの構造単位から構成されているとよく言われている。これらの単位には、ドメイン、モチーフ、フォールドがある。真核生物系では約10万種類のタンパク質が発現しているとう事実にもかかわらず、ドメイン、構造モチーフ、折りたたみ(フォールド)の種類ははるかに少ない。
構造ドメイン
構造ドメインは、自己安定化するタンパク質の全体的な構造の一要素であり、多くの場合、タンパク質鎖の他の部分とは独立して折りたたまれる。多くのドメインは、さまざまなタンパク質で見られ、1つの遺伝子や1つの遺伝子ファミリーのタンパク質産物に固有のものではない。ドメインは、例えば「カルモジュリンのカルシウム結合ドメイン」のように、そのタンパク質の生物学的機能に大きく関与しているために命名され、選択されることが多い。それらのドメインは独立して安定しているため、遺伝子操作で、あるタンパク質と別のタンパク質の間でドメインを「交換」することによってキメラタンパク質を作ることができる。タンパク質チロシンホスファターゼドメインとC2ドメインのペアのように、異なるタンパク質に存在する複数のドメインを保存的に組み合わせたものは、単一のユニットとして進化する可能性のある「スーパードメイン」と呼ばれる[8]。
構造モチーフと配列モチーフ
構造モチーフと配列モチーフは、多数の異なるタンパク質において見られる、タンパク質の三次元構造やアミノ酸配列の短い断片を指す。
超二次構造
超二次構造とは、β-α-βユニットやヘリックス-ターン-ヘリックスモチーフなどの二次構造要素の特定の組み合わせを指す。
タンパク質のフォールド
タンパク質のフォールドとはタンパク質の一般的な構造のことで、ヘリックスバンドル、β-バレル、ロスマンフォールドなど、タンパク質立体構造分類データベース(SCOP)で提供されているさまざまな「折りたたみ」である[9]。関連する概念はタンパク質トポロジーである。
タンパク質ダイナミクスと立体配座アンサンブル
タンパク質は静止した物体ではなく、立体構造状態のアンサンブルを構成している。これらの状態間の遷移は通常、ナノスケールで発生し、アロステリックシグナル伝達[10]や酵素触媒反応[11]などの機能的に重要な現象と関連している。タンパク質ダイナミクスとコンフォメーション変化により、タンパク質は細胞内でナノスケールの生物学的機械として機能し、多くの場合、多タンパク質複合体の形で機能する[12]。たとえば、筋肉の収縮を司るミオシン、細胞内の荷物を微小管に沿って核から遠ざけるキネシン、細胞内の荷物を核に向かって移動させ、運動性の繊毛や鞭毛の軸線方向の拍動を生み出すダイニンなどのモータータンパク質がある。『事実上、運動性繊毛は、おそらく600以上のタンパク質からなる分子複合体で構成されたナノマシンであり、その多くが独立してナノマシンとして機能している。… 柔軟なリンカーは、それによって連結された可動性のあるタンパク質ドメインが結合パートナーを動員し、タンパク質ドメインダイナミクスを介して長距離のアロステリー誘発を可能にする。』[13]
タンパク質は、他のタンパク質との相互作用や酵素活性の影響を受けてコンフォメーション変化する比較的安定した三次構造と考えられている。しかし、タンパク質の安定性の程度はさまざまであり、安定性の低い変異体の中には天然変性タンパク質もある。これらのタンパク質は、安定した三次構造を持たない比較的「無秩序」な状態で存在し、機能している。そのため、それらを単一の固定された三次構造で説明することは困難である。立体配座アンサンブルは、天然変性タンパク質のコンフォメーション状態を、より正確かつ「ダイナミック」に表現する方法として考案された[15][14]。
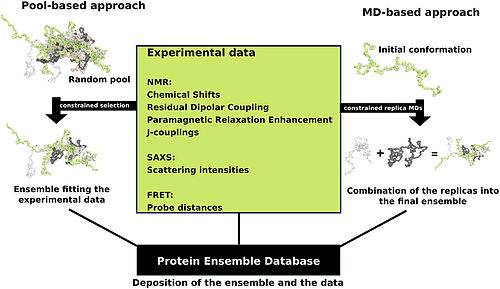
タンパク質アンサンブルファイルは、柔軟な構造を持つと考えられるタンパク質の表現である。これらのファイルを作成するには、理論的に可能なさまざまなタンパク質コンフォメーションのどれが実際に存在するかを判断する必要がある。1つのアプローチは、タンパク質データに計算アルゴリズムを適用して、アンサンブルファイルについて最も可能性の高いコンフォメーションの集合を決定するものである。タンパク質アンサンブルデータベースのデータを作成するには複数の方法があり、一般的にはプールアプローチと分子動力学(MD)アプローチの2つに分類される(図を参照)。 プールベースアプローチでは、タンパク質のアミノ酸配列を利用して、ランダムなコンフォメーションの膨大なプールを作成する。次に、このプールをさらに計算処理して、構造に基づいた各コンフォーメーションの理論的パラメータのセットを作成する。このプールから、平均的な理論パラメータがこのタンパク質の既知の実験データと厳密に一致するコンフォーメーション・サブセットが選択される。もう1つの分子動力学アプローチでは、複数の短いレプリカ(ランダムなコンフォーメーション)のMDシミュレーションを平行して行い、それらのすべてを実験データと比較する。ここでは、実験データは、コンフォメーションにかけられる制約(たとえば、原子間の既知の距離)として機能する。実験データによって設定された制限内に収まっているコンフォメーションのみが受け入れられる。このアプローチでは、大量の実験データをコンフォメーションに適用することが多いが、これは非常に計算量が多い処理である[14]。
立体配座アンサンブルは、Sic1/Cdc4[16]、p15 PAF[17]、MKK7[18]、β-シヌクレイン[19]、P27[20]など、非常にダイナミックで部分的に展開したタンパク質に対して生成されている。
タンパク質のフォールド
この節の加筆が望まれています。 |
翻訳されたポリペプチドは、主にランダムコイルとしてリボソームから出て、その天然状態に折りたたまれる(フォールディングと呼ぶ)[21][22]。タンパク質鎖の最終的な構造は、一般的にアミノ酸配列によって決定されると考えられている(アンフィンセンのドグマ)[23]。
タンパク質の安定性
タンパク質の熱力学的安定性は、折りたたまれた状態と折りたたまれていない状態の自由エネルギーの差を表す。この自由エネルギーの差は温度に非常に敏感であるため、温度が変化すると折りたたみの展開や変性が起こる可能性がある。タンパク質が変性すると、機能が失われたり、天然状態が失われる可能性がある。可溶性球状タンパク質の安定化の自由エネルギーは、通常、50 kJ/molを超えない[要出典]。二次構造の安定化のために行われる多数の水素結合と、疎水性相互作用による内部コアの安定化を考慮すると、安定化の自由エネルギーは、大きな数の間の小さな差として現れる[24]。
タンパク質の構造決定


蛋白質構造データバンク(PDB)に登録されているタンパク質の構造の約90%は、X線結晶構造解析によって決定されている[25]。この方法により、結晶化した状態のタンパク質中の電子の三次元(3-D)密度分布を測定し、それによって決定されるすべての原子の三次元座標を一定の分解能で推測することができる。既知のタンパク質構造の約9%は、核磁気共鳴分光法(NMR)法によって得られた[要出典]。より大きなタンパク質複合体の場合、低温電子顕微鏡でタンパク質の構造を決定することができる。その解像度は通常、X線結晶解析やNMRに比べて低いものであるが、最大解像度は着実に向上している。この技術は、ウイルスのコートタンパク質やアミロイド繊維のような非常に大きなタンパク質複合体において特に価値がある。
一般的な二次構造の組成は、円偏光二色性によって決定することができる。振動分光法は、ペプチド、ポリペプチド、およびタンパク質のコンフォメーションを特徴づけるためにも使用できる[26]。二次元赤外分光法は、他の方法では調べられない柔軟なペプチドやタンパク質の構造を調査する貴重な方法となっている[27][28]。タンパク質構造のより定性的な画像は、タンパク質分解によって得られることが多く、これは結晶化可能なタンパク質サンプルをスクリーニングするのにも有効である。高速平行タンパク質分解(FASTpp)をはじめとするこのアプローチの新しい実装では、精製を必要とせずに構造化画分とその安定性を探ることができる[29]。タンパク質の構造が実験的に決定されると、その構造の分子動力学法シミュレーションにより、さらに詳細な研究を計算機上で行うことができる[30]。
タンパク質構造データベース
タンパク質構造データベースとは、実験的に決定されたさまざまなタンパク質構造をモデル化したデータベースである。ほとんどのタンパク質構造データベースの目的は、タンパク質の構造を整理して注釈を付け、生物学コミュニティに実験データへの便利なアクセス方法を提供することである。タンパク質構造データベースに含まれるデータには、三次元座標のほか、X線結晶構造解析で決定された単位格子の寸法や角度などの実験情報が含まれることが多い。しかし、ほとんどの場合、タンパク質またはタンパク質の特定の構造決定には配列情報も含まれており、一部のデータベースでは、配列に基づいたクエリ(問い合わせ)を行う手段も提供されている。一方、配列データベースは配列情報が中心で、大部分のエントリには構造情報が含まれていない。タンパク質構造データベースは、構造に基づく医薬品設計などの計算生物学の多くの取り組みにおいて、計算手法の開発と、タンパク質の機能に関する洞察を得るために一部の手法で使用される大規模な実験データセットの提供の両面で重要な役割を果たしている[31]。
タンパク質の構造分類
タンパク質の構造は、その構造的な類似性、トポロジークラス、または共通の進化的起源に基づいてグループ化することができる。タンパク質立体構造分類データベース[32]およびCATHデータベース[33]は、タンパク質の2種類の異なる構造分類を提供している。構造の類似性が大きい場合、2つのタンパク質は共通の祖先から分岐した可能性があり[34]、タンパク質間で共有される構造は相同性の証拠と考えられる。次に、構造の類似性を利用して、タンパク質をスーパーファミリーに分類することができる[35]。共有されている構造が大きくても、共有されている割合が小さい場合は、共有されているフラグメントが遺伝子の水平伝播などのより劇的な進化イベントの結果である可能性があり、これらのフラグメントを共有するタンパク質をタンパク質スーパーファミリーに紐付けることはもはや正しいと見なされない[34]。タンパク質のトポロジーは、タンパク質の分類にも使われる。結び目理論と回路トポロジーは、それぞれ鎖交差と鎖内接触に基づいてタンパク質フォールドを分類するために開発された2つのトポロジーフレームワークである。
計算によるタンパク質の構造予測
タンパク質配列の決定は、タンパク質構造の決定よりもはるかに簡単である。しかし、タンパク質の構造は、その配列よりもはるかに多くの機能を知ることができる。そのため、その配列からタンパク質構造を計算で予測するための方法が数多く開発されてきた[36]。Ab initio予測法(第一原理予測法)は、タンパク質の配列のみを用いて立体構造を推定する。また、スレッディング法やホモロジーモデリング法は、タンパク質ファミリーと呼ばれる進化的に関連するタンパク質の実験的構造から、構造が未知であるタンパク質の三次元モデルを構築することができる。
関連項目
脚注
- ^ H. Stephen Stoker (1 January 2015). Organic and Biological Chemistry. Cengage Learning. p. 371. ISBN 978-1-305-68645-8
- ^ “Protein length in eukaryotic and prokaryotic proteomes”. Nucleic Acids Research 33 (10): 3390–3400. (2005-06-10). doi:10.1093/nar/gki615. PMC 1150220. PMID 15951512.
- ^ Sanger, F.; Tuppy, H. (1951-09-01). “The amino-acid sequence in the phenylalanyl chain of insulin. I. The identification of lower peptides from partial hydrolysates”. The Biochemical Journal 49 (4): 463–481. doi:10.1042/bj0490463. ISSN 0264-6021. PMC 1197535. PMID 14886310.
- ^ Sanger, F. (1959-05-15). “Chemistry of Insulin” (英語). Science 129 (3359): 1340–1344. Bibcode: 1959Sci...129.1340G. doi:10.1126/science.129.3359.1340. ISSN 0036-8075. PMID 13658959.
- ^ “The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain”. Proc Natl Acad Sci USA 37 (4): 205–211. (1951). Bibcode: 1951PNAS...37..205P. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ “New classification of supersecondary structures of sandwich-like proteins uncovers strict patterns of strand assemblage.”. Proteins 68 (4): 915–921. (2007). doi:10.1002/prot.21473. PMID 17557333.
- ^ “A periodic table of coiled-coil protein structures”. J. Mol. Biol. 385 (3): 726–32. (January 2009). doi:10.1016/j.jmb.2008.11.028. ISSN 0022-2836. PMID 19059267.
- ^ “Superdomain in the protein structure hierarchy: the case of PTP-C2.”. Protein Science 24 (5): 874–82. (2015). doi:10.1002/pro.2664. PMC 4420535. PMID 25694109.
- ^ “Estimating the total number of protein folds.”. Proteins 35 (4): 408–414. (17 September 1999). doi:10.1002/(SICI)1097-0134(19990601)35:4<408::AID-PROT4>3.0.CO;2-A. hdl:2027.42/34969. PMID 10382668. オリジナルの5 January 2013時点におけるアーカイブ。.
- ^ “Proteins MOVE! Protein dynamics and long-range allostery in cell signaling”. Protein Structure and Diseases. Advances in Protein Chemistry and Structural Biology. 83. (2011). pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668
- ^ “Hidden alternative structures of proline isomerase essential for catalysis”. Nature 462 (7273): 669–673. (Dec 2009). Bibcode: 2009Natur.462..669F. doi:10.1038/nature08615. PMC 2805857. PMID 19956261.
- ^ Donald, Voet (2011). Biochemistry. Voet, Judith G. (4th ed.). Hoboken, NJ: John Wiley & Sons. ISBN 9780470570951. OCLC 690489261
- ^ Satir, Peter; Søren T. Christensen (2008-03-26). “Structure and function of mammalian cilia”. Histochemistry and Cell Biology 129 (6): 687–93. doi:10.1007/s00418-008-0416-9. PMC 2386530. PMID 18365235. 1432-119X.
- ^ a b c Varadi, Mihaly; Vranken, Wim; Guharoy, Mainak; Tompa, Peter (2015-01-01). “Computational approaches for inferring the functions of intrinsically disordered proteins”. Frontiers in Molecular Biosciences 2: 45. doi:10.3389/fmolb.2015.00045. PMC 4525029. PMID 26301226.
- ^ Protein Ensemble Database
- ^ Mittag, Tanja; Marsh, Joseph; Grishaev, Alexander; Orlicky, Stephen; Lin, Hong; Sicheri, Frank; Tyers, Mike; Forman-Kay, Julie D. (2010-03-14). “Structure/function implications in a dynamic complex of the intrinsically disordered Sic1 with the Cdc4 subunit of an SCF ubiquitin ligase”. Structure 18 (4): 494–506. doi:10.1016/j.str.2010.01.020. ISSN 1878-4186. PMC 2924144. PMID 20399186.
- ^ De Biasio, Alfredo; Ibáñez de Opakua, Alain; Cordeiro, Tiago N.; Villate, Maider; Merino, Nekane; Sibille, Nathalie; Lelli, Moreno; Diercks, Tammo et al. (2014-02-18). “p15PAF is an intrinsically disordered protein with nonrandom structural preferences at sites of interaction with other proteins”. Biophysical Journal 106 (4): 865–874. Bibcode: 2014BpJ...106..865D. doi:10.1016/j.bpj.2013.12.046. ISSN 1542-0086. PMC 3944474. PMID 24559989.
- ^ Kragelj, Jaka; Palencia, Andrés; Nanao, Max H.; Maurin, Damien; Bouvignies, Guillaume; Blackledge, Martin; Jensen, Malene Ringkjøbing (2015-03-17). “Structure and dynamics of the MKK7-JNK signaling complex”. Proceedings of the National Academy of Sciences of the United States of America 112 (11): 3409–3414. Bibcode: 2015PNAS..112.3409K. doi:10.1073/pnas.1419528112. ISSN 1091-6490. PMC 4371970. PMID 25737554.
- ^ Allison, Jane R.; Rivers, Robert C.; Christodoulou, John C.; Vendruscolo, Michele; Dobson, Christopher M. (2014-11-25). “A relationship between the transient structure in the monomeric state and the aggregation propensities of α-synuclein and β-synuclein”. Biochemistry 53 (46): 7170–7183. doi:10.1021/bi5009326. ISSN 1520-4995. PMC 4245978. PMID 25389903.
- ^ Sivakolundu, Sivashankar G.; Bashford, Donald; Kriwacki, Richard W. (2005-11-11). “Disordered p27Kip1 exhibits intrinsic structure resembling the Cdk2/cyclin A-bound conformation”. Journal of Molecular Biology 353 (5): 1118–1128. doi:10.1016/j.jmb.2005.08.074. ISSN 0022-2836. PMID 16214166.
- ^ Zhang, Gong; Ignatova, Zoya (2011-02-01). “Folding at the birth of the nascent chain: coordinating translation with co-translational folding” (英語). Current Opinion in Structural Biology 21 (1): 25–31. doi:10.1016/j.sbi.2010.10.008. ISSN 0959-440X. PMID 21111607.
- ^ Alberts, Bruce; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walters (2002). “The Shape and Structure of Proteins”. Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 978-0-8153-3218-3
- ^ Anfinsen, C. (1972). “The formation and stabilization of protein structure”. Biochem. J. 128 (4): 737–49. doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ^ Jaenicke, R.; Heber, U.; Franks, F.; Chapman, D.; Griffin, Mary C. A.; Hvidt, A.; Cowan, D. A. (1990). “Protein Structure and Function at Low Temperatures [and Discussion]”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 326 (1237): 535–553. doi:10.1098/rstb.1990.0030. JSTOR 2398703. PMID 1969647.
- ^ Kendrew, J.C.; Bodo, G.; Dintzis, H. M.; Parrish, R. G.; Wyckoff, H.; Phillips, D.C. (1958). “A Three-Dimensional Model of the Myoglobin Molecule Obtained by X-Ray Analysis”. Nature 181 (4610): 662–666. Bibcode: 1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261.
- ^ “Vibrational spectroscopy and conformation of peptides, polypeptides, and proteins”. Advances in Protein Chemistry Volume 38. Advances in Protein Chemistry. 38. (1986). 181–364. doi:10.1016/S0065-3233(08)60528-8. ISBN 9780120342389. PMID 3541539
- ^ Lessing, J.; Roy, S.; Reppert, M.; Baer, M.; Marx, D.; Jansen, T.L.C.; Knoester, J.; Tokmakoff, A. (2012). “Identifying Residual Structure in Intrinsically Disordered Systems: A 2D IR Spectroscopic Study of the GVGXPGVG Peptide”. J. Am. Chem. Soc. 134 (11): 5032–5035. doi:10.1021/ja2114135. PMID 22356513.
- ^ Jansen, T.L.C.; Knoester, J. (2008). “Two-dimensional infrared population transfer spectroscopy for enhancing structural markers of proteins”. Biophys. J. 94 (5): 1818–1825. Bibcode: 2008BpJ....94.1818J. doi:10.1529/biophysj.107.118851. PMC 2242754. PMID 17981904.
- ^ “Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp”. PLOS ONE 7 (10): e46147. (2012). Bibcode: 2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ “Molecular Dynamics Simulations, Challenges and Opportunities: A Biologist's Prospective”. Curr. Protein Pept. Sci. 18 (11): 1163–1179. (August 2017). doi:10.2174/1389203718666170622074741. PMID 28637405.
- ^ Laskowski, RA (2011). “Protein structure databases”. Mol Biotechnol 48 (2): 183–98. doi:10.1007/s12033-010-9372-4. PMID 21225378.
- ^ Murzin, A. G.; Brenner, S.; Hubbard, T.; Chothia, C. (1995). “SCOP: A structural classification of proteins database for the investigation of sequences and structures”. Journal of Molecular Biology 247 (4): 536–540. doi:10.1016/S0022-2836(05)80134-2. PMID 7723011. オリジナルの26 April 2012時点におけるアーカイブ。.
- ^ Orengo, C. A.; Michie, A. D.; Jones, S.; Jones, D. T.; Swindells, M. B.; Thornton, J. M. (1997). “CATH--a hierarchic classification of protein domain structures”. Structure 5 (8): 1093–1108. doi:10.1016/S0969-2126(97)00260-8. PMID 9309224.
- ^ a b Pascual-García, A.; Abia, D.; Ortiz, A.R.; Bastolla, U. (2009). “Cross-over between discrete and continuous protein structure space: insights into automatic classification and networks of protein structures.”. PLOS Computational Biology 5 (3): e1000331. Bibcode: 2009PLSCB...5E0331P. doi:10.1371/journal.pcbi.1000331. PMC 2654728. PMID 19325884.
- ^ Holm, L; Rosenström, P (July 2010). “Dali server: conservation mapping in 3D.”. Nucleic Acids Research 38 (Web Server issue): W545–9. doi:10.1093/nar/gkq366. PMC 2896194. PMID 20457744.
- ^ Zhang Y (2008). “Progress and challenges in protein structure prediction”. Curr Opin Struct Biol 18 (3): 342–348. doi:10.1016/j.sbi.2008.02.004. PMC 2680823. PMID 18436442.
推薦文献
- 50 Years of Protein Structure Determination Timeline - HTML Version - National Institute of General Medical Sciences at NIH
外部リンク
 ウィキメディア・コモンズには、YasuakiH/noteに関するカテゴリがあります。
ウィキメディア・コモンズには、YasuakiH/noteに関するカテゴリがあります。
Template:Prone to spam Template:Z148
Template:Protein domains Template:Protein structure determination Template:Biomolecular structure
