「オペロン」の版間の差分
編集の要約なし |
|||
| 10行目: | 10行目: | ||
[[遺伝学]]では、 '''オペロン'''は単一の[[プロモーター]]制御下にある[[遺伝子|遺伝子の]]クラスターを含む[[デオキシリボ核酸|DNAの]]機能単位<ref>{{Cite book|first=David E.|last=Sadava|first2=David M.|last2=Hillis|first3=H. Craig|last3=Heller|first4=May|last4=Berenbaum|title=Life: The Science of Biology|publisher=Macmillan|year=2009|edition=9th|isbn=978-1-4292-1962-4|page=349|url=https://books.google.com/books?id=ANT8VB14oBUC&pg=PA349}}</ref>。 遺伝子は一緒に[[転写 (生物学)|転写]]されて[[伝令RNA|mRNA]]鎖になり、細胞質で一緒に[[翻訳 (生物学)|翻訳]]されるか、またはスプライシングを受けて別々に翻訳されるモノシストロン性 mRNA、すなわちそれぞれが単一の遺伝子産物をコードするmRNAのいくつかの鎖を作成。この結果、オペロンに含まれる遺伝子は一緒に発現されるか、まったく発現されない。オペロンを定義するには、いくつかの遺伝子を''同時転写''する必要がある<ref>{{Cite book|last=Lodish|first=Harvey|last2=Zipursky|first2=Lawrence|last3=Matsudaira|first3=Paul|last4=Baltimore|first4=David|last5=Darnel|first5=James|title=Molecular Cell Biology|publisher=W. H. Freeman|year=2000|chapter=Chapter 9: Molecular Definition of a Gene|isbn=978-0-7167-3136-8|url=https://archive.org/details/molecularcellbio00lodi}}</ref>。 |
[[遺伝学]]では、 '''オペロン'''は単一の[[プロモーター]]制御下にある[[遺伝子|遺伝子の]]クラスターを含む[[デオキシリボ核酸|DNAの]]機能単位<ref>{{Cite book|first=David E.|last=Sadava|first2=David M.|last2=Hillis|first3=H. Craig|last3=Heller|first4=May|last4=Berenbaum|title=Life: The Science of Biology|publisher=Macmillan|year=2009|edition=9th|isbn=978-1-4292-1962-4|page=349|url=https://books.google.com/books?id=ANT8VB14oBUC&pg=PA349}}</ref>。 遺伝子は一緒に[[転写 (生物学)|転写]]されて[[伝令RNA|mRNA]]鎖になり、細胞質で一緒に[[翻訳 (生物学)|翻訳]]されるか、またはスプライシングを受けて別々に翻訳されるモノシストロン性 mRNA、すなわちそれぞれが単一の遺伝子産物をコードするmRNAのいくつかの鎖を作成。この結果、オペロンに含まれる遺伝子は一緒に発現されるか、まったく発現されない。オペロンを定義するには、いくつかの遺伝子を''同時転写''する必要がある<ref>{{Cite book|last=Lodish|first=Harvey|last2=Zipursky|first2=Lawrence|last3=Matsudaira|first3=Paul|last4=Baltimore|first4=David|last5=Darnel|first5=James|title=Molecular Cell Biology|publisher=W. H. Freeman|year=2000|chapter=Chapter 9: Molecular Definition of a Gene|isbn=978-0-7167-3136-8|url=https://archive.org/details/molecularcellbio00lodi}}</ref>。 |
||
もともと、オペロンは[[原核生物]] ( [[細菌|バクテリアに]]由来する[[色素体]]のような[[細胞小器官]]を含む)にのみ存在すると考えられていたが、1990年代初期に[[真核生物]]で最初のオペロンが発見されて以来<ref>{{Cite journal|date=May 1993|title=Operons in C. elegans: polycistronic mRNA precursors are processed by trans-splicing of SL2 to downstream coding regions|journal=Cell|volume=73|issue=3|pages=521–32| |
もともと、オペロンは[[原核生物]] ( [[細菌|バクテリアに]]由来する[[色素体]]のような[[細胞小器官]]を含む)にのみ存在すると考えられていたが、1990年代初期に[[真核生物]]で最初のオペロンが発見されて以来<ref>{{Cite journal|date=May 1993|title=Operons in C. elegans: polycistronic mRNA precursors are processed by trans-splicing of SL2 to downstream coding regions|journal=Cell|volume=73|issue=3|pages=521–32|doi=10.1016/0092-8674(93)90139-H|pmid=8098272}}</ref> <ref>{{Cite journal|date=April 1997|title=The Adh-related gene of Drosophila melanogaster is expressed as a functional dicistronic messenger RNA: multigenic transcription in higher organisms|journal=The EMBO Journal|volume=16|issue=8|pages=2023–31|doi=10.1093/emboj/16.8.2023|pmid=9155028|pmc=1169805}}</ref>世代への原核生物オペロンリードの一般的には、表現ポリシストロン性 mRNAを、真核生物のオペロンは、単シストロン性のmRNAにつながるながらも以前に想定されていたよりも一般的<ref name="Operons in eukaryotes">{{Cite journal|date=November 2004|title=Operons in eukaryotes|journal=Briefings in Functional Genomics & Proteomics|volume=3|issue=3|pages=199–211|doi=10.1093/bfgp/3.3.199|pmid=15642184}}</ref>。 |
||
オペロンは[[ファージ|バクテリオファージ]]などのウイルスにも見られ<ref>{{Cite web|title=Definition of Operon|url=http://www.medterms.com/script/main/art.asp?articlekey=32917|website=Medical Dictionary|publisher=MedicineNet.com|accessdate=30 December 2012}}</ref><ref name="Liu_2004">{{Cite journal|date=July 2004|title=Displacements of prohead protease genes in the late operons of double-stranded-DNA bacteriophages|journal=Journal of Bacteriology|volume=186|issue=13|pages=4369–75| |
オペロンは[[ファージ|バクテリオファージ]]などのウイルスにも見られ<ref>{{Cite web|title=Definition of Operon|url=http://www.medterms.com/script/main/art.asp?articlekey=32917|website=Medical Dictionary|publisher=MedicineNet.com|accessdate=30 December 2012}}</ref><ref name="Liu_2004">{{Cite journal|date=July 2004|title=Displacements of prohead protease genes in the late operons of double-stranded-DNA bacteriophages|journal=Journal of Bacteriology|volume=186|issue=13|pages=4369–75|doi=10.1128/JB.186.13.4369-4375.2004|pmid=15205439|pmc=421614}}</ref> たとえば、 [[T7ファージ|T7ファージに]]は2つのオペロンがある。最初のオペロンは、2番目のオペロンに結合して転写できる特別なT7 RNAポリメラーゼを含むさまざまな製品をコード。2番目のオペロンには、宿主細胞をバーストさせるための[[溶菌|溶解]]遺伝子が含まれている<ref>{{Cite web|title=Bacteriophage Use Operons|url=http://www.dartmouth.edu/~cbbc/courses/bio4/bio4-lectures/ProkGeneControl.html|website=Prokaryotic Gene Control|publisher=Dartmouth College|accessdate=30 December 2012}}</ref>。 |
||
== 歴史 == |
== 歴史 == |
||
用語「オペロン」は最初の議事録に短い論文で提案した[[科学アカデミー (フランス)|科学のフランスのアカデミー]] 1960年に<ref name="Jacob1960">{{Cite journal|date=February 1960|title=[Operon: a group of genes with the expression coordinated by an operator]|url=http://www.weizmann.ac.il/complex/tlusty/courses/landmark/JacobMonod1960.pdf|journal=Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences|volume=250|issue=6|pages=1727–9|language=French| |
用語「オペロン」は最初の議事録に短い論文で提案した[[科学アカデミー (フランス)|科学のフランスのアカデミー]] 1960年に<ref name="Jacob1960">{{Cite journal|date=February 1960|title=[Operon: a group of genes with the expression coordinated by an operator]|url=http://www.weizmann.ac.il/complex/tlusty/courses/landmark/JacobMonod1960.pdf|journal=Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences|volume=250|issue=6|pages=1727–9|language=French|pmid=14406329}}</ref> この論文から、いわゆるオペロンの一般理論が開発された。この理論は、すべての場合において、オペロン内の遺伝子は、最初の遺伝子の前に位置する単一の[[オペロン]]で作用する[[リプレッサー]]によって負に制御されることを示唆した。後に、遺伝子が積極的に調節され、転写開始に続くステップで調節されることも発見された。したがって、異なるオペロンには異なるメカニズムがあるため、一般的な規制メカニズムについて話すことはできない。今日、オペロンは、単一のmRNA分子に転写される遺伝子のクラスターとして単純に定義されている。それにもかかわらず、概念の開発は分子生物学の歴史の画期的なイベントと見なされる。記載される最初のオペロンは、 ''[[大腸菌|大腸菌の]]'' [[ラクトースオペロン|lacオペロン]]でした。オペロンは[[ゲノム]]上に存在する機能的な単位のひとつであり、発現制御機構の概念として[[フランソワ・ジャコブ]]と[[ジャック・モノー]]によってその存在が示唆された。彼らは[[大腸菌]]を用いた[[遺伝学]]的解析を通して、[[ラクトース]]代謝系の[[構造遺伝子]]群とその発現を制御する[[塩基配列]]部分とを合わせて一つの単位と考え、このような単位をオペロンと呼んだ(詳しくは[[ラクトースオペロン]]の項を参照)。彼らは[[アンドレ・ルヴォフ]]と共に[[1965年]]、[[ノーベル生理学・医学賞]]を受けた。 |
||
== 概要 == |
== 概要 == |
||
| 29行目: | 29行目: | ||
オペロンは主に[[原核生物|原核生物で]]発生が、 [[カエノラブディティス・エレガンス|''C。elegans'']]や<nowiki><i id="mwTQ">ショウジョウバエ</i></nowiki>などの[[線形動物|線虫]]を含む一部の[[真核生物]]でも発生。[[リボソームRNA|rRNA]]遺伝子は、 [[脊索動物]]を含む真核生物の範囲で見つかったオペロンにしばしば存在。オペロンは、共通の[[プロモーター]]下に配置され、共通のオペレーターによって制御されるいくつかの[[構造遺伝子]]で構成されている。これは、隣接する構造遺伝子のセットに加えて、構造遺伝子の転写に影響する隣接する調節シグナルとして定義される<ref>{{Cite book|title=Introduction to genetic analysis|url=https://archive.org/details/solutionsmanualf0000fixs_u8x1|publisher=W.H. Freeman|location=San Francisco|year=2005|pages=740|isbn=978-0-7167-4939-4|edition=8th}}</ref>。<sup>5</sup> [[リプレッサー|抑制]] 因子 、 共 [[リプレッサー|抑制]] 因子 、および活性化因子を含む、特定のオペロンの調節因子は、そのオペロンによって必ずしもコード化されているわけではない。レギュレーター、プロモーター、オペレーター、および構造DNA配列の位置と状態は、一般的な変異の影響を決定できる。 |
オペロンは主に[[原核生物|原核生物で]]発生が、 [[カエノラブディティス・エレガンス|''C。elegans'']]や<nowiki><i id="mwTQ">ショウジョウバエ</i></nowiki>などの[[線形動物|線虫]]を含む一部の[[真核生物]]でも発生。[[リボソームRNA|rRNA]]遺伝子は、 [[脊索動物]]を含む真核生物の範囲で見つかったオペロンにしばしば存在。オペロンは、共通の[[プロモーター]]下に配置され、共通のオペレーターによって制御されるいくつかの[[構造遺伝子]]で構成されている。これは、隣接する構造遺伝子のセットに加えて、構造遺伝子の転写に影響する隣接する調節シグナルとして定義される<ref>{{Cite book|title=Introduction to genetic analysis|url=https://archive.org/details/solutionsmanualf0000fixs_u8x1|publisher=W.H. Freeman|location=San Francisco|year=2005|pages=740|isbn=978-0-7167-4939-4|edition=8th}}</ref>。<sup>5</sup> [[リプレッサー|抑制]] 因子 、 共 [[リプレッサー|抑制]] 因子 、および活性化因子を含む、特定のオペロンの調節因子は、そのオペロンによって必ずしもコード化されているわけではない。レギュレーター、プロモーター、オペレーター、および構造DNA配列の位置と状態は、一般的な変異の影響を決定できる。 |
||
オペロンはレギュロン 、 刺激剤およびモジュロンに関連している;オペロンには同じオペレーターによって調節される一連の遺伝子が含まれているが、レギュロンには単一の調節タンパク質による調節下にある一連の遺伝子が含まれており、刺激には単一細胞刺激による調節下にある一連の遺伝子が含まれている。著者によると、「オペロン」という用語は「操作する」という動詞に由来している<ref>{{Cite journal|author-link=François Jacob|date=May 2011|title=The birth of the operon|journal=Science|volume=332|issue=6031|pages=767|bibcode=2011Sci...332..767J| |
オペロンはレギュロン 、 刺激剤およびモジュロンに関連している;オペロンには同じオペレーターによって調節される一連の遺伝子が含まれているが、レギュロンには単一の調節タンパク質による調節下にある一連の遺伝子が含まれており、刺激には単一細胞刺激による調節下にある一連の遺伝子が含まれている。著者によると、「オペロン」という用語は「操作する」という動詞に由来している<ref>{{Cite journal|author-link=François Jacob|date=May 2011|title=The birth of the operon|journal=Science|volume=332|issue=6031|pages=767|bibcode=2011Sci...332..767J|doi=10.1126/science.1207943|pmid=21566161}}</ref>。 |
||
== 大腸菌オペロンの種類 == |
== 大腸菌オペロンの種類 == |
||
| 42行目: | 42行目: | ||
== 転写の単位として == |
== 転写の単位として == |
||
オペロンには、一般に1つのポリシストロン性 [[伝令RNA|mRNA]] (複数の[[タンパク質]]をコードする単一のmRNA分子)に転写される1つ以上の[[構造遺伝子]]が含まれている。ただし、オペロンの定義では、mRNAがポリシストロン性である必要はないが、実際には通常はそう<ref name="Operons in eukaryotes">{{Cite journal|date=November 2004|title=Operons in eukaryotes|journal=Briefings in Functional Genomics & Proteomics|volume=3|issue=3|pages=199–211| |
オペロンには、一般に1つのポリシストロン性 [[伝令RNA|mRNA]] (複数の[[タンパク質]]をコードする単一のmRNA分子)に転写される1つ以上の[[構造遺伝子]]が含まれている。ただし、オペロンの定義では、mRNAがポリシストロン性である必要はないが、実際には通常はそう<ref name="Operons in eukaryotes">{{Cite journal|date=November 2004|title=Operons in eukaryotes|journal=Briefings in Functional Genomics & Proteomics|volume=3|issue=3|pages=199–211|doi=10.1093/bfgp/3.3.199|pmid=15642184}}</ref>。 構造遺伝子の上流には、 [[RNAポリメラーゼ]]が結合して転写を開始する部位を提供する[[プロモーター]]配列がある。プロモーターの近くには、 ''オペレーター''と呼ばれるDNAのセクションがある。 |
||
== オペロンと原核生物遺伝子のクラスター化 == |
== オペロンと原核生物遺伝子のクラスター化 == |
||
オペロンのすべての[[構造遺伝子]]は、それらの上流にある単一のプロモーターとオペレーターのために、一緒にオンまたはオフになるが、遺伝子発現をさらに制御する必要がある場合がある。この側面を実現するために、いくつかの細菌遺伝子は近くに配置されているが、それぞれに特定のプロモーターがある。これは遺伝子クラスタリングと呼ばれる。通常、これらの遺伝子は、代謝経路などの同じ経路で一緒に機能するタンパク質をコード。遺伝子クラスタリングは、原核細胞が代謝酵素を正しい順序で生産するのに役立つ<ref>{{Cite journal|date=May 2003|title=Genomic gene clustering analysis of pathways in eukaryotes|journal=Genome Research|volume=13|issue=5|pages=875–82| |
オペロンのすべての[[構造遺伝子]]は、それらの上流にある単一のプロモーターとオペレーターのために、一緒にオンまたはオフになるが、遺伝子発現をさらに制御する必要がある場合がある。この側面を実現するために、いくつかの細菌遺伝子は近くに配置されているが、それぞれに特定のプロモーターがある。これは遺伝子クラスタリングと呼ばれる。通常、これらの遺伝子は、代謝経路などの同じ経路で一緒に機能するタンパク質をコード。遺伝子クラスタリングは、原核細胞が代謝酵素を正しい順序で生産するのに役立つ<ref>{{Cite journal|date=May 2003|title=Genomic gene clustering analysis of pathways in eukaryotes|journal=Genome Research|volume=13|issue=5|pages=875–82|doi=10.1101/gr.737703|pmid=12695325|pmc=430880}}</ref>。 |
||
== 一般的な構造 == |
== 一般的な構造 == |
||
| 83行目: | 83行目: | ||
オペロンの数と構成は、 ''[[大腸菌|大腸菌で]]''最も批判的に研究されている。その結果、生物のゲノム配列に基づいて予測を行うことができる。 |
オペロンの数と構成は、 ''[[大腸菌|大腸菌で]]''最も批判的に研究されている。その結果、生物のゲノム配列に基づいて予測を行うことができる。 |
||
1つの予測方法では、読み取りフレーム間の遺伝子間距離を、ゲノム内のオペロン数の主要な予測因子として使用。分離は単にフレームを変更し、読み取りが効率的であることを保証。オペロンが開始および停止する場所に長いストレッチが存在し、多くの場合、最大40〜50塩基<ref>{{Cite journal|date=June 2000|title=Operons in Escherichia coli: genomic analyses and predictions|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=97|issue=12|pages=6652–7|bibcode=2000PNAS...97.6652S| |
1つの予測方法では、読み取りフレーム間の遺伝子間距離を、ゲノム内のオペロン数の主要な予測因子として使用。分離は単にフレームを変更し、読み取りが効率的であることを保証。オペロンが開始および停止する場所に長いストレッチが存在し、多くの場合、最大40〜50塩基<ref>{{Cite journal|date=June 2000|title=Operons in Escherichia coli: genomic analyses and predictions|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=97|issue=12|pages=6652–7|bibcode=2000PNAS...97.6652S|doi=10.1073/pnas.110147297|pmid=10823905|pmc=18690}}</ref>。 |
||
オペロンを予測する代替方法は、2つ以上のゲノムで遺伝子の順序と方向が保存されている遺伝子クラスターを見つけることに基づいている<ref>{{Cite journal|date=March 2001|title=Prediction of operons in microbial genomes|journal=Nucleic Acids Research|volume=29|issue=5|pages=1216–21| |
オペロンを予測する代替方法は、2つ以上のゲノムで遺伝子の順序と方向が保存されている遺伝子クラスターを見つけることに基づいている<ref>{{Cite journal|date=March 2001|title=Prediction of operons in microbial genomes|journal=Nucleic Acids Research|volume=29|issue=5|pages=1216–21|doi=10.1093/nar/29.5.1216|pmid=11222772|pmc=29727}}</ref>。 |
||
分子の機能クラスを考慮すれば、オペロンの予測はさらに正確になる。細菌は、タンパク質複合体、共通の経路、または共有された基質とトランスポーターへの共関与によって隔離されたユニットにリーディングフレームをクラスター化している。したがって、正確な予測にはこれらのデータすべてが関係することになり、実際に困難な作業になる。 |
分子の機能クラスを考慮すれば、オペロンの予測はさらに正確になる。細菌は、タンパク質複合体、共通の経路、または共有された基質とトランスポーターへの共関与によって隔離されたユニットにリーディングフレームをクラスター化している。したがって、正確な予測にはこれらのデータすべてが関係することになり、実際に困難な作業になる。 |
||
[[パスカル・コサール|Pascale Cossart]]の研究室は、微生物である''[[リステリア・モノサイトゲネス|Listeria monocytogenesの]]''すべてのオペロンを実験的に同定した最初の研究所。517のポリシストロン性オペロンは、さまざまな条件下で''L.モノサイトゲネス''で発生する転写のグローバルな変化を説明する2009年の研究にリストされている<ref>{{Cite journal|date=June 2009|title=The Listeria transcriptional landscape from saprophytism to virulence|journal=Nature|volume=459|issue=7249|pages=950–6|bibcode=2009Natur.459..950T| |
[[パスカル・コサール|Pascale Cossart]]の研究室は、微生物である''[[リステリア・モノサイトゲネス|Listeria monocytogenesの]]''すべてのオペロンを実験的に同定した最初の研究所。517のポリシストロン性オペロンは、さまざまな条件下で''L.モノサイトゲネス''で発生する転写のグローバルな変化を説明する2009年の研究にリストされている<ref>{{Cite journal|date=June 2009|title=The Listeria transcriptional landscape from saprophytism to virulence|journal=Nature|volume=459|issue=7249|pages=950–6|bibcode=2009Natur.459..950T|doi=10.1038/nature08080|pmid=19448609}}</ref>。 |
||
==脚注== |
==脚注== |
||
2020年1月25日 (土) 03:15時点における版
オペロン (Operon) とは
- 一つの形質を発現させる遺伝単位[1]。本項ではこれについて述べる。
- 東レ・デュポン社の登録商標で、ポリウレタン素材のブランド名でもあった。伸縮性に富んだ繊維が特徴で、1999年に米国デュポン社のポリウレタン素材のブランドであるライクラ (Lycra)と統一された。

オペロン (Operon) とは、一つの転写因子によって同時に発現が制御される複数の遺伝子が存在するゲノム上の領域、あるいはその複数の遺伝子の組のことである。
遺伝学では、 オペロンは単一のプロモーター制御下にある遺伝子のクラスターを含むDNAの機能単位[2]。 遺伝子は一緒に転写されてmRNA鎖になり、細胞質で一緒に翻訳されるか、またはスプライシングを受けて別々に翻訳されるモノシストロン性 mRNA、すなわちそれぞれが単一の遺伝子産物をコードするmRNAのいくつかの鎖を作成。この結果、オペロンに含まれる遺伝子は一緒に発現されるか、まったく発現されない。オペロンを定義するには、いくつかの遺伝子を同時転写する必要がある[3]。
もともと、オペロンは原核生物 ( バクテリアに由来する色素体のような細胞小器官を含む)にのみ存在すると考えられていたが、1990年代初期に真核生物で最初のオペロンが発見されて以来[4] [5]世代への原核生物オペロンリードの一般的には、表現ポリシストロン性 mRNAを、真核生物のオペロンは、単シストロン性のmRNAにつながるながらも以前に想定されていたよりも一般的[6]。
オペロンはバクテリオファージなどのウイルスにも見られ[7][8] たとえば、 T7ファージには2つのオペロンがある。最初のオペロンは、2番目のオペロンに結合して転写できる特別なT7 RNAポリメラーゼを含むさまざまな製品をコード。2番目のオペロンには、宿主細胞をバーストさせるための溶解遺伝子が含まれている[9]。
歴史
用語「オペロン」は最初の議事録に短い論文で提案した科学のフランスのアカデミー 1960年に[10] この論文から、いわゆるオペロンの一般理論が開発された。この理論は、すべての場合において、オペロン内の遺伝子は、最初の遺伝子の前に位置する単一のオペロンで作用するリプレッサーによって負に制御されることを示唆した。後に、遺伝子が積極的に調節され、転写開始に続くステップで調節されることも発見された。したがって、異なるオペロンには異なるメカニズムがあるため、一般的な規制メカニズムについて話すことはできない。今日、オペロンは、単一のmRNA分子に転写される遺伝子のクラスターとして単純に定義されている。それにもかかわらず、概念の開発は分子生物学の歴史の画期的なイベントと見なされる。記載される最初のオペロンは、 大腸菌の lacオペロンでした。オペロンはゲノム上に存在する機能的な単位のひとつであり、発現制御機構の概念としてフランソワ・ジャコブとジャック・モノーによってその存在が示唆された。彼らは大腸菌を用いた遺伝学的解析を通して、ラクトース代謝系の構造遺伝子群とその発現を制御する塩基配列部分とを合わせて一つの単位と考え、このような単位をオペロンと呼んだ(詳しくはラクトースオペロンの項を参照)。彼らはアンドレ・ルヴォフと共に1965年、ノーベル生理学・医学賞を受けた。
概要
原義
この研究で提案されたオペロンという考え方では、生物の染色体は、一群の機能的に関連した構造遺伝子をまとめた領域(オペロン)をつなぎ合わせた構造をもち、オペロンを一括して発現調節しているとされる。この考えをオペロン説という。一方で、Jacob がオペロンを形容する際に、"[ . . .] operons containig one or more genes [ . . . ]" といったように、複数の構造遺伝子が1分子の RNA へとまとめて転写されるということは、重要な特徴ではあったがオペロンの必須条件ではなかった (Jacob; 1965 (pdf))。加えると、当時はオペレーターが DNA、RNA、蛋白質のどの段階で働くのか明確にはされておらず、ラクトース代謝酵素群も一分子種の mRNA から翻訳されると証明されてはいなかった。従って、遺伝子の発現はどのように制御されているのか、つまりラクトースオペロンの項で説明されるような塩基配列成分の構成が、オペロンという概念の最も重要な点のひとつだったと言える。
概念の一般化
ところが、その後遺伝子発現の制御の研究が進むに従って、遺伝子発現が転写の段階で調節されるということは至極ありふれた事象となり、これを取り立ててオペロンと呼称することは少なくなった。これはオペロンが普遍的な価値を持つ概念だったためだが、と同時にいささか気の抜ける発音を要求することの不幸な結末かもしれない。さらに、単一プロモーターによって転写された一次転写産物から、複数の遺伝子産物が由来することにのみ着目された結果、オペロンはおもに原核生物に見られ真核生物には基本的に存在しない、と言われるようになる。つまり、この時のオペロンは複数の遺伝子産物を支配していることが必要条件となる。真核生物の例外として、線虫類に多く存在するオペロンとはこちらのことであり、 C. elegans では全遺伝子数の1/4程度がオペロンとして転写されることが知られている。この場合それぞれの遺伝子産物はプロセシングを受けた別々の mRNA 分子から翻訳される。これは、一分子の mRNA から複数種の蛋白質が複数の翻訳開始点から翻訳されるという原核生物の機構とは異なっている。また、これらの転写産物に機能的な関連性があるとは限らない点も異なる。
現在の状況としては、原義で言うところの1遺伝子のみからなるオペロンは遺伝子と呼び、構造遺伝子部分はコーディング・リージョンと呼ぶのが比較的正確かつ円滑な意思疎通を産むといえるのかもしれない。
オペロンは主に原核生物で発生が、 C。elegansや<i id="mwTQ">ショウジョウバエ</i>などの線虫を含む一部の真核生物でも発生。rRNA遺伝子は、 脊索動物を含む真核生物の範囲で見つかったオペロンにしばしば存在。オペロンは、共通のプロモーター下に配置され、共通のオペレーターによって制御されるいくつかの構造遺伝子で構成されている。これは、隣接する構造遺伝子のセットに加えて、構造遺伝子の転写に影響する隣接する調節シグナルとして定義される[11]。5 抑制 因子 、 共 抑制 因子 、および活性化因子を含む、特定のオペロンの調節因子は、そのオペロンによって必ずしもコード化されているわけではない。レギュレーター、プロモーター、オペレーター、および構造DNA配列の位置と状態は、一般的な変異の影響を決定できる。
オペロンはレギュロン 、 刺激剤およびモジュロンに関連している;オペロンには同じオペレーターによって調節される一連の遺伝子が含まれているが、レギュロンには単一の調節タンパク質による調節下にある一連の遺伝子が含まれており、刺激には単一細胞刺激による調節下にある一連の遺伝子が含まれている。著者によると、「オペロン」という用語は「操作する」という動詞に由来している[12]。
大腸菌オペロンの種類
大腸菌には75程度のこうしたオペロンが存在するが、以下の2種類に分けられる。
- 誘導オペロン:ラクトースオペロンのような機能を持つ
- 抑制オペロン:トリプトファンオペロンなど(しくみは以下に述べる)
抑制オペロンは、生合成経路の酵素群をコードしており、その産物によって発現調節を受ける。簡単に、調節ステップを述べる。トリプトファンオペロンの構造遺伝子はトリプトファンリプレッサーによって調節を受ける。しかしながら、トリプトファンリプレッサーは単体ではオペレーター部位に結合することができず、トリプトファン存在下でトリプトファンリプレッサー複合体を作って、初めてオペレーターに結合する。
すなわち、先ほど述べたラクトースオペロンによく似るが、異なるのはリプレッサーが複合体を作った後に、リプレッサーとして機能するかどうかの違いである。このリプレッサーの性質の違いが、異なる2つのオペロンを見分ける方法である。
転写の単位として
オペロンには、一般に1つのポリシストロン性 mRNA (複数のタンパク質をコードする単一のmRNA分子)に転写される1つ以上の構造遺伝子が含まれている。ただし、オペロンの定義では、mRNAがポリシストロン性である必要はないが、実際には通常はそう[6]。 構造遺伝子の上流には、 RNAポリメラーゼが結合して転写を開始する部位を提供するプロモーター配列がある。プロモーターの近くには、 オペレーターと呼ばれるDNAのセクションがある。
オペロンと原核生物遺伝子のクラスター化
オペロンのすべての構造遺伝子は、それらの上流にある単一のプロモーターとオペレーターのために、一緒にオンまたはオフになるが、遺伝子発現をさらに制御する必要がある場合がある。この側面を実現するために、いくつかの細菌遺伝子は近くに配置されているが、それぞれに特定のプロモーターがある。これは遺伝子クラスタリングと呼ばれる。通常、これらの遺伝子は、代謝経路などの同じ経路で一緒に機能するタンパク質をコード。遺伝子クラスタリングは、原核細胞が代謝酵素を正しい順序で生産するのに役立つ[13]。
一般的な構造
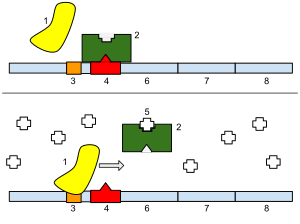
オペロンは、3つの基本的なDNAで構成されている。
- プロモーター –遺伝子の転写を可能にするヌクレオチド配列。プロモーターは、 RNAポリメラーゼによって認識され、転写を開始。RNA合成では、プロモーターはメッセンジャーRNAの作成にどの遺伝子を使用する必要があるかを示し、さらに細胞が生成するタンパク質を制御
- オペレーター – リプレッサーが結合するDNAのセグメント。lacオペロンでは、 オペロンのプロモーターと遺伝子間のセグメントとして古典的に定義されている[14]。 lacオペロンのメインオペレーター(O1)は、プロモーターの少し下流にある。2つの追加演算子、O1とO3は、それぞれ-82と+412にある。リプレッサーの場合、リプレッサータンパク質はRNAポリメラーゼが遺伝子を転写することを物理的に妨害
- 構造遺伝子 –オペロンによって共調節される遺伝子
オペロンに常に含まれるわけではないが、その機能において重要なのは、 抑制遺伝子を抑制している遺伝子である調節遺伝子。制御遺伝子は、オペロンを制御するためにオペロンの中、隣接、または近くにある必要はない[15]。
インデューサー (小分子)は、オペレーターサイト(DNA)からリプレッサー(タンパク質)を置き換えて、抑制されていないオペロンをもたらすことができる。
あるいは、 コリプレッサーはリプレッサーに結合して、オペレーター部位への結合を可能にすることができる。このタイプの規制の良い例は、 trpオペロンで見られる。
規制
オペロンの制御は、生物が環境条件に応じてさまざまな遺伝子の発現を調節できるようにする遺伝子調節の一種。オペロン制御は、誘導または抑制によって負または正のいずれかになる[14]。
ネガティブ制御には、転写を防ぐためのリプレッサーのオペレーターへの結合が含まれる。
- 負の誘導性オペロンでは、通常、調節リプレッサータンパク質がオペレーターに結合しており、オペロン上の遺伝子の転写を防ぐ。インデューサー分子が存在する場合、リプレッサーに結合し、そのコンフォメーションを変更して、オペレーターに結合できないように。これにより、オペロンの表現が可能になる。lacオペロンは負に制御された誘導性オペロンであり、誘導分子はアロラクトース
- 負の抑制可能なオペロンでは、通常、オペロンの転写が行われます。リプレッサータンパク質はレギュレーター遺伝子によって産生されるが、通常のコンフォメーションではオペレーターに結合できない。しかし、コリプレッサーと呼ばれる特定の分子はリプレッサータンパク質に結合され、活性部位に立体構造の変化を引き起こ。活性化されたリプレッサータンパク質はオペレーターに結合し、転写を防ぎます。トリプトファン (それ自体がコリプレッサーとして機能する)の合成に関与するtrpオペロンは、ネガティブに制御された抑制可能なオペロン 。
オペロンも積極的に制御できる。ポジティブコントロールでは、 アクチベータータンパク質はDNAに結合することにより転写を刺激(通常はオペレーター以外の部位で)。
- 正の誘導性オペロンでは 、活性化タンパク質は通常、関連するDNAに結合できない。インデューサーがアクチベータータンパク質に結合すると、コンフォメーションが変化するため、DNAに結合して転写を活性化できる
- 正の抑制可能なオペロンでは、活性化タンパク質は通常、関連するDNAセグメントに結合している。ただし、 インヒビターがアクチベーターと結合すると、DNAとの結合が妨げられる。これにより、システムのアクティベーションとトランスクリプションが停止する。
ラックオペロン
モデル細菌大腸菌のlacオペロンは、発見された最初のオペロンであり、オペロン機能の典型的な例を提供。これは、3つの隣接する構造遺伝子 、 プロモーター 、 ターミネーター 、およびオペレーターで構成されます。lacオペロンは、 グルコースとラクトースの入手可能性を含むいくつかの要因によって規制されている。アロラクトースによって活性化できる。ラクトースはリプレッサータンパク質に結合し、遺伝子転写の抑制を防ぎます。これは、 抑制不可能な (上から:負の誘導可能)モデルの例。
TRPオペロン
ジャック・モノドと同僚によって1953年に発見された大腸菌の trpオペロンは、発見された最初の抑圧可能なオペロンでした。lacオペロンは化学物質( アロラクトース )によって活性化できるが、トリプトファン(Trp)オペロンは化学物質(トリプトファン)によって阻害される。このオペロンには、trp E、trp D、trp C、trp B、およびtrp Aの5つの構造遺伝子が含まれている。これらは、 トリプトファン合成酵素をエンコード。また、RNAポリメラーゼに結合するプロモーターと、オペレーターに結合するリプレッサー遺伝子(trp R)によって合成されたタンパク質に結合すると転写をブロックするオペレーターも含まれる。lacオペロンでは、ラクトースはリプレッサータンパク質に結合して遺伝子転写を抑制しません。一方、trpオペロンでは、トリプトファンはリプレッサータンパク質に結合して遺伝子転写を抑制。また、lacオペロンとは異なり、trpオペロンには、段階的な調節を可能にするリーダーペプチドと減衰配列が含まれている[16]。 これは、 コアプレス可能なモデルの例。
オペロンの数と組織の予測
オペロンの数と構成は、 大腸菌で最も批判的に研究されている。その結果、生物のゲノム配列に基づいて予測を行うことができる。
1つの予測方法では、読み取りフレーム間の遺伝子間距離を、ゲノム内のオペロン数の主要な予測因子として使用。分離は単にフレームを変更し、読み取りが効率的であることを保証。オペロンが開始および停止する場所に長いストレッチが存在し、多くの場合、最大40〜50塩基[17]。
オペロンを予測する代替方法は、2つ以上のゲノムで遺伝子の順序と方向が保存されている遺伝子クラスターを見つけることに基づいている[18]。
分子の機能クラスを考慮すれば、オペロンの予測はさらに正確になる。細菌は、タンパク質複合体、共通の経路、または共有された基質とトランスポーターへの共関与によって隔離されたユニットにリーディングフレームをクラスター化している。したがって、正確な予測にはこれらのデータすべてが関係することになり、実際に困難な作業になる。
Pascale Cossartの研究室は、微生物であるListeria monocytogenesのすべてのオペロンを実験的に同定した最初の研究所。517のポリシストロン性オペロンは、さまざまな条件下でL.モノサイトゲネスで発生する転写のグローバルな変化を説明する2009年の研究にリストされている[19]。
脚注
- ^ デジタル大辞泉
- ^ Sadava, David E.; Hillis, David M.; Heller, H. Craig; Berenbaum, May (2009). Life: The Science of Biology (9th ed.). Macmillan. p. 349. ISBN 978-1-4292-1962-4
- ^ Lodish, Harvey; Zipursky, Lawrence; Matsudaira, Paul; Baltimore, David; Darnel, James (2000). “Chapter 9: Molecular Definition of a Gene”. Molecular Cell Biology. W. H. Freeman. ISBN 978-0-7167-3136-8
- ^ “Operons in C. elegans: polycistronic mRNA precursors are processed by trans-splicing of SL2 to downstream coding regions”. Cell 73 (3): 521–32. (May 1993). doi:10.1016/0092-8674(93)90139-H. PMID 8098272.
- ^ “The Adh-related gene of Drosophila melanogaster is expressed as a functional dicistronic messenger RNA: multigenic transcription in higher organisms”. The EMBO Journal 16 (8): 2023–31. (April 1997). doi:10.1093/emboj/16.8.2023. PMC 1169805. PMID 9155028.
- ^ a b “Operons in eukaryotes”. Briefings in Functional Genomics & Proteomics 3 (3): 199–211. (November 2004). doi:10.1093/bfgp/3.3.199. PMID 15642184.
- ^ “Definition of Operon”. Medical Dictionary. MedicineNet.com. 2012年12月30日閲覧。
- ^ “Displacements of prohead protease genes in the late operons of double-stranded-DNA bacteriophages”. Journal of Bacteriology 186 (13): 4369–75. (July 2004). doi:10.1128/JB.186.13.4369-4375.2004. PMC 421614. PMID 15205439.
- ^ “Bacteriophage Use Operons”. Prokaryotic Gene Control. Dartmouth College. 2012年12月30日閲覧。
- ^ “[Operon: a group of genes with the expression coordinated by an operator”] (French). Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences 250 (6): 1727–9. (February 1960). PMID 14406329.
- ^ Introduction to genetic analysis (8th ed.). San Francisco: W.H. Freeman. (2005). pp. 740. ISBN 978-0-7167-4939-4
- ^ “The birth of the operon”. Science 332 (6031): 767. (May 2011). Bibcode: 2011Sci...332..767J. doi:10.1126/science.1207943. PMID 21566161.
- ^ “Genomic gene clustering analysis of pathways in eukaryotes”. Genome Research 13 (5): 875–82. (May 2003). doi:10.1101/gr.737703. PMC 430880. PMID 12695325.
- ^ a b Lewin, Benjamin (1990). Genes IV (4th ed.). Oxford: Oxford University Press. pp. 243–58. ISBN 978-0-19-854267-4
- ^ Mayer, Gene. “Bacteriology – Chapter Nine Genetic Regulatory Mechanisms”. Microbiology and Immunology Online. University of South Carolina School of Medicine 2012年12月30日閲覧。
- ^ Concepts of genetics (8th ed.). Upper Saddle River, NJ: Pearson Education. (2006). pp. 394–402. ISBN 978-0-13-191833-7
- ^ “Operons in Escherichia coli: genomic analyses and predictions”. Proceedings of the National Academy of Sciences of the United States of America 97 (12): 6652–7. (June 2000). Bibcode: 2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
- ^ “Prediction of operons in microbial genomes”. Nucleic Acids Research 29 (5): 1216–21. (March 2001). doi:10.1093/nar/29.5.1216. PMC 29727. PMID 11222772.
- ^ “The Listeria transcriptional landscape from saprophytism to virulence”. Nature 459 (7249): 950–6. (June 2009). Bibcode: 2009Natur.459..950T. doi:10.1038/nature08080. PMID 19448609.
外部リンク
- The Nobel Prize in Physiology or Medicine 1965
- 結核菌 H37Rvオペロン相関ブラウザ
- OBD -Operonデータベース(ただし、少し使いにくい)
参考文献
- * Jacob, F., 1965 Nobel lecture - Genetics of bacterial cell
Jacob, F., Monod, J. 1961. Genetic regulatory mechanisms in the synthesis of proteins. J. Mol. Biol. 3, 318-356.
