大腸菌
| 大腸菌 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 大腸菌の電子顕微鏡写真
| |||||||||||||||||||||
| 分類 | |||||||||||||||||||||
| |||||||||||||||||||||
| 学名 | |||||||||||||||||||||
| エスケリキア・コリ Escherichia coli (Migula 1895)[1] Castellani and Chalmers 1919[2] (IJSEMリストに掲載 1980)[3] | |||||||||||||||||||||
| タイプ株 | |||||||||||||||||||||
| ATCC 11775 CCUG 24 CCUG 29300 CIP 54.8 DSM 30083 JCM 1649 LMG 2092 NBIMCC 3398 NBRC 102203 NCCB 54008 NCTC 9001 | |||||||||||||||||||||
| シノニム | |||||||||||||||||||||
|
大腸菌(だいちょうきん、学名: Escherichia coli、発音:[ˌɛʃəˈrɪkiə ˈkoʊlaɪ])は、グラム陰性の桿菌で通性嫌気性菌に属し、環境中に存在する細菌(バクテリア)の主要な種の一つである。腸内細菌の一種でもあり、温血動物(鳥類、哺乳類)の下流の消化管内、特にヒトなどの場合は大腸に生息する。短縮表記はE. coli(発音:[ˌiː ˈkoʊlaɪ])。
大腸菌には非常に多数の株が存在する。大半の大腸菌株は無害であるが、その中には病原性を持つものも存在する。特に一部の血清型(EPEC、ETECなど)は宿主に深刻な食中毒を引き起こす可能性があり、製品のリコールを伴う食品汚染事故の原因となる場合がある[4][5]。無害な菌株は、腸内の正常な微生物叢(マイクロバイオーム)の一部を構成し、ビタミンK 2を生成して血液の凝固を助けたり[6]、腸内で病原菌のコロニー形成を防止する等、共生関係にある宿主に利益をもたらしうる[7][8]。
大腸菌および他の通性嫌気性菌は腸内微生物叢の約0.1%を構成する[9]。腸内の大腸菌は、糞便を通じて外環境に排出され、糞便から口腔への感染(糞口経路)は、細菌の病原性株が疾患を引き起こす主な経路となる。細胞は限られた時間、体外で生存することができる。排出された大腸菌は、好気性条件下で3日間、新鮮な糞便中で大量に増殖するが、その後は徐々に減少することが報告されている[10]。大腸菌は株ごとに異なる特徴を持ち、また異なる動物の腸内には異なる株の大腸菌が生息している。そのため、糞便汚染を検出するための潜在的な指標生物として利用されている[11][12]。例えば、環境水を汚染している糞便が人間から出たものか、鳥類から出たものかを推定することができる。一方で近年の研究から、宿主の外で何日も生存し増殖するような、環境的に持続的な大腸菌の存在が明らかになっている[13]。
大腸菌は実験室で簡単かつ安価に増殖および培養でき、原核生物のモデル生物の一つとして、60年以上に渡って徹底的に研究されてきた。大腸菌は化学合成生物(ヘテロトロフ)であり、炭素源とエネルギー源を含む化学的に定義された培地で培養することができる[14]。また大腸菌はバイオテクノロジーおよび微生物学の分野で重要な種であり、組換えDNAに基づく科学研究において宿主生物として多用されている。良好な培養条件下では、細胞分裂にわずか20分ほどしかかからない[15]。遺伝子を組み込むことで、化学物質の生産にも利用される。

生物学的・生化学的特徴
[編集]形態
[編集]大腸菌はグラム陰性の通性嫌気性菌であり、非胞子形成細菌である。酸素存在条件下では好気性呼吸によってATPを生成するが、酸素非存在下では発酵または嫌気性呼吸に切り替わる[16]。細胞は典型的には棒状であり、大きさは通常、短軸0.4-0.7μm、長軸2.0-4.0μm、直径0.25-1.0μm、細胞容積は0.6-0.7 μm3程度である[17][18]。長軸が短くなり球形に近いものもいる[19]。
大腸菌は、薄いペプチドグリカン層と外膜で細胞壁が構成されており、グラム染色は陰性である。グラム染色では、サフラニンの色を取り込むため、ピンクに染色される。細胞壁を囲む外膜は、特定の抗生物質に対するバリアとして機能し、例えばペニシリンによる損傷を防ぐ[20]。
大腸菌の株のうち、べん毛を持つものは運動性を持っている[21]。また、インチミンと呼ばれる接着分子を介して、腸の微絨毛に付着したり剥がれたりする[22]。
代謝
[編集]大腸菌はさまざまな基質を利用して生育できる。嫌気性条件下では、混合酸発酵によって乳酸やコハク酸、エタノール、酢酸、二酸化炭素を生産する。混合酸発酵の多くの経路では水素ガスを生成させるため、経路を進めるためには水素レベルを低く保っておく必要があり、例えばメタン生成菌や硫酸還元菌などの水素消費生物と共生している場合などが理想的である[23]。
さらに、大腸菌の代謝を人為的に操作することで、炭素源として二酸化炭素のみを利用させる事もできる。すなわち、本来は絶対従属栄養の形を取る代謝は、炭素固定遺伝子やギ酸脱水素酵素の異種発現という実験室進化を経ることで、独立栄養能力を示すように改変することができる、ということを示している。これは、ギ酸塩を使用して電子キャリアを減らし、同化経路に必要なアデノシン三リン酸(ATP)を供給することによって行うことができる[24]。
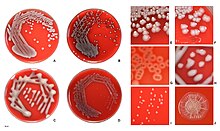
培養
[編集]
大腸菌の最適な増殖は37 ℃であるが、実験室株の中には49 ℃の温度でも増殖するものもいる[25]。大腸菌は、LB培養液(グルコース、リン酸アンモニウム、塩化ナトリウム、硫酸マグネシウム、リン酸カリウム、および水を含む)などの、成分が定義されたさまざまな任意の実験用培地を用いて増殖させることができる。細胞の成長と増殖は好気性または嫌気性呼吸によって促進される。その過程で、ピルビン酸、ギ酸、水素、アミノ酸等の酸化プロセスと、酸素、硝酸塩、フマル酸塩、ジメチルスルホキシド、トリメチルアミンN-オキシドなどの基質の還元プロセスといった、多種多様な酸化還元反応を利用している[26]。大腸菌は通性嫌気性菌に分類されるが、酸素が利用可能な状況下では酸素を利用する。一方で、酸素がない環境下では発酵または嫌気性呼吸を利用して成長し続けることができる。酸素が存在しなくても生育できる能力によって、例えば水中のような嫌気的な環境でも増殖できるためようになるため、これは生存に有利な能力である[27]。

細胞周期
[編集]
細胞周期は3つの段階に分かれている。B期は、細胞分裂の完了とDNA複製の開始との間に発生する。C期は、染色体DNAを複製するのにかかる時間を含む。D期は、DNA複製の終了と細胞分裂の終わりの間の段階を指す[28]。より多くの栄養素が利用可能である場合、大腸菌の倍加率はより高くなる。ただし、倍加時間がC期とD期の合計より短くなっても、C期とD期の長さ自体には変化はない。最速の成長率を示す状況下では、複製ラウンドが完了する前に次の複製が開始され、DNAに沿って複数の複製フォークが形成され、細胞周期が重複する[29]。
急速に成長する大腸菌の複製フォークの数は、通常2n(n=1、2、または3)である。これは同期複製と呼ばれ、複製が複製起点から同時に開始された場合にのみ発生する。ただし、培養物内の細胞は、全てが同期的に複製されるわけではない。複数ペアの複製フォークが存在しない細胞においては、複製開始は非同期になる[30]。この非同期は、たとえばDnaA [30]やDnaAイニシエーター関連タンパク質DiaAへの変異によって引き起こされている可能性がある[31]。
遺伝的適応
[編集]大腸菌および多くの関連する細菌は、接合や形質導入を介してDNAを転移する能力を持っており、これによって遺伝物質を既存の集団全体に水平的に広げることができる(遺伝子の水平伝播)。例えば志賀毒素をコードする遺伝子は、バクテリオファージと呼ばれるバクテリアウイルスを介した形質導入プロセスを通じて、赤痢菌から大腸菌に広がり、志賀毒素を持つ大腸菌 O157:H7が生まれたと考えられている[32]。
系統学的分類
[編集]大腸菌は、系統分類学的にはPseudomonadota門、ガンマプロテオバクテリア綱、エンテロバクター目、腸内細菌科に分類されている。しかしながら、大腸菌と呼ばれるグループの中には、非常に多様な遺伝的・表現型的形質が見られる。そのため、近年の大腸菌や関連細菌の分離株ゲノム配列決定に伴い、本来はこのグループを系統分類学的に再分類することが望ましいと考えられている[33]。しかしながら、主にその医学的重要性のために、再分類は行うことができておらず[33]、大腸菌は現在でも最も多様な細菌種の1つであり続けている。例えば赤痢菌属のメンバー(S. dysenteriae、S. flexneri、S. boydii、S. sonnei)は、本来なら大腸菌株として分類しなければならない[34]。同様に、他の大腸菌株(例えば、組換えDNAの研究で一般的に使用されるK-12株)は、再分類に値するほど十分に異なっている。典型的な大腸菌ゲノムの遺伝子のうち、すべての株で共有されているものはわずか20%程度である[35]。
菌株は、他の菌株と区別されるような、独特の特徴を持つ種内サブグループである。この株間の差異は、分子レベルでしか検出できないが、細菌の生理機能やライフサイクルに変化をもたらす、というようなことがよくある。たとえば、菌株は、病原性能力、独特の炭素源を利用する能力、特定の生態学的ニッチを獲得する能力、または抗菌剤に抵抗する能力を獲得する可能性がある。大腸菌の異なる株は、しばしば宿主特異的であり、環境サンプル中の糞便汚染の原因を特定することを可能にする[36][37]。たとえば、水のサンプルにどの大腸菌株が存在するかを知ることにより、汚染源が人間、他の哺乳類、鳥など、どの生物から発生したのかを推測することができる。
血清型
[編集]病原性との関連を重視して、菌の表面にある抗原(O抗原、H抗原、K抗原)にも基づいて細かく分類されている[38][39]。O抗原は外膜のリポ多糖由来のもの、H抗原はべん毛由来のもの、K抗原はカプセル(capsule)由来のものである。O抗原は現在約190種類ほどに分類されている[38][40]。例えば「O157(オーいちごーなな)」という名称は、O抗原としては157番目に発見されたものを持つ菌ということを意味しており[38]、「O111(オーいちいちいち)」はO抗原としては111番目に発見されたものを持つ、ということを意味する。H抗原は約70種類に分類されている。なお、さらに細かく分けるとO抗原とH抗原の両方を考慮した分類になる。例えばO157でも、H抗原に関する違いでさらに細かく分類することができ、H7のものとH抗原を持たないものがあるので、「O157:H7」と「O157:H-」という2種類に分けることができる[38]。一方で、一般的な実験室株はO抗原の形成を妨げる変異を持っているため、分類することはできない。
ゲノムの可塑性と進化
[編集]他のすべての生命体と同様に、大腸菌は突然変異や遺伝子重複、遺伝子の水平移動などの自然な生物学的プロセスを通じて進化する。特に、実験室株MG1655のゲノムの18%は、サルモネラからの分岐以降に水平的に取得されたものである[41]。E. coli K-12株およびE. coli B株は、実験目的で最も頻繁に利用される品種である。他の大腸菌のいくつかの株は、宿主動物に有害な形質を持つ。これらの毒性の強い株は通常、下痢の発作を引き起こす。下痢は、健康な成人では抑制的であるが、発展途上国の子供ではしばしば致命傷となる[42]。O157:H7などのより毒性の強い菌株は、高齢者、若年者、免疫不全の人などに深刻な病気や死を引き起こしうる[42][43]。
エシェリヒア属とサルモネラ属は約1億200万年前に分岐したと考えられている(信頼区間:57-176 mya)。これは、各細菌の宿主の分岐とよく一致している。すなわち、前者は哺乳類から発見され、後者は鳥や爬虫類から発見される細菌である[44]。この祖先細菌から、5種の大腸菌の祖先種(E. albertii、E. coli、E. fergusonii、E. hermannii、E. vulneris)が分岐したと考えられている。最後の大腸菌の祖先種は、2000万から3000万年前に分裂したと見積もられる[45]。
1988年にRichard Lenskiによって開始された、大腸菌を使用した長期進化実験により、研究室で65,000世代を超えるゲノム進化の直接観察が可能になった[46]。たとえば、大腸菌は通常、クエン酸を炭素源として好気性に増殖する能力を持たない。このことは、大腸菌をサルモネラ菌などの他の密接に関連する細菌から区別するための診断基準として使用される。しかしながらこの進化実験では、大腸菌の1つの集団が、好気的にクエン酸を代謝する能力を進化させることが確認された。これは、微生物の種分化を引き起こすような、主要な進化的シフトの特徴であると考えられる。
微生物の世界でも動物と同様に、捕食の関係が成立する。そして大腸菌は、Myxococcus xanthusのような複数のジェネラリスト捕食者の餌食であることが知られている。この捕食者と被食者の両種は並行進化していることが、ゲノムや表現型の変化の観察から考えられている。大腸菌の場合、ムコイド産生(アルギン酸エキソプラズマ酸の過剰産生)とOmpT遺伝子の抑制という、病原性に関与する2つの側面を伴う、赤の女王仮説で実証された共進化モデルに従って、他よりも適応的な進化個体が選択的に生き残ると考えられている[47]。
ネオタイプ株
[編集]E. coliはEscherichia属のタイプ種であり、Escherichia属はEnterobacteriaceae科のタイプ属である。この科名はEnterobacter+"i"(sic.)+"aceae"であり、"enterobacterium"+"aceae"ではない(Enterobacteriumは属名ではなく、腸内細菌の別名となっている)[48][49][50]。
本来は最初にEscherichと記載された原株がタイプ株とされるべきであるが、既に失われていると考えられている。そのため、その代替となる新しいタイプ株(ネオタイプ)が幾つか選択されている。具体的には、U5/41T(DSM 30083)[51][52]、ATCC 11775 [53]、およびNCTC 9001[54]である。なお、NCTC 9001株は鶏に病原性を持っており、O1:K1:H7血清型を持っている[55]。一方でほとんどの研究では、代表的な大腸菌としてO157:H7、K-12 MG1655、またはK-12 W3110のいずれかが使用される。タイプ株のゲノムは配列決定されている[51]。
大腸菌株の系統
[編集]今までの研究により、この種に属する多くの株が分離され、特徴付けられている。大腸菌は、血清型(上記参照)に加えて、系統学に6つのグループに分類される[56][57]。特に全ゲノム配列の情報を利用することで、高度に信頼性の高い系統関係を推定することが可能である。このようなゲノムデータに基づいて、大腸菌には5つの亜種が定義されている[58]。
系統学的距離は一方で、病理学な特徴とはあまり関連していない[59]。たとえばO157:H7血清型株は、グループEと形容されるクレードを形成し、すべて腸管出血性株(EHEC)に含まれるが、すべてのEHEC株が系統学的に密接に関連しているわけではない。実際、Shigella属の4つの異なる種が、大腸菌株の間に入れ子状になっている(上記を参照)。一方で、EHEC株であるE. albertiiおよびE. fergusoniiは、このグループの外に位置している。そして、タイプ株を含むすべての赤痢菌種は、大腸菌の単一亜種内に位置している[59]。このため、適切な再分類は困難な状況になっている。研究分野で一般的に利用されるすべての大腸菌研究株はグループAに属し、主にクリフトンのK-12株(λ⁺F⁺; O16)やデレーユのBacillus coli株(B株)(O7)に由来している。
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ゲノミクス
[編集]
大腸菌(実験室株K-12に由来するMG1655株)の最初の完全なDNA配列は1997年に公開された。大腸菌のゲノムは460万塩基対の長さの環状DNA分子であり、4,288の注釈付きタンパク質コーディング遺伝子(2584オペロンに編成)、7のリボソームRNA (rRNA)オペロン、86のトランスファーRNA (tRNA)遺伝子を含んでいた。それまでの約40年間、大腸菌は集中的な遺伝子分析の対象であったにもかかわらず、これらの遺伝子の多くは以前は知られていなかった。コーディング配列の密度は非常に高く、遺伝子間の平均距離はわずか118塩基対であることがわかった。ゲノムには、かなりの数のトランスポゾン、リピート反復配列、詳細不明なプロファージ、バクテリオファージの残骸、などが含まれていることが判明した[60]。
今日、Escherichia属とShigella属(赤痢菌)では、300を超える完全ゲノム配列が報告されている。大腸菌のタイプ株のゲノム配列は、2014年までにこのコレクションに追加されている[61]。これらのシーケンスの比較から、驚くべき多様性の存在が示唆されており、各ゲノムの約20%のみが全分離株に共通している一方で、約80%は分離株の各ゲノムによって異なっている[62]。個々のゲノムには、約4,000から5,500の遺伝子が含まれているが、シーケンスされたすべての大腸菌株(パンゲノム)が持つ全遺伝子(遺伝子カタログ)の総数は16,000を超えている。この非常に多種多様な構成遺伝子は、大腸菌のパンゲノムの2/3が他の種に由来し、水平遺伝子導入の過程で到達したことを意味している、と解釈されている[63]。
遺伝子の命名法
[編集]大腸菌の遺伝子は一般的には、機能が既知の場合、その機能に由来する4文字の頭字語によって斜体で示される。たとえばrecAは、相同組み換え(homologous recombination)におけるその役割+文字A、から名付けられた。機能的に関連する遺伝子は順に、recB、recC、recDなどと名付けられている。タンパク質の場合は、大文字の頭字語で名前が付けられている。例えばRecA、RecBなどであり、大腸菌ゲノムが配列決定された際に、すべての遺伝子がゲノム上で順番に番号が付けられ、b2819(=recD)などのb番号で表記された。この”b”の名前は、ゲノムシーケンスの取り組みを主導したFred B lattnerにちなんで作成された[60]。別の番号付けシステムは、日本でシーケンスされた別の大腸菌株W3110のシーケンスで導入され、JW(Japanese W3110)で始まる番号が使用されており、例えばJW2787(=recD)などである[64]。したがって、recD=b2819=JW2787となる。ただし、ほとんどのデータベースにおいて、それぞれに独自の番号付けシステムがあるため、注意が必要である。たとえばEcoGeneデータベース[65]では、recDにはEG10826が使用される。最後に、ECK番号は、特に大腸菌 K-12のMG1655株の対立遺伝子に使用される[65]。遺伝子とその同義語の完全なリストは、EcoGeneやUniprotなどのデータベースから取得することが可能である。
プロテオミクス
[編集]いくつかの研究において、大腸菌のプロテオームが調べられており、2006年までに4,237のオープンリーディングフレーム (ORF)のうち1,627(38%Iが実験的に特定されている[66]。大腸菌K-12株の4,639,221塩基対の配列中には、注釈が付けられた4,288個のタンパク質コード遺伝子があるが、そのうち38%は機能が特定されていない。シーケンスされた他の5つの微生物との比較により、広い系統間や狭い系統間、そして大腸菌内における類似遺伝子(遺伝子ファミリー)の分布を明らかにする研究が行われた。パラログ遺伝子によるタンパク質の最大のファミリーの一つとして、80個のABCトランスポーターが挙げられる。ゲノムは全体として、特に複製に関わるものについては、非常に体系化されている。ゲノムには、挿入配列(IS)要素、ファージの残骸、および水平移動によるゲノムの可塑性を示す異常な組成の他の多くのパッチも含まれている[60]。
インタラクトーム
[編集]大腸菌のインタラクトームは、アフィニティー精製と質量分析(AP/MS)を用いて、タンパク質間の相互作用の解析が進められている。
タンパク質複合体
[編集]2006年の研究では、K-12株の培養液から4,339種類のタンパク質を精製し、2,667種類のタンパク質の相互作用パートナーを報告している[67]。2009年の研究では、同じE. coli株のタンパク質間の5,993の相互作用を発見したが、これらのデータは2006年の論文に記載された相互作用と、ほとんど重複していなかった[68]。
バイナリインタラクション
[編集]Rajagopalaらは2014年に、ほとんどの大腸菌タンパク質を用いて体系的な酵母2ハイブリッドスクリーニングを実施し、合計2,234のタンパク質間相互作用を報告した[69]。この研究ではさらに、遺伝的相互作用とタンパク質構造を統合し、227のタンパク質複合体内における458の相互作用のマッピングも行った。
生命科学研究におけるモデル生物としての大腸菌
[編集]実験室培養の長い歴史とその操作のしやすさから、大腸菌は現代の生物工学と工業微生物学において重要な役割を果たしている[70]。例えば、Stanley Norman CohenとHerbert Boyerにより、プラスミドと制限酵素を使用して組換えDNAを作成する手法が考案され、バイオテクノロジーの基礎となった[71]。
大腸菌は異種タンパク質の生産を行う上でよく利用される宿主であり[72]、大腸菌での組換えタンパク質の生産を可能にするさまざまなタンパク質発現システムが開発されている。タンパク質の高レベル発現を可能にするプラスミドを使用して、大腸菌に遺伝子を導入することで、特定のタンパク質を工業的発酵プロセスで大量生産することが可能となる。大腸菌を用いた組換えDNA技術の最初の有用なアプリケーションの1つは、ヒトのインスリンの生産である[73]。
従来は大腸菌での発現が困難または不可能と思われていたタンパク質であっても、近年可能になりつつあるものも多く知られている。例えば、細胞膜周辺腔や細胞質で製造されると考えられている複数のジスルフィド結合を有するタンパク質は、ジスルフィド結合を可能にするように十分に酸化され[74]、グリコシル化などの翻訳後修飾が安定性や機能のために必要となるタンパク質は、Campylobacter jejuniが持つN-結合型グリコシル化システムを導入した大腸菌において発現された[75][76][77]。
改変された大腸菌細胞は、ワクチンの開発、バイオレメディエーション、バイオ燃料の生産[78]、酵素の生産などに利用されている[79][80]。例えば、K-12株は、酵素アルカリフォスファターゼ(ALP)を過剰発現する大腸菌の変異型である[81]。この変異は、この酵素をコードする遺伝子の欠陥が原因である。特に阻害作用を持たないタンパク質を生産している遺伝子は、構成的活性を持っていると言われています。この特定の変異型は、前述の酵素を単離および精製するために使用される[81]。またOP50株は、Caenorhabditis elegans培養の維持に使用される。他の例としては、recAとendAが欠損した大腸菌の変異型であるJM109株がある。この菌株が受精因子エピソームを持っている場合、この株は青/白のスクリーニングに利用できる[82]。recAの欠如は、余計なDNA断片の挿入制限の可能性を減らし、endAの欠如はプラスミドDNA分解を阻害するため、JM109はクローニングおよび発現系に有用である。
モデル生物
[編集]大腸菌は微生物学研究のモデル生物として頻繁に使用されている。培養された菌株(例えばK12株)は実験室環境によく適応しており、野生型株とは異なり腸内で繁殖する能力を失っている他、多くの実験室株は バイオフィルムを形成する能力も失っている[83][84]。これらの機能は、野生型株を抗体やその他の化学的攻撃から保護するが、一方でエネルギーと材料リソースに多大な支出を必要とする。大腸菌は、光触媒作用を含む新しい水処理や滅菌方法に関する研究においても、代表的な微生物としてよく使用されている。標準的なプレートカウント法、連続希釈、および寒天ゲルプレート上での増殖により、既知の処理水量における生菌またはCFU(コロニー形成単位)の濃度を評価でき、材料の性能を比較評価することができる[85]。
1946年に、ジョシュア・レーダーバーグ(Joshua Lederberg)とエドワード・テイタム(Edward Tatum)は、大腸菌をモデル細菌として使用し、細菌の接合として知られている現象を発見した[86][87]。大腸菌は、ファージの遺伝学を理解する最初の実験の不可欠な部分であり[88]、シーモア・ベンツァー(Seymour Benzer)などの初期の研究者は、大腸菌とファージT4を使用して遺伝子構造のトポグラフィーを解析した[89]。ベンツァーの研究以前は、遺伝子が線状構造であるのか、それとも分岐パターンがあるのかは不明であった[90]。
大腸菌は、ゲノムが配列決定された最初の生物の1つである。大腸菌K-12株の完全なゲノムは、Science誌上で1997年に公開された[60]。
2002年から2010年にかけて、ハンガリー科学アカデミーのチームがMDS42と呼ばれる大腸菌株を作成した。これは現在、ウィスコンシン州のScarab Genomics of Madison社から「Clean Genome. E.coli」という名前で販売されている[91]。この株では、親株(E. coli K-12 MG1655株)のゲノムの15%が除去され、分子生物学の効率を高めるようにIS要素や偽遺伝子およびファージを除去して、トランスポゾンによって不活化されることが多いプラスミドにコード化された毒性遺伝子の維持を改善したものである[92][93][94]。生化学および複製機構は変更されていない。
ナノテクノロジーとランドスケープエコロジーの可能な組み合わせを評価することにより、複雑な生息地ランドスケープをナノスケールの詳細で生成できる[95]。そのような合成生態系では、大腸菌を用いた進化的実験が行われ、島嶼生物地理学における適応の空間生物物理学をチップ上で再現する研究などが行われている。
応用利用
[編集]ヒトに対して、大腸菌の死骸を含んだ液体(大腸菌死菌浮遊液)が、直腸部に塗布されると、白血球が呼び寄せられるため、感染防御の役に立つことが知られており、これを利用した薬剤が実用化されている[96]。また、遺伝子組み換え技術を用いて、大腸菌にヒト型インスリンを作らせる遺伝子を導入して、インスリンを生産することに利用されている。他にも、顆粒球コロニー刺激因子 (G-CSF) や組織プラスミノーゲン活性化因子(t-PA)などの生産も、同様の方法で行われている。大腸菌に感光性を与えて撮像素子として利用できる研究も実施される[97]。
大腸菌をプログラムしてハミルトン経路問題などの複雑な数学問題を解決しようとする研究も行われている[98]。
非病原性大腸菌 Nissle 1917株(Mutaflor)およびO83:K24:H31株(Colinfant)[99][100]は、炎症性腸疾患を含むさまざまな胃腸疾患の治療のために、医薬品のプロバイオティック剤として使用されている[101][102]。
病原性
[編集]大腸菌は、温血動物の胃腸管で見られる大腸菌群として非公式に知られている細菌のグループに属している[103]。大腸菌は通常、食物や水を通じて、子供を扱う個人から伝来し、出生から40時間以内に乳児の胃腸管にコロニーを形成する。腸内では、大腸菌は大腸粘液に付着する。大腸菌はヒト消化管における主要な通性嫌気性菌の一つである[104]。これらの細菌は、病原性因子をコードする遺伝的要素を獲得しない限り、それらは良性であり、ヒトと共生関係を維持することができる[105]。
ほとんどの大腸菌は無害な腸内細菌として腸管内に生息している[106]。ただし、いくつかの場合では疾患の原因となることがあり、病原性株は胃腸炎、尿路感染症、新生児髄膜炎、出血性大腸炎、およびクローン病を引き起こす可能性があることが知られている。一般的な兆候と症状には、重度の腹部痙攣、下痢、出血性大腸炎、嘔吐、場合によっては発熱があげられる。まれなケースでは、毒性菌株は 溶血性尿毒症症候群、腹膜炎、乳房炎、敗血症、グラム陰性肺炎などを引き起こすこと無く、腸の壊死(組織死)と穿孔の原因になることもある。非常に幼い子供は、溶血性尿毒症症候群などの重症の病気にかかりやすいが、大人を含むすべての年齢の健康な個人であっても、大腸菌感染による深刻な疾患発生の潜在的リスクがあると考えられている[107][108]。
ヒトの場合、大腸内ではなく、血液中や尿路系に侵入した場合(異所感染した場合)に病原体となる。内毒素(リポ多糖)を産生するため、大腸菌による敗血症は重篤な内毒素ショック(エンドトキシンショック)を引き起こす。敗血症の原因(明らかになる場合)として最も多いのは尿路感染症であるが、大腸菌は尿路感染症の原因菌として最も多いものである。
大腸菌の株は多数報告されており、一部では動物に害となりうる性質を持つ株も存在する。大部分の健康な成人の持っている株では下痢を起こす程度で何の症状も示さないものがほとんどであるが、幼児や病気などによって衰弱している者、あるいはある種の薬物を服用している者などでは、特殊な株が病気を引き起こすことがあり、時として死亡に至ることもある。
大腸菌の株の中でも特に強い病原性を示すものは病原性大腸菌とよばれる。食品衛生学分野では病原大腸菌ともよぶ。ただし、病原性大腸菌の中でも赤痢を起こす株については特に赤痢菌とよび、衛生管理上の問題から別種扱いされる。
O111やO157などの腸管出血性大腸菌は牛の腸内に生息しているとされ、保健所は「内臓と他部位の肉は調理器具を使い分けるのが好ましい」としている。
1996年、スコットランドのウィショーで世界で最悪の大腸菌食中毒が発生し、21人が死亡した[109]。
指標生物として
[編集]腸内に生息する菌であることから、この菌の存在は糞便による水の汚染を示唆し、河川、湖、海水浴場などの環境水の汚れの程度の指標として用いられる。
ヒト成人が一日に排泄する糞便中に含まれる菌体数は、平均で1011から1013個である。ただしヒトの消化管において、大腸菌が全体の微生物に占める割合は極めて少なく、ヒト腸内常在細菌の0.01%以下にすぎない。残りの大部分は、バクテロイデス属やユーバクテリウム属などの偏性嫌気性菌である。
水の浄化や汚水処理技術の分野では、培養可能な大腸菌の量は人間の糞便の混入の程度を示唆するものとして、水の汚染レベルの指標としてかなり早い時期から用いられてきた。研究に使われている大腸菌それ自体は無害であり、大腸菌がこれらの指標に用いられるのは、他の病原性のある菌(サルモネラなど)よりもこれらの糞便由来の大腸菌の方が遥かに多く含まれるとされるためである。また、日本の水道法により上水道の浄水からは「検出されてはならない」と規定されている。
大腸菌群
[編集]大腸菌群とは、細菌学用語ではなく衛生上の用語である。ラクトース発酵(乳糖分解し、酸とガスを発生)するグラム陰性、好気性・通性嫌気性で芽胞を形成しない桿菌の全てである。大腸菌であってもこれに該当しないものが多く存在する。
その多くは、汚水菌(クレブジエラ属菌、サイトロバクター属菌、エンテロバクター属菌)や土壌中の非常によく似た性質のバクテリア(よく知られたものとしてはAerobacter aerogenes)が大腸菌群として分類される。なお、病原性大腸菌はこの検査法での検出は非常に困難である。また、水中に含まれる大腸菌群を数値化したものを大腸菌群数といい、水質汚濁の指標に用いられる。
日本の食品衛生法
[編集]食品衛生法では、大腸菌群陰性とは加熱済み食品の加熱ができており、加熱後の二次汚染がないことと規定されている。また、食品衛生法の規格基準にある検査法で検出する菌を E. coli と記述しているが E. coli であってもこれにあてはまらない菌も多く、食品衛生上の行政用語である。これは検査法では、大腸菌群の培養温度が異なるだけの糞便性大腸菌群とほぼ同一の内容である。
歴史
[編集]
1885年、ドイツとオーストリアの小児科医テオドールエシェリヒは、健康な人の糞便からこの微生物を発見した。それは結腸で発見されたので、彼はそれをバクテリア・コリ・コミューン(Bacterium coli commune)と呼んだ。原核生物の初期の分類では、形状と運動性に基づいてこれらを少数の属に分類していた(当時、モネラ界におけるエルンストヘッケルの細菌の分類が実施されていた)[110][111][112]。
以前のタイプの種("Bacterium triloculare")が失われており、Bacterium coliは現在無効な属のタイプ種となっている[113]。Bacteriumの改訂後、1895年にMigulaによってBacillus coliとして再分類され[1]、その後新しく作成されたEscherichia属に再分類され、元の発見者にちなんで属名が名付けられた[114]。
学名
[編集]学名(ラテン語名)は Escherichia coli で、属名は発見者のオーストリア人医学者テオドール・エシェリヒにちなみ、これに屈折語尾を加えてラテン語化したもの。種形容語はラテン語で大腸を意味する「colon」の属格「coli」である。学名の正式な読みというものは存在しないが、語源を重視するとエシェリヒア・コリー、語源を無視して属名もラテン語読みするとエスケリキア・コリーとなる。英語ではエシェリキア・コーライと読む。全体として「大腸のエシェリヒ菌」の意を表す。
属名を省略してE. coli(イー・コライ、イー・コリー)と略す表記もある。ただし正式には、これはEscherichia属が既出の場合に認められる略記である。最初からE. coliと略すのは、文脈からEscherichia属のことを言っているのが明らかでも、不適切である。
大腸菌属は腸内細菌科のタイプ属として指定されているが、腸内細菌科の学名はEscherichiaceaeではなく、Enterobacteriaceaeとなっている。
検査器材
[編集]大腸菌及び大腸菌群の検査には用途に応じて多くの培地が使用される。以下に主な物を列挙する。
- エオシン・メチレンブルー寒天培地
- BGLB培地
- デソキシコレート寒天培地
- VRB寒天培地
- VRBG寒天培地
- 乳糖ブイヨン培地
脚注
[編集]出典
[編集]- ^ a b c Migula W. (1895). “Bacteriaceae (Stabchenbacterien)”. In Engler A, Prantl K. Die Naturlichen Pfanzenfamilien, Teil I, Abteilung Ia, W. Engelmann, Leipzig. pp. 20-30
- ^ Castellani A, Chalmers AJ (1919). Manual of Tropical Medicine, 3rd ed.. Williams Wood and Co., New York
- ^ V. B. D. Skerman, Vicki McGowan and P. H. A. Sneath (01 January 1980). “Approved Lists of Bacterial Names”. International Journal of Systematic and Evolutionary Microbiology 30. doi:10.1099/00207713-30-1-225.
- ^ “Escherichia coli”. CDC National Center for Emerging and Zoonotic Infectious Diseases. 2 October 2012閲覧。
- ^ “Escherichia coli O157:H7 outbreak associated with consumption of ground beef, June-July 2002”. Public Health Reports 120 (2): 174-78. (2005). doi:10.1177/003335490512000211. PMC 1497708. PMID 15842119.
- ^ “Biosynthesis of vitamin K (menaquinone) in bacteria”. Microbiological Reviews 46 (3): 241-80. (September 1982). doi:10.1128/MMBR.46.3.241-280.1982. PMC 281544. PMID 6127606.
- ^ “Escherichia coli strains colonising the gastrointestinal tract protect germfree mice against Salmonella typhimurium infection”. Gut 49 (1): 47-55. (July 2001). doi:10.1136/gut.49.1.47. PMC 1728375. PMID 11413110.
- ^ “Can bacterial interference prevent infection?”. Trends in Microbiology 9 (9): 424-28. (September 2001). doi:10.1016/S0966-842X(01)02132-1. PMID 11553454.
- ^ “Diversity of the human intestinal microbial flora”. Science 308 (5728): 1635-38. (June 2005). Bibcode: 2005Sci...308.1635E. doi:10.1126/science.1110591. PMC 1395357. PMID 15831718.
- ^ “Practical mechanisms for interrupting the oral-fecal lifecycle of Escherichia coli”. Journal of Molecular Microbiology and Biotechnology 3 (2): 265-72. (April 2001). PMID 11321582.
- ^ Feng P (1 September 2002). “Enumeration of Escherichia coli and the Coliform Bacteria”. Bacteriological Analytical Manual (8th ed.). FDA/Center for Food Safety & Applied Nutrition. 19 May 2009時点のオリジナルよりアーカイブ。25 January 2007閲覧。
- ^ Thompson, Andrea (4 June 2007). “E. coli Thrives in Beach Sands”. Live Science 3 December 2007閲覧。
- ^ “Risk Factors for Detection, Survival, and Growth of Antibiotic-Resistant and Pathogenic Escherichia coli in Household Soils in Rural Bangladesh”. Applied and Environmental Microbiology 84 (24): e01978-18. (December 2018). doi:10.1128/AEM.01978-18. PMC 6275341. PMID 30315075.
- ^ Tortora, Gerard (2010). Microbiology: An Introduction. San Francisco, CA: Benjamin Cummings. pp. 85-87, 161, 165. ISBN 978-0-321-55007-1
- ^ “Bacteria”. Microbiologyonline. 27 February 2014時点のオリジナルよりアーカイブ。27 February 2014閲覧。
- ^ “E.Coli”. Redorbit. 27 November 2013閲覧。
- ^ “Monitoring bacterial growth using tunable resistive pulse sensing with a pore-based technique”. Applied Microbiology and Biotechnology 98 (2): 855-62. (January 2014). doi:10.1007/s00253-013-5377-9. PMID 24287933.
- ^ “Cell volume increase in Escherichia coli after shifts to richer media”. Journal of Bacteriology 172 (1): 94-101. (January 1990). doi:10.1128/jb.172.1.94-101.1990. PMC 208405. PMID 2403552.
- ^ 巌佐庸・倉谷滋・斎藤成也・塚谷裕一『岩波生物学辞典 第5版』岩波書店、2013年2月26日、p.858b「大腸菌」頁。ISBN 9784000803144。
- ^ Tortora, Gerard (2010). Microbiology: An Introduction. San Francisco, CA: Benjamin Cummings. pp. 85-87, 161, 165. ISBN 978-0-321-55007-1
- ^ “On torque and tumbling in swimming Escherichia coli”. Journal of Bacteriology 189 (5): 1756-64. (March 2007). doi:10.1128/JB.01501-06. PMC 1855780. PMID 17189361.
- ^ “E. Coli O157 in North America - microbewiki”. 2020年8月2日閲覧。
- ^ Brock Biology of microorganisms (11th ed.). Pearson. (2006). ISBN 978-0-13-196893-6
- ^ “2” (English). Cell 179 (6): 1255-1263.e12. (November 2019). doi:10.1016/j.cell.2019.11.009. PMC 6904909. PMID 31778652.
- ^ “Growth of Escherichia coli at elevated temperatures”. Journal of Basic Microbiology 45 (5): 403-04. (2005). doi:10.1002/jobm.200410542. PMID 16187264.
- ^ “The respiratory chains of Escherichia coli”. Microbiological Reviews 48 (3): 222-71. (September 1984). doi:10.1128/MMBR.48.3.222-271.1984. PMC 373010. PMID 6387427.
- ^ Tortora, Gerard (2010). Microbiology: An Introduction. San Francisco, CA: Benjamin Cummings. pp. 85-87, 161, 165. ISBN 978-0-321-55007-1
- ^ “Metabolism, cell growth and the bacterial cell cycle”. Nature Reviews. Microbiology 7 (11): 822-27. (November 2009). doi:10.1038/nrmicro2202. PMC 2887316. PMID 19806155.
- ^ “Chromosome replication and the division cycle of Escherichia coli B/r”. Journal of Molecular Biology 31 (3): 519-40. (February 1968). doi:10.1016/0022-2836(68)90425-7. PMID 4866337.
- ^ a b Skarstad, K.; Boye, E.; Steen, H.b. (1986-07-01). “Timing of initiation of chromosome replication in individual Escherichia coli cells.”. The EMBO Journal 5 (7): 1711-17. doi:10.1002/j.1460-2075.1986.tb04415.x. ISSN 0261-4189. PMID 3527695.
- ^ Ishida, Takuma; Akimitsu, Nobuyoshi; Kashioka, Tamami; Hatano, Masakazu; Kubota, Toshio; Ogata, Yasuyuki; Sekimizu, Kazuhisa; Katayama, Tsutomu (2004-10-29). “DiaA, a Novel DnaA-binding Protein, Ensures the Timely Initiation of Escherichia coli Chromosome Replication” (英語). Journal of Biological Chemistry 279 (44): 45546-55. doi:10.1074/jbc.M402762200. ISSN 0021-9258. PMID 15326179.
- ^ “Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion”. Microbiology and Molecular Biology Reviews 68 (3): 560-602, table of contents. (September 2004). doi:10.1128/MMBR.68.3.560-602.2004. PMC 515249. PMID 15353570.
- ^ a b Krieg, N. R.; Holt, J. G., eds (1984). Bergey's Manual of Systematic Bacteriology. 1 (First ed.). Baltimore: The Williams & Wilkins Co. pp. 408-20. ISBN 978-0-683-04108-8
- ^ “Escherichia coli in disguise: molecular origins of Shigella”. Microbes and Infection 4 (11): 1125-32. (September 2002). doi:10.1016/S1286-4579(02)01637-4. PMID 12361912.
- ^ “Comparison of 61 sequenced Escherichia coli genomes”. Microbial Ecology 60 (4): 708-20. (November 2010). doi:10.1007/s00248-010-9717-3. PMC 2974192. PMID 20623278.
- ^ Feng P (1 September 2002). “Enumeration of Escherichia coli and the Coliform Bacteria”. Bacteriological Analytical Manual (8th ed.). FDA/Center for Food Safety & Applied Nutrition. 19 May 2009時点のオリジナルよりアーカイブ。25 January 2007閲覧。
- ^ Thompson, Andrea (4 June 2007). “E. coli Thrives in Beach Sands”. Live Science 3 December 2007閲覧。
- ^ a b c d 腸管出血性大腸菌Q&A
- ^ “Serology, chemistry, and genetics of O and K antigens of Escherichia coli”. Bacteriological Reviews 41 (3): 667-710. (September 1977). doi:10.1128/MMBR.41.3.667-710.1977. PMC 414020. PMID 334154.
- ^ “The structures of Escherichia coli O-polysaccharide antigens”. FEMS Microbiology Reviews 30 (3): 382-403. (May 2006). doi:10.1111/j.1574-6976.2006.00016.x. PMID 16594963.
- ^ “Molecular archaeology of the Escherichia coli genome”. Proceedings of the National Academy of Sciences of the United States of America 95 (16): 9413-17. (August 1998). Bibcode: 1998PNAS...95.9413L. doi:10.1073/pnas.95.16.9413. PMC 21352. PMID 9689094.
- ^ a b “Diarrheagenic Escherichia coli”. Clinical Microbiology Reviews 11 (1): 142-201. (January 1998). doi:10.1128/CMR.11.1.142. PMC 121379. PMID 9457432.
- ^ “Outbreak of diarrhoea due to Escherichia coli O111:B4 in schoolchildren and adults: association of Vi antigen-like reactivity”. Lancet 336 (8719): 831-34. (October 1990). doi:10.1016/0140-6736(90)92337-H. PMID 1976876.
- ^ “A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land”. BMC Evolutionary Biology 4: 44. (November 2004). doi:10.1186/1471-2148-4-44. PMC 533871. PMID 15535883.
- ^ “Escherichia coli molecular phylogeny using the incongruence length difference test”. Molecular Biology and Evolution 15 (12): 1685-95. (December 1998). doi:10.1093/oxfordjournals.molbev.a025895. PMID 9866203.
- ^ Bacteria make major evolutionary shift in the lab New Scientist
- ^ Nair, Ramith R.; Vasse, Marie; Wielgoss, Sébastien; Sun, Lei; Yu, Yuen-Tsu N.; Velicer, Gregory J. "Bacterial predator-prey coevolution accelerates genome evolution and selects on virulence-associated prey defences", Nature Communications, 2019, 10:4301.
- ^ Brenner, Don J.; Krieg, Noel R.; Staley, James T. (26 July 2005) [1984 (Williams & Wilkins)]. Garrity, George M.. ed. The Gammaproteobacteria. Bergey's Manual of Systematic Bacteriology. 2B (2nd ed.). New York: Springer. p. 1108. ISBN 978-0-387-24144-9. British Library no. GBA561951
- ^ Discussion of nomenclature of Enterobacteriaceae entry in LPSN; Euzéby, J.P. (1997). "List of Bacterial Names with Standing in Nomenclature: a folder available on the Internet". International Journal of Systematic and Evolutionary Microbiology (英語). 47 (2): 590–592. doi:10.1099/00207713-47-2-590. PMID 9103655。
- ^ International Bulletin of Bacteriological Nomenclature and Taxonomy 8:73-74 (1958)
- ^ a b “Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy”. Standards in Genomic Sciences 9: 2. (2013). doi:10.1186/1944-3277-9-2. PMC 4334874. PMID 25780495.
- ^ “Details: DSM-30083”. dsmz.de. 10 January 2017閲覧。
- ^ “Escherichia coli (Migula) Castellani and Chalmers ATCC ® 11775&tra”. atcc.org. 10 January 2017閲覧。
- ^ “Escherichia”. LPSN. 6 February 2011閲覧。
- ^ “Escherichia coli (Migula 1895) Castellani and Chalmers 1919”. JCM Catalogue. 2020年8月13日閲覧。
- ^ “Whole-genome phylogeny of Escherichia coli/Shigella group by feature frequency profiles (FFPs)”. Proceedings of the National Academy of Sciences of the United States of America 108 (20): 8329-34. (May 2011). Bibcode: 2011PNAS..108.8329S. doi:10.1073/pnas.1105168108. PMC 3100984. PMID 21536867.
- ^ “Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC)”. Archives of Microbiology 193 (12): 883-91. (December 2011). doi:10.1007/s00203-011-0725-6. PMC 3219860. PMID 21713444.
- ^ “Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy”. Standards in Genomic Sciences 9: 2. (2013). doi:10.1186/1944-3277-9-2. PMC 4334874. PMID 25780495.
- ^ a b “Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy”. Standards in Genomic Sciences 9: 2. (2013). doi:10.1186/1944-3277-9-2. PMC 4334874. PMID 25780495.
- ^ a b c d “The complete genome sequence of Escherichia coli K-12”. Science 277 (5331): 1453-62. (September 1997). doi:10.1126/science.277.5331.1453. PMID 9278503.
- ^ “Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy”. Standards in Genomic Sciences 9: 2. (2013). doi:10.1186/1944-3277-9-2. PMC 4334874. PMID 25780495.
- ^ “Comparison of 61 sequenced Escherichia coli genomes”. Microbial Ecology 60 (4): 708-20. (November 2010). doi:10.1007/s00248-010-9717-3. PMC 2974192. PMID 20623278.
- ^ “Lateral gene transfer”. Current Biology 21 (7): R242-46. (April 2011). doi:10.1016/j.cub.2011.01.045. PMID 21481756.
- ^ “Highly accurate genome sequences of Escherichia coli K-12 strains MG1655 and W3110”. Molecular Systems Biology 2: 2006.0007. (2006). doi:10.1038/msb4100049. PMC 1681481. PMID 16738553.
- ^ a b “EcoGene 3.0”. Nucleic Acids Research 41 (Database issue): D613-24. (January 2013). doi:10.1093/nar/gks1235. PMC 3531124. PMID 23197660.
- ^ “The Escherichia coli proteome: past, present, and future prospects”. Microbiology and Molecular Biology Reviews 70 (2): 362-439. (June 2006). doi:10.1128/MMBR.00036-05. PMC 1489533. PMID 16760308.
- ^ “Large-scale identification of protein-protein interaction of Escherichia coli K-12”. Genome Research 16 (5): 686-91. (May 2006). doi:10.1101/gr.4527806. PMC 1457052. PMID 16606699.
- ^ “Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins”. PLOS Biology 7 (4): e96. (April 2009). doi:10.1371/journal.pbio.1000096. PMC 2672614. PMID 19402753.
- ^ “The binary protein-protein interaction landscape of Escherichia coli”. Nature Biotechnology 32 (3): 285-90. (March 2014). doi:10.1038/nbt.2831. PMC 4123855. PMID 24561554.
- ^ “High cell-density culture of Escherichia coli”. Trends in Biotechnology 14 (3): 98-105. (March 1996). doi:10.1016/0167-7799(96)80930-9. PMID 8867291.
- ^ “The birth of biotechnology”. Nature 421 (6921): 456-57. (January 2003). Bibcode: 2003Natur.421..456R. doi:10.1038/nj6921-456a. PMID 12540923.
- ^ “Expressing genes in different Escherichia coli compartments”. Current Opinion in Biotechnology 11 (5): 450-54. (October 2000). doi:10.1016/S0958-1669(00)00131-2. PMID 11024362.
- ^ Tof (1994年). “Recombinant DNA Technology in the Synthesis of Human Insulin”. Little Tree Pty. Ltd.. 30 November 2007閲覧。
- ^ “Efficient folding of proteins with multiple disulfide bonds in the Escherichia coli cytoplasm”. Proceedings of the National Academy of Sciences of the United States of America 96 (24): 13703-08. (November 1999). Bibcode: 1999PNAS...9613703B. doi:10.1073/pnas.96.24.13703. PMC 24128. PMID 10570136.
- ^ “Production of glycoprotein vaccines in Escherichia coli”. Microbial Cell Factories 9 (61): 61. (August 2010). doi:10.1186/1475-2859-9-61. PMC 2927510. PMID 20701771.
- ^ “N-linked glycosylation in Campylobacter jejuni and its functional transfer into E. coli”. Science 298 (5599): 1790-93. (November 2002). Bibcode: 2002Sci...298.1790W. doi:10.1126/science.298.5599.1790. PMID 12459590.
- ^ “Industrial production of recombinant therapeutics in Escherichia coli and its recent advancements”. Journal of Industrial Microbiology & Biotechnology 39 (3): 383-99. (March 2012). doi:10.1007/s10295-011-1082-9. PMID 22252444.
- ^ Summers, Rebecca (24 April 2013) Bacteria churn out first ever petrol-like biofuel New Scientist, Retrieved 27 April 2013
- ^ “Expressing genes in different Escherichia coli compartments”. Current Opinion in Biotechnology 11 (5): 450-54. (October 2000). doi:10.1016/S0958-1669(00)00131-2. PMID 11024362.
- ^ Nic Halverson (15 August 2013). “Bacteria-Powered Light Bulb Is Electricity-Free”
- ^ a b Ninfa, Alexander J.; Ballou, David P. (2009). Fundamental Laboratory Approaches for Biochemistry and Biotechnology. Wiley. pp. 230. ISBN 978-0470087664
- ^ ."Cloning, sequence analysis, and expression of cDNA coding for the major house dust mite allergen, Der f 1, in Escherichia coli" Brazilian Journal of Medical and Biological Research
- ^ “Can laboratory reference strains mirror "real-world" pathogenesis?”. Trends in Microbiology 13 (2): 58-63. (February 2005). doi:10.1016/j.tim.2004.11.001. PMID 15680764.
- ^ “Isolation of an Escherichia coli K-12 mutant strain able to form biofilms on inert surfaces: involvement of a new ompR allele that increases curli expression”. Journal of Bacteriology 180 (9): 2442-49. (May 1998). doi:10.1128/JB.180.9.2442-2449.1998. PMC 107187. PMID 9573197.
- ^ TiO2 coatings as bactericidal photocatalysts in water treatment.
- ^ “Gene recombination in Escherichia coli”. Nature 158 (4016): 558. (October 1946). Bibcode: 1946Natur.158..558L. doi:10.1038/158558a0. PMID 21001945. Source: National Library of Medicine - The Joshua Lederberg Papers
- ^ Biological Activity of Crystal. pp. 169
- ^ “The Cold Spring Harbor Phage Course (1945-1970): a 50th anniversary remembrance”. Genetics 139 (3): 1101-06. (March 1995). PMC 1206443. PMID 7768426.
- ^ “On the Topography of the Genetic Fine Structure”. Proceedings of the National Academy of Sciences of the United States of America 47 (3): 403-15. (March 1961). Bibcode: 1961PNAS...47..403B. doi:10.1073/pnas.47.3.403. PMC 221592. PMID 16590840.
- ^ “Facts about E.Coli”. Encyclopedia of Life. 27 November 2013閲覧。
- ^ “Scarab Genomics LLC. Company web site.”. 2021年10月6日閲覧。
- ^ “Reduced evolvability of Escherichia coli MDS42, an IS-less cellular chassis for molecular and synthetic biology applications”. Microbial Cell Factories 9: 38. (May 2010). doi:10.1186/1475-2859-9-38. PMC 2891674. PMID 20492662.
- ^ “Emergent properties of reduced-genome Escherichia coli”. Science 312 (5776): 1044-46. (May 2006). Bibcode: 2006Sci...312.1044P. doi:10.1126/science.1126439. PMID 16645050.
- ^ “Engineering a reduced Escherichia coli genome”. Genome Research 12 (4): 640-47. (April 2002). doi:10.1101/gr.217202. PMC 187512. PMID 11932248.
- ^ “Bacterial metapopulations in nanofabricated landscapes”. Proceedings of the National Academy of Sciences of the United States of America 103 (46): 17290-95. (November 2006). Bibcode: 2006PNAS..10317290K. doi:10.1073/pnas.0607971103. PMC 1635019. PMID 17090676.
- ^ ポステリザン
- ^ Anselm Levskaya、Aaron A. Chevalier「Synthetic biology: Engineering Escherichia coli to see light」『ネイチャー』、Nature Publishing Group、2005年11月24日、441-442頁。
- ^ “Solving a Hamiltonian Path Problem with a bacterial computer”. Journal of Biological Engineering 3: 11. (July 2009). doi:10.1186/1754-1611-3-11. PMC 2723075. PMID 19630940.
- ^ “E. Coli”. European Journal of Microbiology & Immunology 6 (3): 147-61. (September 2016). doi:10.1556/1886.2016.00029. PMC 5063008. PMID 27766164.
- ^ “Oral administration of probiotic Escherichia coli after birth reduces frequency of allergies and repeated infections later in life (after 10 and 20 years)”. International Archives of Allergy and Immunology 131 (3): 209-11. (July 2003). doi:10.1159/000071488. PMID 12876412.
- ^ “Analysis of the genome structure of the nonpathogenic probiotic Escherichia coli strain Nissle 1917”. Journal of Bacteriology 186 (16): 5432-41. (August 2004). doi:10.1128/JB.186.16.5432-5441.2004. PMC 490877. PMID 15292145.
- ^ “Nonpathogenic Escherichia coli strain Nissle1917 prevents murine acute and chronic colitis”. Inflammatory Bowel Diseases 11 (5): 455-63. (May 2005). doi:10.1097/01.MIB.0000158158.55955.de. PMID 15867585.
- ^ Brenner, Don J.; Krieg, Noel R.; Staley, James T. (26 July 2005) [1984 (Williams & Wilkins)]. Garrity, George M.. ed. The Gammaproteobacteria. Bergey's Manual of Systematic Bacteriology. 2B (2nd ed.). New York: Springer. p. 1108. ISBN 978-0-387-24144-9. British Library no. GBA561951
- ^ “Pathogenic E. coli”. Online Textbook of Bacteriology. University of Wisconsin-Madison Department of Bacteriology. 30 November 2007閲覧。
- ^ Evans Jr.. “Escherichia Coli”. Medical Microbiology, 4th edition. The University of Texas Medical Branch at Galveston. 2 November 2007時点のオリジナルよりアーカイブ。2 December 2007閲覧。
- ^ “E. coli”. mayoclinic.org - Mayo Clinic. 10 January 2017閲覧。
- ^ “Pathogenic E. coli”. Online Textbook of Bacteriology. University of Wisconsin-Madison Department of Bacteriology. 30 November 2007閲覧。
- ^ “A brief overview of Escherichia coli O157:H7 and its plasmid O157”. Journal of Microbiology and Biotechnology 20 (1): 5-14. (January 2010). doi:10.4014/jmb.0908.08007. PMC 3645889. PMID 20134227.
- ^ “BBC News | Health | Sheriff criticises E. Coli butcher”. 2021年10月6日閲覧。
- ^ Farrar, Jeremy; Hotez, Peter; Junghanss, Thomas et al., eds (2013). Manson's Tropical Diseases (23rd ed.). Oxford: Elsevier/Saunders. ISBN 978-0702053061
- ^ Haeckel, Ernst (1867). Generelle Morphologie der Organismen. Reimer, Berlin. ISBN 978-1-144-00186-3
- ^ “Die Darmbakterien des Neugeborenen und Säuglinge”. Fortschr. Med. 3: 515-22. (1885).
- ^ “The Status of the Generic Term Bacterium Ehrenberg 1828”. Journal of Bacteriology 31 (5): 517-18. (May 1936). doi:10.1128/JB.31.5.517-518.1936. PMC 543738. PMID 16559906.
- ^ Manual of Tropical Medicine (3rd ed.). New York: Williams Wood and Co.. (1919)


