RecBCD
| Exodeoxyribonuclease V | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 識別子 | |||||||||
| EC番号 | 3.1.11.5 | ||||||||
| CAS登録番号 | 37350-26-8 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
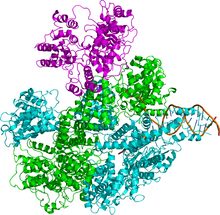
RecBCDまたはエキソヌクレアーゼV(exonuclease V、EC 3.1.11.5)は、大腸菌Escherichia coliでDNAの二本鎖切断の組換え修復を開始する酵素である。二本鎖切断は、電離放射線、DNA複製のエラー、エンドヌクレアーゼ、酸化損傷やその他の多くの因子によって生じる可能性があり、その影響は致死的となりうる[2][3]。RecBCDは、DNA鎖を巻き戻したり引き離したりするヘリカーゼであり、DNAに一本鎖のニックを形成するヌクレアーゼでもある[1]。
構造
[編集]この酵素複合体は3つの異なるサブユニットRecB、RecC、RecDから構成され、そのため複合体はRecBCDと呼ばれている。recD遺伝子の発見[4]以前は、酵素は"RecBC"として知られていた。各サブユニットは異なる遺伝子によってコードされている。
| 遺伝子 | 鎖 | タンパク質 | 機能 |
|---|---|---|---|
| recB | beta | RecB(P08394) | 3'-5'ヘリカーゼ、ヌクレアーゼ |
| recC | gamma | RecC(P07648) | Chi(crossover hotspot instigator)の認識 |
| recD | alpha | RecD(P04993) | 5'-3'ヘリカーゼ |
機能
[編集]
RecDとRecBはどちらもヘリカーゼであり、エネルギー依存的にDNAを巻き戻す分子モーターである。それに加えて、RecBサブユニットにはヌクレアーゼとしての機能も存在する[5]。RecBCD酵素(おそらくRecCサブユニット)がDNA中の特定の配列(5'-GCTGGTGG-3')を認識する。この配列はChi部位(ギリシア文字のχで表されることもある)として知られている。
RecBCDは異なる速度で移動する2つのヘリカーゼを持っている点[6]、そしてChi部位の認識に伴って活性に変化が生じる点で、珍しいヘリカーゼである[7][8]。RecBCDは二本鎖DNAの直鎖状の末端に結合する。RecDヘリカーゼは5'末端側の鎖を移動し、RecBは3'末端側の鎖を移動する。RecBはRecDよりも遅いため、RecBの前方には一本鎖のDNAループ構造が形成される。この過程によって、短い3'末端と長い5'末端の2つの一本鎖テールと1つの一本鎖ループからなるDNA構造が形成され、この構造は電子顕微鏡によっても観察される[9]。一本鎖テールどうしは再びアニーリングし、1つ目の一本鎖ループに相補的な2つ目の一本鎖ループが形成される。こうした2つのループ構造は「ウサギの耳」("rabbit ears")と呼ばれていた。
作用機構
[編集]
巻き戻しの過程で、RecBのヌクレアーゼ活性は反応条件、特ににMg2+イオンとATPの濃度に依存して異なる作用を示す。(1) ATPが過剰に存在する場合、酵素は単にChi部位が存在する鎖(3'末端の鎖)にニックを入れる[10][11]。巻き戻しは継続し、Chi部位が末端付近に存在する3'一本鎖テールが形成される。このテールにRecAタンパク質が結合し、損傷のない相同なDNA二本鎖との鎖交換を促進する[12]。RecBCDがDNAの末端に到達すると3つのサブユニットは解体し、酵素は1時間以上不活性状態となり[13]、Chi部位で作用したRecBCD分子が他のDNA分子を攻撃することはない。(2) Mg2+イオンが過剰に存在する場合、RecBCDは双方のDNA鎖をエンドヌクレアーゼのように切断するが、5'テールの切断頻度は低い[14]。RecBCDが3'末端側の鎖のChi部位に遭遇すると、巻き戻しは一時停止し、3'テールの分解も低下する[15]。RecBCDが巻き戻しを再開すると、今度は反対側の5'テールを切断するようになり[16][17]、3'末端側の鎖にはRecAタンパク質がロードされる[12]。あるDNA分子に対する反応が完結すると、酵素はすぐに次のDNAを攻撃し、同じ反応が行われる。
反応の中間体の一過的性質のため、どちらの反応も細胞内のDNAの分析による検証は行われていない。しかし、遺伝学的な証拠は前者が細胞内での反応をよりよく模倣したものであることを示している[2]。例えば、細胞内での反応もATP過剰存在下での反応も、Chi部位の活性は3'側のヌクレオチドの影響を受けるが、Mg2+過剰存在下ではそうした影響は見られない[18][19]。検出可能なエキソヌクレアーゼ活性を持たないRecBCDの変異体は、細胞内での高いChiホットスポット活性と細胞外でのChi部位でのニック形成活性を保持している[20]。細胞内のあるDNA分子のChi部位は他の分子のChi部位の活性を低下または消失させ、これはおそらくATP過剰存在下で観察されるChi部位依存的なRecBCDの解体を反映している[21][22]。
どちらの反応条件下でも、Chi部位の下流の3'鎖は無傷のまま維持される。その後、RecAタンパク質はRecBCDによって活発に3'テールにロードされる[12]。RecBCDがDNAから解離する地点は未決定であるが、RecBCDはDNAから脱落することなく少なくとも60 kbを巻き戻すことができる。RecAは自身が結合しているDNA鎖と、損傷のない相同なDNA二本鎖との間での鎖交換を開始する。この鎖交換によって、Dループなどの連結されたDNA分子が形成される。連結されたDNA構造は、侵入してきた3'末端鎖によってプライミングされたDNA複製、またはDループの切断とホリデイジャンクションの形成によって解消される。ホリデイジャンクションはRuvABC複合体による直鎖状DNAへの解消、またはRecGタンパク質による解離が行われる。こうしたイベントによって、親のDNAとは異なる、遺伝子の新たな組み合わせを持つDNAが作り出される可能性がある。この過程(相同組換え)によってDNAの二本鎖切断修復は完結する。
応用
[編集]RecBCDは、タンパク質-DNA間相互作用の機能をより良く理解するために用いられる実験技術である、1分子FRETを利用する際のモデル酵素となっている[23]。また、この酵素の活性にはDNAの末端が必要であるため、環状二本鎖DNAの調製の際に、一本鎖または二本鎖の直鎖状DNAを除去するためにも有用である。
出典
[編集]- ^ a b “Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks”. Nature 432 (7014): 187–93. (November 2004). Bibcode: 2004Natur.432..187S. doi:10.1038/nature02988. PMID 15538360.
- ^ a b Smith, GR (June 2012). “How RecBCD Enzyme and Chi Promote DNA Break Repair and Recombination: a Molecular Biologist's View”. Microbiol Mol Biol Rev 76 (2): 217–28. doi:10.1128/MMBR.05026-11. PMC 3372252. PMID 22688812.
- ^ Higgins P, ed (2003). “Homologous recombination by RecBCD and RecF pathways”. Bacterial Chromosomes. Washington, D.C: ASM Press. pp. 389–403. ISBN 1-55581-232-5
- ^ “recD: the gene for an essential third subunit of exonuclease V.”. Proc Natl Acad Sci U S A 83 (15): 5558–62. (August 1986). Bibcode: 1986PNAS...83.5558A. doi:10.1073/pnas.83.15.5558. PMC 386327. PMID 3526335.
- ^ “The 30-kDa C-terminal domain of the RecB protein is critical for the nuclease activity, but not the helicase activity, of the RecBCD enzyme from Escherichia coli”. Proc Natl Acad Sci U S A 95 (3): 981–6. (February 1998). doi:10.1073/pnas.95.3.981. PMC 18645. PMID 9448271.
- ^ “RecBCD enzyme is a DNA helicase with fast and slow motors of opposite polarity”. Nature 423 (6942): 889–93. (June 2003). Bibcode: 2003Natur.423..889T. doi:10.1038/nature01674. PMID 12815437.
- ^ “RecBCD enzyme is altered upon cutting DNA at a Chi recombination hotspot”. Proc Natl Acad Sci U S A 89 (12): 5226–30. (June 1992). Bibcode: 1992PNAS...89.5226T. doi:10.1073/pnas.89.12.5226. PMC 49264. PMID 1535156.
- ^ “Intersubunit signaling in RecBCD enzyme, a complex protein machine regulated by Chi hot spots”. Genes Dev 21 (24): 3296–307. (December 2007). doi:10.1101/gad.1605807. PMC 2113030. PMID 18079176.
- ^ “Unwinding and rewinding of DNA by the RecBC enzyme”. Cell 22 (2 Pt 2): 447–57. (November 1980). doi:10.1016/0092-8674(80)90355-4. PMID 6256081.
- ^ Ponticelli, A. S.; Schultz, D. W.; Taylor, A. F.; Smith, G. R. (May 1985). “Chi-dependent DNA strand cleavage by RecBC enzyme”. Cell 41 (1): 145–151. doi:10.1016/0092-8674(85)90069-8. PMID 3888404.
- ^ “RecBC enzyme nicking at Chi sites during DNA unwinding: location and orientation-dependence of the cutting”. Cell 41 (1): 153–63. (May 1985). doi:10.1016/0092-8674(85)90070-4. PMID 3888405.
- ^ a b c “The translocating RecBCD enzyme stimulates recombination by directing RecA protein onto ssDNA in a Chi-regulated manner”. Cell 90 (1): 77–86. (July 1997). doi:10.1016/S0092-8674(00)80315-3. PMID 9230304.
- ^ “Regulation of homologous recombination: Chi inactivates RecBCD enzyme by disassembly of the three subunits”. Genes Dev. 13 (7): 890–900. (April 1999). doi:10.1101/gad.13.7.890. PMC 316601. PMID 10197988.
- ^ “The recombination hotspot Chi is a regulatory sequence that acts by attenuating the nuclease activity of the E. coli RecBCD enzyme”. Cell 73 (1): 87–96. (April 1993). doi:10.1016/0092-8674(93)90162-J. PMID 8384931.
- ^ “RecBCD enzyme switches lead motor subunits in response to Chi recognition”. Cell 131 (4): 694–705. (November 2007). doi:10.1016/j.cell.2007.09.023. PMC 2151923. PMID 18022364.
- ^ “Strand specificity of nicking of DNA at Chi sites by RecBCD enzyme. Modulation by ATP and magnesium levels”. J Biol Chem 270 (41): 24459–67. (October 1995). doi:10.1074/jbc.270.41.24459. PMID 7592661.
- ^ “The recombination hot spot chi is a regulatory element that switches the polarity of DNA degradation by the RecBCD enzyme”. Genes Dev. 11 (5): 571–81. (March 1997). doi:10.1101/gad.11.5.571. PMID 9119222.
- ^ Amundsen, Susan K.; Sharp, Jake W.; Smith, Gerald R. (09 2016). “RecBCD Enzyme "Chi Recognition" Mutants Recognize Chi Recombination Hotspots in the Right DNA Context”. Genetics 204 (1): 139–152. doi:10.1534/genetics.116.191056. ISSN 1943-2631. PMC 5012381. PMID 27401752.
- ^ Taylor, Andrew F.; Amundsen, Susan K.; Smith, Gerald R. (09 30, 2016). “Unexpected DNA context-dependence identifies a new determinant of Chi recombination hotspots”. Nucleic Acids Research 44 (17): 8216–8228. doi:10.1093/nar/gkw541. ISSN 1362-4962. PMC 5041463. PMID 27330137.
- ^ “Chi hotspot activity in Escherichia coli without RecBCD exonuclease activity: implications for the mechanism of recombination”. Genetics 175 (1): 41–54. (January 2007). doi:10.1534/genetics.106.065524. PMC 1774988. PMID 17110484.
- ^ “Interaction with the recombination hot spot Chi in vivo converts the RecBCD enzyme of Escherichia coli into a Chi-independent recombinase by inactivation of the RecD subunit”. Proc Natl Acad Sci U S A 92 (14): 6249–53. (July 1995). Bibcode: 1995PNAS...92.6249K. doi:10.1073/pnas.92.14.6249. PMC 41495. PMID 7541534.
- ^ “The recombination hot spot Chi activates RecBCD recombination by converting Escherichia coli to a recD mutant phenocopy”. Proc Natl Acad Sci U S A 92 (14): 6244–8. (July 1995). Bibcode: 1995PNAS...92.6244M. doi:10.1073/pnas.92.14.6244. PMC 41494. PMID 7603978.
- ^ “Processive translocation and DNA unwinding by individual RecBCD enzyme molecules”. Nature 409 (6818): 374–8. (January 2001). Bibcode: 2001Natur.409..374B. doi:10.1038/35053131. PMID 11201750.
