コハク酸デヒドロゲナーゼ
| コハク酸デヒドロゲナーゼ | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 サブユニット: SdhA, SdhB, SdhC, SdhD | |||||||||
| 識別子 | |||||||||
| EC番号 | 1.3.5.1 | ||||||||
| CAS登録番号 | 9028-11-9 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| 遺伝子オントロジー | AmiGO / QuickGO | ||||||||
| |||||||||
コハク酸デヒドロゲナーゼ (succinate dehydrogenase, SDH)は、コハク酸をフマル酸へ酸化する酸化還元酵素である。コハク酸脱水素酵素とも。このとき同時にユビキノンなどのキノンを還元することから、コハク酸キノンレダクターゼ(succinate-quinone reductase, SQR)とも呼ばれる。クエン酸回路の8段階目の反応を担い、また呼吸鎖においては複合体II(Complex II)と呼ばれている。真核生物ではミトコンドリア内膜に、原核生物では細胞膜に固定されている酵素複合体である。[1]。
反応
[編集]触媒する化学反応は次の通りである。
この反応は可逆であるが好気的条件では通常右向きに進む。嫌気的条件では逆反応のフマル酸レダクターゼとして働くこともできるが、普通は逆反応を担う専門の複合体が存在する(大腸菌など)か、豚回虫のように一部のサブユニットを入れ替えることが知られている[2]。
構造
[編集]サブユニット構成
[編集]
一般的に4つのサブユニットから構成されており、親水性の2つがSdhAとSdhB、疎水性の2つがSdhCとSdhDである[3]。
- SdhA
- フラボタンパク質(Fp)サブユニットとも呼ばれる。補因子としてフラビンアデニンジヌクレオチド(FAD)が共有結合しており、その近傍にコハク酸結合部位(後述)が存在している。
- SdhB
- 鉄硫黄タンパク質(Ip)サブユニットとも呼ばれる。[2Fe-2S], [4Fe-4S], および[3Fe-4S]の3種の鉄・硫黄クラスターが含まれている。
- SdhC・SdhD
- 疎水性サブユニット2つで6個の膜貫通ヘリックスとヘムbを含むシトクロムbを構成する。リン脂質であるカルジオリピンとホスファチジルエタノールアミンが結合している。
ヒトの場合、Fpサブユニットに2種類のアイソタイプ(FpI, FpII)が存在している。豚回虫およびシノラブディス・エレガンス(線虫の一種)でもFpサブユニットのアイソタイプが見付かっている[2]。
基質結合部位
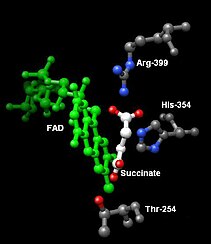
[編集]コハク酸の結合部位はサブユニットAのThr254, His354, およびArg399の側鎖で構成され、そこでFADによる酸化と最初の鉄硫黄クラスター[2Fe-2S]への電子伝達が起きる[4]。
ユビキノンの結合部位はSdhB, SdhC, およびSdhDで構成される間隙に位置している。ユビキノンはサブユニットBのHis207、サブユニットCのSer27とArg31、そしてサブユニットDのTyr83のそれぞれの側鎖で安定化されている。キノン環はサブユニットCのIle28とサブユニットBのPro160に取り囲まれている。これらの残基はサブユニットBのIle209, Trp163およびTrp164と、サブユニットCのSer27(炭素原子)と共にキノン結合ポケットの疎水的環境を形成している[5]。
|

|
酸化還元中心
[編集]コハク酸結合部位とユビキノン結合部位の間には、FADと鉄硫黄クラスターから成る酸化還元中心が連なっている。これは複合体をほぼ縦断し総距離は40 Åに達するが、それぞれの酸化還元中心間の距離は、生理的電子移動の限界として提案されている14 Åよりも短い[3]。
反応機構
[編集]コハク酸酸化:正確なコハク酸の酸化機構はほとんど分かっていない。しかし、結晶構造からサブユニットAのFAD, Glu255, Arg286, およびHis242が最初の脱プロトン過程の候補として挙がっている。したがって、E2もしくはE1cbの2種の可能な脱離機構が考えられる。E2脱離では、塩基性残基または補因子によるα炭素からの脱プロトンが起こり、FADがβ炭素からのヒドリドの受容体として作用することによりコハク酸がフマル酸に酸化される(image 6)。E1cbではFADがヒドリドを受ける前にエノラート中間体が形成する(image 7)。


電子トンネル効果:電子はFADを経由してコハク酸から派生したのち、トンネル効果によって[Fe-S]から[3Fe-4S]クラスターに中継される。この電子はその後、活性部位のユビキノン分子まで移動する。鉄硫黄電子トンネル系は図[1]参照。
ユビキノンの還元:ユビキノンのO1カルボニル酸素は、サブユニットDのTyr83との水素結合相互作用によって活性部位において正しい位置に置かれる。さらに[3Fe-4S]鉄硫黄クラスター中の電子の存在により、ユビキノンは2番目の位置に動く。これはユビキノンのO4カルボニル酸素とサブユニットCのSer27との間の2番目の水素結合相互作用により容易となる。まず、一個の電子を受けセミキノンラジカルが形成され、二個目の電子を[3Fe-4S]クラスターから受けることによりユビキノールに完全に還元される(image 8)。

ヘムの機能:コハク酸デヒドロゲナーゼにおけるヘムの機能はまだ研究段階である。いくつかの研究では、最初に[3Fe-4S]を用いて電子をユビキノンへ伝える逆のトンネル効果が主張されている。この経路ではヘムは電子を受容する補因子として作用する。これは反応中間体として酸素分子からできる活性酸素(ROS)との相互作用を防ぐ効果がある。ヘムはimage 4のようにユビキノンと関連している。
また、電子が[3Fe-4S]クラスターからヘムへ直接トンネリングするのを防ぐ開閉機構も提唱されている。電位の候補はHis207残基で、ヘムとクラスターの間に位置している。サブユニットBのHis207は[3Fe-4S]クラスターに近く、ユビキノンおよびヘムに結合しており、酸化還元中心として電子の流れを調節することが可能である[7]。
プロトン移動:SQRでキノンを完全に還元するには2個の電子と2個のプロトンが必要である。水分子(HOH39)が活性部位に付き、それがサブユニットBのHis207、サブユニットCのArg31、そしてサブユニットDのAsp82に配位されると主張されている。セミキノンはHOH39から誘導されたプロトンによりプロトン化され、ユビキノールへの還元が完了する。おそらく、His207とAsp82がこの機構を容易にしていると考えられる。他の研究では、サブユニットDのTyr83が隣のヒスチジンとユビキノンのO1カルボニル酸素に配位していると提唱されている。これは、ヒスチジン残基はチロシンのpKaを減少させ、そのプロトンをユビキノン中間体に提供するというものである。
分類
[編集]複合体IIは膜結合サブユニットに注目して以下の5種類に分類されている[8]。
| タイプ | サブユニット | 結合様式 | ヘムb | 分布 |
|---|---|---|---|---|
| A | C+D | 膜貫通 | 2 | ほとんどの古細菌 |
| B | C | 膜貫通 | 2 | ほとんどの真正細菌 |
| C | C+D | 膜貫通 | 1 | 真核生物、プロテオバクテリア(α・β・γ) |
| D | C+D | 膜貫通 | 0 | γプロテオバクテリアのフマル酸レダクターゼ |
| E | E+F | 膜表在 | 0 | スルフォロブス目古細菌 |
この他に、種子植物の複合体IIは7~8サブユニットで[9]、 トリパノソーマでは12サブユニットで構成されている[10]。
脚注
[編集]- ^ Oyedotun KS, Lemire BD (March 2004). “The quaternary structure of the Saccharomyces cerevisiae succinate dehydrogenase. Homology modeling, cofactor docking, and molecular dynamics simulation studies”. J. Biol. Chem. 279 (10): 9424–31. doi:10.1074/jbc.M311876200. PMID 14672929.
- ^ a b Tomitsuka E, Hirawake H, Goto Y, Taiwaki M, Harada S, Kita K (2003). “Direct evidence for two distinct forms of the flavoprotein subunit of human mitochondrial complex II (succinate-ubiquinone reductase)”. J. Biochem 134 (2): 191–5. doi:10.1093/jb/mvg144.
- ^ a b Yankovskaya V, Horsefield R, Törnroth S, et al. (January 2003). “Architecture of succinate dehydrogenase and reactive oxygen species generation”. Science 299 (5607): 700–4. doi:10.1126/science.1079605. PMID 12560550.
- ^ Kenney WC (April 1975). “The reaction of N-ethylmaleimide at the active site of succinate dehydrogenase”. J. Biol. Chem. 250 (8): 3089–94. PMID 235539.
- ^ Horsefield R, Yankovskaya V, Sexton G, et al. (March 2006). “Structural and computational analysis of the quinone-binding site of complex II (succinate-ubiquinone oxidoreductase): a mechanism of electron transfer and proton conduction during ubiquinone reduction”. J. Biol. Chem. 281 (11): 7309–16. doi:10.1074/jbc.M508173200. PMID 16407191.
- ^ Pettersen EF, Goddard TD, Huang CC, et al. (October 2004). “UCSF Chimera--a visualization system for exploratory research and analysis”. J Comput Chem 25 (13): 1605–12. doi:10.1002/jcc.2008410.1002/jcc.20084. PMID 15264254.
- ^ Tran QM, Rothery RA, Maklashina E, Cecchini G, Weiner JH (October 2006). “The quinone binding site in Escherichia coli succinate dehydrogenase is required for electron transfer to the heme b”. J. Biol. Chem. 281 (43): 32310–7. doi:10.1074/jbc.M607476200. PMID 16950775.
- ^ Lemos et al. (2002). “Quinol:fumarate oxidoreductases and succinate:quinone oxidoreductases: phylogenetic relationships, metal centres and membrane attachment”. Biochim. Biophys. Acta 1553 (1–2): 158–170. doi:10.1016/S0005-2728(01)00239-0.
- ^ Huang et al. (2010). “Functional and composition differences between mitochondrial complex II in Arabidopsis and rice are correlated with the complex genetic history of the enzyme”. Plant Mol. Biol. 72 (3): 331–342. doi:10.1007/s11103-009-9573-z.
- ^ Morales et al. (2009). “Novel mitochondrial complex II isolated from Trypanosoma cruzi is composed of 12 peptides including a heterodimeric Ip subunit”. J. Biol. Chem. 284 (11): 7255–7263. doi:10.1074/jbc.M806623200.
参考文献
[編集]- Molecular graphics images were produced using the UCSF Chimera package from the Resource for Biocomputing, Visualization, and Informatics at the University of California, San Francisco (supported by NIH P41 RR-01081).



