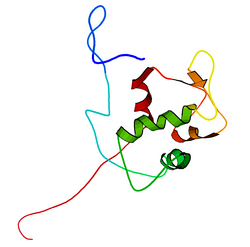
Mdm2
Mdm2はがん抑制因子であるp53の活動を抑制的に調節するタンパク質で、ヒトではMDM2遺伝子にコードされる[5][6]。Mdm2タンパク質は、p53のN末端のトランスアクティベーションドメイン(TAD)を認識するE3ユビキチンリガーゼとして、またp53の転写活性化の阻害因子として機能する。
発見とその役割
[編集]Mdm2タンパク質をコードするマウス二重微小染色体(murine double minute)がん遺伝子(mdm2)は、形質転換したマウス細胞株3T3-DMから他の2つの遺伝子(mdm1とmdm3)とともにクローニングされた。Mdm2の過剰発現は発がん性のあるRasと協同的に齧歯類初代線維芽細胞の形質転換を促進し、mdm2の発現はヌードマウスで腫瘍形成をもたらす。このタンパク質のヒトホモログが後に同定され、それはHdm2と呼ばれることもある。mdm2のがん遺伝子としての役割を支持するものとして、軟部肉腫や骨肉腫、乳腺腫瘍などヒトの腫瘍のタイプのいくつかではMDM2のレベルが上昇していることが示されている。MDM2がんタンパク質はp53に対しユビキチン化による拮抗を行うするが、p53非依存的な機能も持っている可能性がある。MDM2はPolycombを介した細胞系譜特異的な遺伝子抑制を補助し、この過程ははp53非依存的である。p53非存在下でのMDM2の欠失は、ヒトの間葉系幹細胞の分化を促進し、がん細胞のコロニー形成能を喪失させる。MDM2によって制御される遺伝子の大部分は、PRC2(polycomb repressor complex 2)やその触媒コンポーネントEZH2の不活性化にも応答する。MDM2はクロマチン上でEZH2と物理的に結合し、標的遺伝子のヒストン3のリジン27番残基(H3K27)のトリメチル化とヒストン2Aのリジン119番残基(H2AK119)のユビキチン化を向上させる。H2AK119に対するE3リガーゼRing1B/RNF2とMDM2を同時に除去すると遺伝子発現の誘導はさらに強化され、合成的に細胞増殖を停止させる[7]。
Mdm2ファミリーの別のメンバーMdm4(MdmXとも呼ばれる)が発見されており、これもまたp53の重要な負の調節因子である。
またMDM2は器官発生や組織の恒常性にも必要とされるが、それはp53の活性化が抑制されなければpodoptosisと呼ばれるp53の過剰活性化による細胞死が引き起こされるためである。podoptosisはカスパーゼ非依存的であり、そのためアポトーシスとは異なる過程である。MDM2の細胞分裂促進機能は組織傷害後の創傷治癒にも必要であり、MDM2の阻害によって上皮の損傷後の再生が損なわれる。加えて、核内でのNF-κBの活性化においてMDM2はp53非依存的な転写因子様の働きをする。そのため、組織傷害においてMDM2は組織の炎症を促進し、その阻害は強力な抗炎症効果をもたらす。MDM2の阻害は抗炎症、抗細胞分裂効果があり、がんのような炎症や過剰増殖を伴う疾患や、全身性エリテマトーデスや急速進行性糸球体腎炎といったリンパ増殖性自己免疫疾患に対し、相加的な治療効果がある可能性がある[8]。
またMdm2の過剰発現は、Mdm2とNbs1の間の直接的な相互作用によってp53非依存的にDNAの二本鎖切断修復を阻害することが示されている[9]。p53の状態に関わらず、Mdm2レベルの上昇はDNA二本鎖切断修復の遅れ、染色体異常、ゲノム不安定性を引き起こすが、Nbs1結合ドメインを欠くMdm2ではこれらの現象はみられない。これらのデータはMdm2によって誘導されるゲノム不安定性はMdm2-Nbs1間の相互作用によって媒介され、p53との結合とは独立したものである可能性を示している。
ユビキチン化の標的: p53
[編集]Mdm2の主要な標的はp53がん抑制因子である。Mdm2は、p53と相互作用しその転写活性を抑制するタンパク質として同定された。Mdm2はp53のN末端のTADに結合してブロックすることで抑制を行う。Mdm2はp53応答遺伝子であり、すなわち、Mdm2の遺伝子のの転写活性はp53によって活性化される。そのためp53が安定化さてているときにはMdm2の転写も誘導され、Mdm2のタンパク質レベルが上昇する。
E3リガーゼ活性
[編集]E3ユビキチンリガーゼであるMDM2は、p53がん抑制タンパク質の負の調節因子である。MDM2はp53に結合してユビキチン化を行い、その分解を促進する。p53はMDM2の転写を誘導するため、ネガティブフィードバックループが形成されている[10]。Mdm2は自身とp53をプロテアソームによる分解の標的とする。p53のC末端のいくつかのリジン残基がユビキチン化部位として同定されており、p53のタンパク質レベルはMdm2によってプロテアソーム依存的にダウンレギュレーションされることが示されている。Mdm2は自己ポリユビキチン化も行うとともに、協調的E3ユビキチンリガーゼであるp300と複合体を形成してp53のポリユビキチン化を行うこともできる。このように、Mdm2とp53はネガティブフィードバックによる制御ループを形成しており、p53の安定化シグナルがない状態ではp53のレベルは低く保たれる。DNA傷害などp53安定化シグナルが強いときには、このループはキナーゼやp14arfなどによって阻害される。
構造と機能
[編集]mdm2遺伝子の全長の転写産物は491アミノ酸、約56kDaのタンパク質をコードしている。このタンパク質はいくつかの保存された構造ドメインを含んでおり、N末端のp53相互作用ドメインの構造はX線結晶構造解析によって解かれている。Mdm2タンパク質にはcentral acidic domainと呼ばれる領域(230-300番残基)が存在する。このドメイン内の残基のリン酸化は、Mdm2の機能の調節に重要なようである。加えて、この領域は核外搬出シグナルと核移行シグナルを含んでおり、これらはMdm2の適切な核-細胞質間輸送に必須である。Mdm2内で保存された他のドメインとしてはジンクフィンガードメインがあるが、その機能は未解明である。
また、Mdm2はC末端にRINGドメイン(430-480番残基)を含んでおり、2つの亜鉛イオンを配位するC3-H2-C3コンセンサス配列を含んでいる。これらの残基は亜鉛の結合に必要であり、RINGドメインの適切なフォールディングに必須である。Mdm2のRINGドメインはE3ユビキチンリガーゼ活性を持ち、Mdm2の自己ユビキチン化にはこのドメインで十分である。Mdm2のRINGドメインは、核小体局在化配列を含むとともに、ヌクレオチド結合タンパク質に特徴的なWalkerモチーフが含まれているという点で独特である。RINGドメインはRNAに特異的に結合するが、その機能は未解明である。
調節
[編集]Mdm2の調節にはいくつかの機構が知られている。その機構の1つは、Mdm2タンパク質のリン酸化である。Mdm2は細胞内では複数の部位がリン酸化される。DNA傷害後のMdm2のリン酸化はタンパク質の機能の変化をもたらし、p53を安定化する。さらに、Mdm2のcentral acidic domainの特定残基のリン酸化はp53の分解標的化を促進することもあり、HIPK2はこの方法でMdm2を調節するタンパク質である。また、p16遺伝子座の代替的読み枠(alternate reading frame)の産物であるp14arfタンパク質の誘導は、p53-Mdm2相互作用を負に調節する。p14arfはMdm2と直接相互作用し、p53の転写応答をアップレギュレーションする。p14arfはMdm2を核小体へ隔離し、p53の核外輸送を阻害して活性化をもたらす。適切なp53の分解には核外輸送が必須である。
MDM2-p53相互作用の阻害剤には、cis-イミダゾリンのアナログであるヌトリンがある[11]。
Mdm2のレベルと安定性は、ユビキチン化によっても調節される。Mdm2は自己ユビキチン化を行い、プロテアソームによって分解される。また、Mdm2はユビキチン特異的プロテアーゼであるUSP7とも相互作用し、Mdm2のユビキチン化を戻してプロテアソームによる分解から防ぐ。USP7はMdm2の主要標的であるp53の分解も防ぐ。このように、Mdm2とUSP7は複雑な回路を形成してp53の安定性と活性を細かく調節しており、これらの因子のレベルはp53の機能に重要である。
相互作用
[編集]
Mdm2は次に挙げる因子と相互作用することが示されている。
- ABL1[12]
- ARRB1[13][14]
- ARRB2[13][14][15]
- CCNG1[16]
- CTBP1[17]
- CTBP2[17]
- DAXX[18]
- DHFR[19]
- EP300[20]
- ERICH3[21]
- FKBP3[22]
- FOXO4[23]
- GNL3[24]
- HDAC1[25]
- HIF1A[26][27]
- HTATIP[28]
- IGF1R[29]
- MDM4[30][31][32][33]
- NUMB[34][35]
- P16[18][36][37][38][39]
- P53[40][41]
- P73[42][43]
- PCAF[44]
- PSMD10[45]
- PSME3[46]
- RPL5[24][36][47]
- RPL11[24][36]
- PML[48][49][50][51]
- RPL26[52]
- RRM2B[53]
- RYBP[54]
- TBP[55][56]
- UBC[18][57][58]
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000135679 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000020184 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ “Amplification of a gene encoding a p53-associated protein in human sarcomas”. Nature 358 (6381): 80–3. (July 1992). doi:10.1038/358080a0. hdl:2027.42/62637. PMID 1614537.
- ^ “Hdmx modulates the outcome of p53 activation in human tumor cells”. The Journal of Biological Chemistry 281 (44): 33036–44. (November 2006). doi:10.1074/jbc.M605405200. PMID 16905769.
- ^ “MDM2 Associates with Polycomb Repressor Complex 2 and Enhances Stemness-Promoting Chromatin Modifications Independent of p53”. Molecular Cell 61 (1): 68–83. (January 2016). doi:10.1016/j.molcel.2015.12.008. PMC 6284523. PMID 26748827.
- ^ “MDM2 beyond cancer: podoptosis, development, inflammation, and tissue regeneration”. Histology and Histopathology 30 (11): 1271–82. (November 2015). doi:10.14670/HH-11-636. PMID 26062755.
- ^ Alt, Jodi R.; Bouska, Alyssa; Fernandez, Mario R.; Cerny, Ronald L.; Xiao, Hua; Eischen, Christine M. (2005-05-13). “Mdm2 binds to Nbs1 at sites of DNA damage and regulates double strand break repair”. The Journal of Biological Chemistry 280 (19): 18771–18781. doi:10.1074/jbc.M413387200. ISSN 0021-9258. PMID 15734743.
- ^ “The Functional Roles of the MDM2 Splice Variants P2-MDM2-10 and MDM2-∆5 in Breast Cancer Cells”. Translational Oncology 10 (5): 806–817. (October 2017). doi:10.1016/j.tranon.2017.07.006. PMC 5576977. PMID 28844019.
- ^ “In vivo activation of the p53 pathway by small-molecule antagonists of MDM2”. Science 303 (5659): 844–8. (February 2004). doi:10.1126/science.1092472. PMID 14704432.
- ^ “Tyrosine phosphorylation of Mdm2 by c-Abl: implications for p53 regulation”. The EMBO Journal 21 (14): 3715–27. (July 2002). doi:10.1093/emboj/cdf384. PMC 125401. PMID 12110584.
- ^ a b “Subcellular localization of beta-arrestins is determined by their intact N domain and the nuclear export signal at the C terminus”. The Journal of Biological Chemistry 278 (13): 11648–53. (March 2003). doi:10.1074/jbc.M208109200. PMID 12538596.
- ^ a b “Nedd4 mediates agonist-dependent ubiquitination, lysosomal targeting, and degradation of the beta2-adrenergic receptor”. The Journal of Biological Chemistry 283 (32): 22166–76. (August 2008). doi:10.1074/jbc.M709668200. PMC 2494938. PMID 18544533.
- ^ “Beta-arrestin 2 functions as a G-protein-coupled receptor-activated regulator of oncoprotein Mdm2”. The Journal of Biological Chemistry 278 (8): 6363–70. (February 2003). doi:10.1074/jbc.M210350200. PMID 12488444.
- ^ “Cyclin G1 has growth inhibitory activity linked to the ARF-Mdm2-p53 and pRb tumor suppressor pathways”. Molecular Cancer Research 1 (3): 195–206. (January 2003). PMID 12556559.
- ^ a b “Hdm2 recruits a hypoxia-sensitive corepressor to negatively regulate p53-dependent transcription”. Current Biology 13 (14): 1234–9. (July 2003). doi:10.1016/S0960-9822(03)00454-8. PMID 12867035.
- ^ a b c “p14ARF interacts with DAXX: effects on HDM2 and p53”. Cell Cycle 7 (12): 1836–50. (June 2008). doi:10.4161/cc.7.12.6025. PMID 18583933.
- ^ “MDM2 regulates dihydrofolate reductase activity through monoubiquitination”. Cancer Research 68 (9): 3232–42. (May 2008). doi:10.1158/0008-5472.CAN-07-5271. PMC 3536468. PMID 18451149.
- ^ “p300/MDM2 complexes participate in MDM2-mediated p53 degradation”. Molecular Cell 2 (4): 405–15. (October 1998). doi:10.1016/S1097-2765(00)80140-9. PMID 9809062.
- ^ “A comprehensive resource of interacting protein regions for refining human transcription factor networks”. PLOS ONE 5 (2): e9289. (Feb 2010). doi:10.1371/journal.pone.0009289. PMC 2827538. PMID 20195357.
- ^ “FKBP25, a novel regulator of the p53 pathway, induces the degradation of MDM2 and activation of p53”. FEBS Letters 583 (4): 621–6. (February 2009). doi:10.1016/j.febslet.2009.01.009. PMID 19166840.
- ^ “Mdm2 induces mono-ubiquitination of FOXO4”. PLOS ONE 3 (7): e2819. (2008). doi:10.1371/journal.pone.0002819. PMC 2475507. PMID 18665269.
- ^ a b c “Aberrant expression of nucleostemin activates p53 and induces cell cycle arrest via inhibition of MDM2”. Molecular and Cellular Biology 28 (13): 4365–76. (July 2008). doi:10.1128/MCB.01662-07. PMC 2447154. PMID 18426907.
- ^ “MDM2-HDAC1-mediated deacetylation of p53 is required for its degradation”. The EMBO Journal 21 (22): 6236–45. (November 2002). doi:10.1093/emboj/cdf616. PMC 137207. PMID 12426395.
- ^ “Direct interactions between HIF-1 alpha and Mdm2 modulate p53 function”. The Journal of Biological Chemistry 278 (16): 13595–8. (April 2003). doi:10.1074/jbc.C200694200. PMID 12606552.
- ^ “Regulation of tumor angiogenesis by p53-induced degradation of hypoxia-inducible factor 1alpha”. Genes & Development 14 (1): 34–44. (January 2000). doi:10.1101/gad.14.1.34. PMC 316350. PMID 10640274.
- ^ “Tip60 is targeted to proteasome-mediated degradation by Mdm2 and accumulates after UV irradiation”. The EMBO Journal 21 (7): 1704–12. (April 2002). doi:10.1093/emboj/21.7.1704. PMC 125958. PMID 11927554.
- ^ “Identification of c-Cbl as a new ligase for insulin-like growth factor-I receptor with distinct roles from Mdm2 in receptor ubiquitination and endocytosis”. Cancer Research 68 (14): 5669–77. (July 2008). doi:10.1158/0008-5472.CAN-07-6364. PMID 18632619.
- ^ “MdmX inhibits Smad transactivation”. Oncogene 21 (57): 8776–85. (December 2002). doi:10.1038/sj.onc.1205993. PMID 12483531.
- ^ “MDM2 interacts with MDMX through their RING finger domains”. FEBS Letters 447 (1): 5–9. (March 1999). doi:10.1016/S0014-5793(99)00254-9. PMID 10218570.
- ^ “MdmX is a RING finger ubiquitin ligase capable of synergistically enhancing Mdm2 ubiquitination”. The Journal of Biological Chemistry 277 (51): 49668–75. (December 2002). doi:10.1074/jbc.M208593200. PMID 12393902.
- ^ “Structure of the MDM2/MDMX RING domain heterodimer reveals dimerization is required for their ubiquitylation in trans”. Cell Death and Differentiation 15 (5): 841–8. (May 2008). doi:10.1038/sj.cdd.4402309. PMID 18219319.
- ^ “Mammalian Numb is a target protein of Mdm2, ubiquitin ligase”. Biochemical and Biophysical Research Communications 302 (4): 869–72. (March 2003). doi:10.1016/S0006-291X(03)00282-1. PMID 12646252.
- ^ “NUMB controls p53 tumour suppressor activity”. Nature 451 (7174): 76–80. (January 2008). doi:10.1038/nature06412. PMID 18172499.
- ^ a b c “Ribosomal protein L11 negatively regulates oncoprotein MDM2 and mediates a p53-dependent ribosomal-stress checkpoint pathway”. Molecular and Cellular Biology 23 (23): 8902–12. (December 2003). doi:10.1128/MCB.23.23.8902-8912.2003. PMC 262682. PMID 14612427.
- ^ “ARF promotes MDM2 degradation and stabilizes p53: ARF-INK4a locus deletion impairs both the Rb and p53 tumor suppression pathways”. Cell 92 (6): 725–34. (March 1998). doi:10.1016/S0092-8674(00)81401-4. PMID 9529249.
- ^ “Multiple interacting domains contribute to p14ARF mediated inhibition of MDM2”. Oncogene 21 (29): 4498–507. (July 2002). doi:10.1038/sj.onc.1205558. PMID 12085228.
- ^ “The Ink4a tumor suppressor gene product, p19Arf, interacts with MDM2 and neutralizes MDM2's inhibition of p53”. Cell 92 (6): 713–23. (March 1998). doi:10.1016/S0092-8674(00)81400-2. PMID 9529248.
- ^ “Mdm2 promotes the rapid degradation of p53”. Nature 387 (6630): 296–9. (May 1997). doi:10.1038/387296a0. PMID 9153395.
- ^ “Oncoprotein MDM2 is a ubiquitin ligase E3 for tumor suppressor p53”. FEBS Letters 420 (1): 25–7. (December 1997). doi:10.1016/S0014-5793(97)01480-4. PMID 9450543.
- ^ “Mdm2 binds p73 alpha without targeting degradation”. Oncogene 18 (27): 3923–9. (July 1999). doi:10.1038/sj.onc.1202781. PMID 10435614.
- ^ “MDM2 suppresses p73 function without promoting p73 degradation”. Molecular and Cellular Biology 19 (5): 3257–66. (May 1999). doi:10.1128/mcb.19.5.3257. PMC 84120. PMID 10207051.
- ^ “MDM2 inhibits PCAF (p300/CREB-binding protein-associated factor)-mediated p53 acetylation”. The Journal of Biological Chemistry 277 (34): 30838–43. (August 2002). doi:10.1074/jbc.M204078200. PMID 12068014.
- ^ “Retinoblastoma protein modulates gankyrin-MDM2 in regulation of p53 stability and chemosensitivity in cancer cells”. Oncogene 27 (29): 4034–43. (July 2008). doi:10.1038/onc.2008.43. PMID 18332869.
- ^ “Proteasome activator PA28 gamma regulates p53 by enhancing its MDM2-mediated degradation”. The EMBO Journal 27 (6): 852–64. (March 2008). doi:10.1038/emboj.2008.25. PMC 2265109. PMID 18309296.
- ^ “The ribosomal L5 protein is associated with mdm-2 and mdm-2-p53 complexes”. Molecular and Cellular Biology 14 (11): 7414–20. (November 1994). doi:10.1128/mcb.14.11.7414. PMC 359276. PMID 7935455.
- ^ “PML regulates p53 stability by sequestering Mdm2 to the nucleolus”. Nature Cell Biology 6 (7): 665–72. (July 2004). doi:10.1038/ncb1147. PMID 15195100.
- ^ “MDM2 and promyelocytic leukemia antagonize each other through their direct interaction with p53”. The Journal of Biological Chemistry 278 (49): 49286–92. (December 2003). doi:10.1074/jbc.M308302200. PMID 14507915.
- ^ “Cellular stress and DNA damage invoke temporally distinct Mdm2, p53 and PML complexes and damage-specific nuclear relocalization”. Journal of Cell Science 116 (Pt 19): 3917–25. (October 2003). doi:10.1242/jcs.00714. PMID 12915590.
- ^ “Physical and functional interactions between PML and MDM2”. The Journal of Biological Chemistry 278 (31): 29288–97. (August 2003). doi:10.1074/jbc.M212215200. PMID 12759344.
- ^ “Mdm2 regulates p53 mRNA translation through inhibitory interactions with ribosomal protein L26”. Molecular Cell 32 (2): 180–9. (October 2008). doi:10.1016/j.molcel.2008.08.031. PMC 2587494. PMID 18951086.
- ^ “ATM-mediated serine 72 phosphorylation stabilizes ribonucleotide reductase small subunit p53R2 protein against MDM2 to DNA damage”. Proceedings of the National Academy of Sciences of the United States of America 105 (47): 18519–24. (November 2008). doi:10.1073/pnas.0803313105. PMC 2587585. PMID 19015526.
- ^ “RYBP stabilizes p53 by modulating MDM2”. EMBO Reports 10 (2): 166–72. (February 2009). doi:10.1038/embor.2008.231. PMC 2637313. PMID 19098711.
- ^ “The MDM2 C-terminal region binds to TAFII250 and is required for MDM2 regulation of the cyclin A promoter”. The Journal of Biological Chemistry 272 (49): 30651–61. (December 1997). doi:10.1074/jbc.272.49.30651. PMID 9388200.
- ^ “Repression of p53-mediated transcription by MDM2: a dual mechanism”. Genes & Development 11 (15): 1974–86. (August 1997). doi:10.1101/gad.11.15.1974. PMC 316412. PMID 9271120.
- ^ “The tumour suppressor RASSF1A promotes MDM2 self-ubiquitination by disrupting the MDM2-DAXX-HAUSP complex”. The EMBO Journal 27 (13): 1863–74. (July 2008). doi:10.1038/emboj.2008.115. PMC 2486425. PMID 18566590.
- ^ “CARPs enhance p53 turnover by degrading 14-3-3sigma and stabilizing MDM2”. Cell Cycle 7 (5): 670–82. (March 2008). doi:10.4161/cc.7.5.5701. PMID 18382127.
関連文献
[編集]- “Molecular analysis and chromosomal mapping of amplified genes isolated from a transformed mouse 3T3 cell line”. Somatic Cell and Molecular Genetics 13 (3): 235–44. (May 1987). doi:10.1007/BF01535205. PMID 3474784.
- “Regulation of transcription functions of the p53 tumor suppressor by the mdm-2 oncogene”. Molecular Medicine 1 (2): 142–52. (January 1995). doi:10.1007/BF03401562. PMC 2229942. PMID 8529093.
- “Mdm2 is a RING finger-dependent ubiquitin protein ligase for itself and p53”. The Journal of Biological Chemistry 275 (12): 8945–51. (March 2000). doi:10.1074/jbc.275.12.8945. PMID 10722742.
- “Functions of the MDM2 oncoprotein”. Cellular and Molecular Life Sciences 55 (1): 96–107. (January 1999). doi:10.1007/s000180050273. PMID 10065155.
- “Multiple sites of in vivo phosphorylation in the MDM2 oncoprotein cluster within two important functional domains”. FEBS Letters 478 (1–2): 183–6. (July 2000). doi:10.1016/S0014-5793(00)01850-0. PMID 10922493.
- “Oncoprotein MDM2 is a ubiquitin ligase E3 for tumor suppressor p53”. FEBS Letters 420 (1): 25–7. (December 1997). doi:10.1016/S0014-5793(97)01480-4. PMID 9450543.
- “Activity of MDM2, a ubiquitin ligase, toward p53 or itself is dependent on the RING finger domain of the ligase”. Oncogene 19 (11): 1473–6. (March 2000). doi:10.1038/sj.onc.1203464. PMID 10723139.
- “Regulation of p53 stability by Mdm2”. Nature 387 (6630): 299–303. (May 1997). doi:10.1038/387299a0. PMID 9153396.
- “Structure of the MDM2 oncoprotein bound to the p53 tumor suppressor transactivation domain”. Science 274 (5289): 948–53. (November 1996). doi:10.1126/science.274.5289.948. PMID 8875929.
- “Posttranslational modification of MDM2”. Molecular Cancer Research 1 (14): 1017–26. (December 2003). PMID 14707285.
- “An N-terminal p14ARF peptide blocks Mdm2-dependent ubiquitination in vitro and can activate p53 in vivo”. Oncogene 19 (19): 2312–23. (May 2000). doi:10.1038/sj.onc.1203593. PMID 10822382.
- “MDM2--master regulator of the p53 tumor suppressor protein”. Gene 242 (1–2): 15–29. (January 2000). doi:10.1016/S0378-1119(99)00487-4. PMID 10721693.
- “The mdm-2 oncogene product forms a complex with the p53 protein and inhibits p53-mediated transactivation”. Cell 69 (7): 1237–45. (June 1992). doi:10.1016/0092-8674(92)90644-R. PMID 1535557.
- “DNA damage-induced phosphorylation of p53 alleviates inhibition by MDM2”. Cell 91 (3): 325–34. (October 1997). doi:10.1016/S0092-8674(00)80416-X. PMID 9363941.
- “P19(ARF) stabilizes p53 by blocking nucleo-cytoplasmic shuttling of Mdm2”. Proceedings of the National Academy of Sciences of the United States of America 96 (12): 6937–41. (June 1999). doi:10.1073/pnas.96.12.6937. PMC 22020. PMID 10359817.
- “Nucleocytoplasmic shuttling of oncoprotein Hdm2 is required for Hdm2-mediated degradation of p53”. Proceedings of the National Academy of Sciences of the United States of America 96 (6): 3077–80. (March 1999). doi:10.1073/pnas.96.6.3077. PMC 15897. PMID 10077639.