遺伝子発現の調節
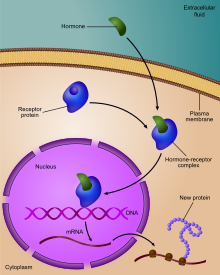

遺伝子発現の調節(いでんしはつげんのちょうせつ)には、細胞が特定の遺伝子産物(タンパク質やRNA)の合成を増加または減少させる幅広いメカニズムが含まれる。生物学において、遺伝子発現のための高度なプログラムは、発達経路の誘導、環境刺激への応答、新たな食料源への適応など、幅広い現象で観察される。転写の開始からRNAのプロセシング、そしてタンパク質の翻訳後修飾に至るまで、遺伝子発現のあらゆるステップが事実上調節が可能である。遺伝子発現の制御因子はしばしば相互に影響し合い、遺伝子調節ネットワークを形成している。
遺伝子発現の調節は、ウイルス、原核生物そして真核生物において必須のプロセスである。遺伝子発現を調節し、必要時にタンパク質の発現を行うことで、生物の多能性、適応性は高められている。1951年にバーバラ・マクリントックはトウモロコシの種子の色の形成におけるActivator(Ac)とDissociator(Ds)という2つの遺伝子座間の相互作用を示していた。しかしながら一般的には、1961年のフランソワ・ジャコブとジャック・モノーによるラクトースオペロンの同定が遺伝子調節システムの最初の発見であると見なされている。彼らは、ラクトースの代謝に関与するいくつかの酵素が、ラクトースが存在しグルコースが存在しないときにのみ、大腸菌で発現されることを示した。
多細胞生物では、遺伝子発現調節は胚における細胞の分化や形態形成を駆動し、異なる遺伝子発現プロファイルを持つ異なるタイプの細胞を、同一のゲノム配列から作り出す。このプロセスは、進化がどのように起こっているかを分子レベルで説明するものであり、進化発生生物学(通称"evo-devo")の研究の核心をなしている。
遺伝子発現の変化は、受容体の活性化または不活性化などによって開始される。
調節される遺伝子発現のステージ
[編集]DNAからRNAが転写されるステップからタンパク質の翻訳後修飾のステップまで、遺伝子発現のあらゆる段階が調節されうる。遺伝子発現が調節されるステージのリストを次に挙げるが、最も広く調節に利用されているのは転写の開始である。
DNAの修飾
[編集]真核生物では、DNAの大部分の領域で、DNAへのアクセシビリティはその領域のクロマチン構造に依存する。クロマチン構造はDNAのメチル化、ノンコーディングRNA、またはDNA結合タンパク質によるヒストンの修飾によって変化する。そのため、これらの修飾によって遺伝子の発現は上昇もしくは低下する。これら遺伝子発現を調節する修飾のうちのいくつかは遺伝し、エピジェネティックな調節と呼ばれる。
構造的な変化
[編集]DNAの転写レベルは、その構造によって規定される。一般的に、DNAのパッキングの密度は転写の頻度の指標となる。ヌクレオソームと呼ばれる、ヒストン八量体からなるタンパク質複合体はDNAのスーパーコイル構造を形成するが、その構造は、ヒストンのリン酸化のような修飾によって一時的に、そしてメチル化のような修飾によってより恒久的に変化する。このような変化は、多かれ少なかれ遺伝子発現レベルの恒久的な変化に関与していると考えられている[1]。
化学的な変化
[編集]DNAのメチル化は遺伝子のサイレンシングの一般的な方法である。一般的にDNAは、メチル基転移酵素によってCpGジヌクレオチド配列のシトシン残基がメチル化される。CpG配列が密集している領域はCpGアイランドとも呼ばれる。DNAの特定の領域のメチル化のパターンの分析は、バイサルファイトシークエンシングと呼ばれる手法によって行われる。バイサルファイト処理によって、メチル化されていないシトシン残基はウラシル残基に変換されるが、メチル化されたシトシン残基は変化しない。この違いがDNAシーケンシングや、もしくはパイロシークエンシングやMassArrayといったSNP定量のための手法を用いてCGジヌクレオチドのC/T比を測定することで分析される。メチル化のパターンの異常は発がんに関与していると考えられている[2]。
ヒストンのアセチル化も、転写に重要な過程である。CREB結合タンパク質のようなヒストンアセチル基転移酵素は、DNAをヒストン複合体から解離させ、転写を進行させる。しばしば、DNAのメチル化とヒストンの脱アセチル化は遺伝子のサイレンシングにおいて協調的に働く。これらはDNAをより密にパッキングし遺伝子発現を低下させるためのシグナルとなっていると考えられる[要出典]。
転写の調節
[編集]
転写の調節とは、いつ転写が起こるか、どれだけのRNAがつくられるかの制御である。RNAポリメラーゼによる遺伝子の転写はいくつかのメカニズムによって調節される。
Specificity factor(原核生物の転写におけるσ因子など)は、あるプロモーターまたはプロモーターのセットについてRNAポリメラーゼの特異性を変化させ、結合しやすくまたは結合しにくくする。
リプレッサーは、オペレーターと呼ばれる、プロモーターと近接もしくは重複する配列に結合する。リプレッサーの結合は、RNAポリメラーゼがDNA鎖に沿って進行するのを妨げ、遺伝子の発現を妨げる。右の図は、lacオペロンのリプレッサーによる調節機構を示している。
基本転写因子は、RNAポリメラーゼをタンパク質コーディング領域の開始位置に配置し、その後RNAポリメラーゼを解放してmRNAを転写させる。
アクチベーターは、RNAポリメラーゼと特定のプロモーターの間の相互作用を強化し、遺伝子の発現を促進する。アクチベーターは、RNAポリメラーゼのサブユニットとの直接的な相互作用、またはDNA構造の変化のような間接的な方法で、RNAポリメラーゼのプロモーターへの誘引を強化する。
エンハンサーは、アクチベーターが結合するDNA上の領域であり、アクチベーターはDNAをループさせて特定のプロモーターを転写開始複合体へもたらす。エンハンサーは原核生物よりも真核生物ではるかに一般的であり、原核生物では現在までに数例しか見つかっていない[3]。
サイレンサーは、特定の転写因子が結合したときに遺伝子の発現が抑制されるDNA配列の領域である。
がんにおける転写調節
[編集]脊椎動物では、大部分の遺伝子プロモーターはCpGアイランドと呼ばれる無数のCpG部位を持つ領域を含んでいる[4]。遺伝子のプロモーターのCpG部位の多くがメチル化されているとき、遺伝子はサイレンシングされる[5]。大腸がんでは一般的に、3から6個のドライバー変異(driver mutation)と33から66個のヒッチハイカー(hitchhiker)もしくはパッセンジャー変異(passenger mutation)が見つかる[6]が、がんの進行においては突然変異よりも転写のサイレンシングが重要である可能性がある。例えば、大腸がんでは600から800の遺伝子がCpGアイランドのメチル化によって転写がサイレンシングされている(がんにおける転写調節を参照)。がんにおける転写の抑制は、miRNAの発現の変化などのエピジェネティックなメカニズムによっても引き起こされる[7]。乳がんでは、BRCA1の転写抑制は、BRCA1のプロモーター領域の過剰なメチル化によるものよりも、miRNA-182の過剰発現によるものがより高頻度である(Low expression of BRCA1 in breast and ovarian cancersを参照)。
嗜癖における転写調節
[編集]嗜癖の主要な特徴の1つはその持続性である。持続的な行動の変化は、脳の特定の領域におけるエピジェネティックな変化に起因する、長期的な変化によるものであると考えられている[8]。薬物の乱用は脳に3つのエピジェネティックな変化を引き起こす。(1) ヒストンのアセチル化とメチル化、(2) DNAのCpG部位のメチル化、(3) miRNAのエピジェネティックな発現上昇や発現抑制、の3つである[8][9]。(コカイン嗜癖のエピジェネティクスを参照)
マウスでは、慢性的なニコチンの摂取は、ヒストンのアセチル化によって遺伝子発現のエピジェネティックな制御が変化する。この変化によって、嗜癖に重要なFosBタンパク質の脳での発現が上昇する[10]。タバコへの依存については、非喫煙者、喫煙者、30年以下の禁煙期間の人を含め、16,000人を対象とした研究がなされている[11]。喫煙者の血球細胞では、解析されたゲノム中の450,000のCpG部位のうち、18,000以上のCpG部位において、メチル化が高頻度で変化していた。これらのCpG部位は7,000以上の遺伝子にわたって位置しており、これはヒトの既知の遺伝子の約1/3を占める。CpG部位のメチル化の変化の大部分は、5年の禁煙期間によって非喫煙者のレベルに戻っていた。しかしながら、942遺伝子、2,568ヶ所のCpG部位に関しては、喫煙経験者と非喫煙者とでメチル化の差が存在したままであった。このような持続的なエピジェネティックな変化は、遺伝子発現に影響を与える "moleular scars"[9]として見ることができる。
コカイン[12]、メタンフェタミン[13][14]、アルコール[15]、タバコの喫煙産物[16]を含め、乱用薬物はすべて、齧歯類モデルで脳のDNA損傷を引き起こす。DNA損傷の修復の際に、損傷部位のDNAのメチル化やヒストンのアセチル化/メチル化パターンが変化し、これによってクロマチンにエピジェネティックな傷が残される[17]。このようなエピジェネティックな傷が、嗜癖でみられる持続的なエピジェネティックな変化に寄与していると考えられる。
転写後調節
[編集]DNAが転写されてmRNAが合成された後、どれだけのmRNAがタンパク質へ翻訳されるかについて、何らかの調節機構が存在しなければならない。細胞では、キャッピングの調節、スプライシング、ポリ(A)テールの付加、配列特異的な核外輸送、そして、いくつかの状況においてはRNA転写産物の隔離によって、調節が行われている。これらの過程は真核生物では起こるが、原核生物では起こらない。
3'-UTRとmiRNA
[編集]mRNAの3'非翻訳領域(3'-UTR)は、しばしば転写後に遺伝子発現に影響を与える調節配列を含んでいる[18]。このような3'-UTRは、調節タンパク質やmiRNAの結合部位を含んでいる。miRNAは3'-UTRの特定の部位に結合することで、翻訳を阻害するか、もしくは直接的に転写産物の分解を引き起こし、さまざまなmRNAの遺伝子発現を低下させる。また3'-UTRは、サイレンサー領域を持っていることもあり、リプレッサータンパク質がこの領域に結合してmRNAの発現阻害が行われる。
3'-UTRのmiRNAが結合する配列は、miRNA認識配列(miRNA response element, MRE)と呼ばれ、MREは3'-UTR中の制御モチーフのうち約半分を占める。
2014年の段階で、miRNAの配列とアノテーションのアーカイブであるmiRBaseウェブサイト[19]には、233の生物種で28,645のエントリーが存在する。これらのうち、1,881のmiRNAはヒトの遺伝子座にアノテーションされている。miRNAは平均して約400の標的mRNAを持ち、数百の遺伝子の発現に影響を与えると予測されている[20]。Freidmanら[20]は、ヒトのmRNAの3'-UTRのmiRNAの標的部位のうち、45,000以上の部位がバックグラウンドレベルよりも高く保存されており、ヒトのタンパク質をコードする遺伝子の60%以上がmiRNAと対合を保つよう選択圧を受けていると予測している。
1種類のmiRNAが数百種類の異なるmRNAの安定性を低下させることは、直接的な実験により示されている[21]。他の実験では、1種類のmiRNAが数百のタンパク質の合成を抑制するが、一方でその抑制はしばしば1/2以下と穏やかであることが示されている[22][23]。
miRNAによる遺伝子発現の調節異常の影響は、がんにおいて重要であると考えられている[24]。2015年の論文では、消化器がんにおいて、9つのmiRNAがエピジェネティックな変化を受けており、DNA修復酵素の発現抑制に寄与していると報告されている[25]。
また、遺伝子発現のmiRNAによる調節異常の影響は、統合失調症、双極性障害、うつ病、パーキンソン病、アルツハイマー病、自閉症といった精神・神経疾患においても重要であると考えられている[26][27][28]。
翻訳の調節
[編集]mRNAの翻訳もまた多数のメカニズムによって制御されているが、そのほとんどは開始の段階で行われる。リボソーム小サブユニットのリクルートは、mRNAの二次構造、アンチセンスRNAの結合、タンパク質の結合などによって制御される。原核生物と真核生物の双方において、多数のRNA結合タンパク質が存在し、それらはしばしば、転写産物の二次構造によって標的配列へ差し向けられる。その二次構造自体も、温度やリガンドの存在(アプタマー)などの条件によって変化する。いくつかの転写産物はリボザイムとして機能し、自身の発現を制御する。
遺伝子発現制御の例
[編集]- 酵素誘導 (enzyme induction) は、分子(薬剤など)が酵素の発現を誘導(開始または向上)する過程である。
- キイロショウジョウバエにおける熱ショックタンパク質の誘導。
- lacオペロンは、遺伝子の発現がどのように調節されるかの興味深い例である。
- ウイルスは、わずかな遺伝子しか持っていないにもかかわらず、自身の遺伝子の発現を調節するメカニズムを持っている。一般的に遺伝子発現は、初期(early phase)と後期(late phase)に分かれており、anti-terminator(λファージ)や splicing modulator(HIV)によって調節されている。
- Gal4は、GAL1、GAL7、GAL10(いずれも酵母のガラクトース代謝に関わる)の発現を制御する転写活性化因子である。GAL4/UASシステムは、遺伝子発現の研究のため、門を越えて幅広い生物種で利用されている[29]。
発生生物学
[編集]多数の制御システムが発生生物学の研究によって明らかにされている。その例を次に挙げる:
- ホメオティック遺伝子:遺伝子クラスターのゲノム上の位置と、体の前後軸に沿った入れ子状の発現パターン、発生過程での時間的な発現順序が対応している(共直線性, colinearity)。
- 手のパターン形成(指-指間部): 極性化活性体から分泌される誘導因子 Shhの濃度勾配は、活性型 Gli3の勾配を作り出す。Gli3はグレムリンを活性化し、分泌性因子であるBMPを阻害する。この反応拡散系の結果、交互の活性パターンが形成される。
- 体節形成:均一な組織(未分節中胚葉)から体節を作り出す過程であり、体節は体の前側から後側へ順番に作られてゆく。この過程は有羊膜類ではおそらく、2つの対向する勾配(前側のレチノイン酸、後側のWntとFGF)によって支配されており、この勾配とFGF、Notch、逆位相のWntによる振動パターン(分節時計、segmentation clock)との共役によって分節が行われていると考えられている[30]。
- ショウジョウバエの体細胞における性決定:性染色体にコードされる遺伝子と常染色体遺伝子との比の検出を必要とし、その結果雌ではsex-lethalスプライシング因子が合成されて、doublesex遺伝子の雌型アイソフォームが発現する[31]。
遺伝子回路
[編集]アップレギュレーションとダウンレギュレーション
[編集]アップレギュレーションは細胞内外からのシグナルによって細胞内に引き起こされる過程であり、1つもしくはそれ以上の遺伝子の発現が上昇し、それらの遺伝子によってコードされるタンパク質が増加する過程である。逆に、ダウンレギュレーションは遺伝子発現が低下し、対応するタンパク質が減少する過程である。
- アップレギュレーションは、例えば、細胞にある種の受容体が欠乏しているときに起こる。この場合、より多くの受容体タンパク質が合成されて細胞膜に輸送され、その結果、細胞の感受性が通常の状態に戻り、恒常性が再構築される。
- ダウンレギュレーションは、例えば、細胞が神経伝達物質やホルモン、薬剤によって長期間過剰に刺激されたときに起こる。細胞を保護するために受容体タンパク質の発現は減少する(タキフィラキシーを参照)。
誘導システムと抑制システム
[編集]遺伝子調節はシステムの応答によって次のようにまとめられる:
- 誘導システム (inducible system) - 誘導システムは、インデューサーと呼ばれる、遺伝子を発現させる分子が存在しない限り、オフの状態である。この分子は「発現を誘導する」と表現される。その様式は、原核細胞と真核細胞の違いだけでなく制御機構にも依存する。
- 抑制システム (reressible system) - 抑制システムでは、コリプレッサーと呼ばれる、遺伝子の発現を抑制する因子が存在する場合を除き、オンの状態である。この分子は「発現を抑制する」と表現される。その様式は、原核細胞と真核細胞の違いだけでなく制御機構にも依存する。
GAL4/UASシステムは、誘導システムと抑制システムの両方の例である。Gal4はGAL1/GAL7/GAL10の転写を活性化するために上流の活性化配列 (upstream activation sequence, UAS) に結合し、発現を誘導する。一方で、グルコースの存在に応答するMIG1はGAL4を阻害し、GAL1/GAL7/GAL10の発現を抑制する[32]。
理論的な回路
[編集]既知の遺伝子を組み合わせることで、人工的な遺伝子発現ネットワークを形成することができる。
- リプレッサー/インデューサー: 遺伝子の発現を変化させるセンサー
- ネガティブフィードバック: 遺伝子産物が直接的・間接的に自らの合成をダウンレギュレーションする。次のような結果が考えられる。
- 転写産物のレベルを一定、もしくは因子に比例したレベルに保つ
- ポジティブフードバックループと組み合わせて、反応の暴走を防ぐ
- 転写と翻訳の時間差を利用して振動子を作り出す(mRNAとタンパク質の半減期が短い場合)
- ポジティブフィードバック: 遺伝子産物が直接的・間接的に自らの合成をアップレギュレーションする。次のような結果が考えられる。
- シグナルの増幅
- 双安定性(bistable)スイッチ(2つの遺伝子が相互に阻害し合い、双方がポジティブフィードバックを形成している場合)
- パターン形成
遺伝子発現調節の研究手法
[編集]一般的に、遺伝子発現の差を調べる実験では、どの遺伝子がどの程度変化したかを決定するために、定常状態の全細胞からの抽出RNAが用いられる。この手法では、どこで調節が行われているかについての情報は得られず、競合する調節プロセスを覆い隠してしまう可能性があるが、現在でも定量PCRやDNAマイクロアレイなどで最も広く用いられる手法である。
遺伝子発現の研究には、さまざまなステージを観察するいくつかの手法がある。真核生物においては、次のようなものが含まれる:
- ある領域の局所的なクロマチンの環境は、RNAポリメラーゼII、ヒストンH3の修飾、Trithorax群タンパク質、Polycomb群タンパク質や、そのほか、良い抗体が利用できるDNA結合エレメントを用いたプルダウンによって、ChIP-chip法で決定される。
- エピスタティックな相互作用は、synthetic genetic arrayによって調べられる。
- 転写後調節のために、転写率と全RNA量は大きく異なる。転写率の測定には、nuclear run-onアッセイが用いられる。放射性同位体を用いたラベリングに代わって、チオール基を用いたラベリング法も開発されている[33]。
- 核内で合成されるRNAのうち核外に出るのは5%のみであり[34]、イントロンだけでなく、abortive initiation(転写開始のの失敗)による産物、ナンセンスな転写産物が分解される。核内と細胞質の量の差は、穏和な細胞溶解によって2つのフラクションに分画することで観察が可能になる[35]。
- オルタナティブスプライシングは、スプライシングアレイやタイリングアレイによって分析される。(DNAマイクロアレイを参照)
- 生体内のRNAはタンパク質と結合し、リボヌクレオタンパク質複合体を形成している。特定のタンパク質に結合している転写産物の量は RIP-Chipによって分析される。デキャッピング酵素 Dcp2に結合するRNAは隔離された転写産物の、リボソームに結合するものは活発に翻訳されている転写産物の指標となる。
- タンパク質の量は質量分析によって分析される。質量分析のデータは定量PCRのデータと比較可能である。マイクロアレイのデータは相対値であり絶対値ではないことに注意を要する。
- RNAとタンパク質の分解率は、転写阻害剤(アクチノマイシンDやα-アマニチン)や翻訳阻害剤(シクロヘキシミド)をそれぞれ利用して計測される。
脚注
[編集]- ^ “DNA methylation patterns associate with genetic and gene expression variation in HapMap cell lines”. Genome Biology 12 (1): R10. (2011). doi:10.1186/gb-2011-12-1-r10. PMC 3091299. PMID 21251332.
- ^ “Altered chromosomal methylation patterns accompany oncogene-induced transformation of human bronchial epithelial cells”. Cancer Research 53 (7): 1684–9. (Apr 1993). PMID 8453642.
- ^ “The prokaryotic enhancer binding protein NTRC has an ATPase activity which is phosphorylation and DNA dependent”. The EMBO Journal 11 (6): 2219–28. (Jun 1992). PMC 556689. PMID 1534752.
- ^ “A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters”. Proc. Natl. Acad. Sci. U.S.A. 103 (5): 1412–7. (2006). Bibcode: 2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC 1345710. PMID 16432200.
- ^ “DNA methylation patterns and epigenetic memory”. Genes Dev. 16 (1): 6–21. (2002). doi:10.1101/gad.947102. PMID 11782440.
- ^ “Cancer genome landscapes”. Science 339 (6127): 1546–58. (2013). Bibcode: 2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ “MicroRNAs in the DNA Damage/Repair Network and Cancer”. Int J Genom 2014: 820248. (2014). doi:10.1155/2014/820248. PMC 3926391. PMID 24616890.
- ^ a b “Epigenetic mechanisms of drug addiction”. Neuropharmacology 76 Pt B: 259–68. (January 2014). doi:10.1016/j.neuropharm.2013.04.004. PMC 3766384. PMID 23643695.
- ^ a b “Transcriptional and epigenetic mechanisms of addiction”. Nat. Rev. Neurosci. 12 (11): 623–37. (October 2011). doi:10.1038/nrn3111. PMC 3272277. PMID 21989194.
- ^ “Molecular mechanism for a gateway drug: epigenetic changes initiated by nicotine prime gene expression by cocaine”. Sci Transl Med 3 (107): 107ra109. (November 2011). doi:10.1126/scitranslmed.3003062. PMC 4042673. PMID 22049069.
- ^ “Epigenetic Signatures of Cigarette Smoking”. Circ Cardiovasc Genet 9 (5): 436–447. (October 2016). doi:10.1161/CIRCGENETICS.116.001506. PMC 5267325. PMID 27651444.
- ^ “Cocaine induces DNA damage in distinct brain areas of female rats under different hormonal conditions”. Clin. Exp. Pharmacol. Physiol. 41 (4): 265–9. (April 2014). doi:10.1111/1440-1681.12218. PMID 24552452.
- ^ “Methamphetamine induces DNA damage in specific regions of the female rat brain”. Clin. Exp. Pharmacol. Physiol. 42 (6): 570–5. (June 2015). doi:10.1111/1440-1681.12404. PMID 25867833.
- ^ “The peroxidative DNA damage and apoptosis in methamphetamine-treated rat brain”. J. Med. Invest. 55 (3-4): 241–5. (August 2008). PMID 18797138.
- ^ “Alcohol induces DNA damage and the Fanconi anemia D2 protein implicating FANCD2 in the DNA damage response pathways in brain”. Alcohol. Clin. Exp. Res. 32 (7): 1186–96. (July 2008). doi:10.1111/j.1530-0277.2008.00673.x. PMID 18482162.
- ^ “Biomarkers of disease can be detected in mice as early as 4 weeks after initiation of exposure to third-hand smoke levels equivalent to those found in homes of smokers”. Clin. Sci. 131 (19): 2409–2426. (October 2017). doi:10.1042/CS20171053. PMID 28912356.
- ^ “Epigenome Maintenance in Response to DNA Damage”. Mol. Cell 62 (5): 712–27. (June 2016). doi:10.1016/j.molcel.2016.04.006. PMC 5476208. PMID 27259203.
- ^ “Processing and transcriptome expansion at the mRNA 3′ end in health and disease: finding the right end”. Eur J Physiol 468: 993–1012. (2016). doi:10.1007/s00424-016-1828-3. PMC 4893057. PMID 27220521.
- ^ miRBase.org
- ^ a b “Most mammalian mRNAs are conserved targets of microRNAs”. Genome Research 19 (1): 92–105. (Jan 2009). doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ “Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs”. Nature 433 (7027): 769–73. (Feb 2005). Bibcode: 2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193.
- ^ “Widespread changes in protein synthesis induced by microRNAs”. Nature 455 (7209): 58–63. (Sep 2008). Bibcode: 2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040.
- ^ “The impact of microRNAs on protein output”. Nature 455 (7209): 64–71. (Sep 2008). Bibcode: 2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ “Mechanisms and role of microRNA deregulation in cancer onset and progression”. Genetics and Molecular Biology 34 (3): 363–70. (Jul 2011). doi:10.1590/S1415-47572011000300001. PMC 3168173. PMID 21931505.
- ^ “Epigenetic reduction of DNA repair in progression to gastrointestinal cancer”. World Journal of Gastrointestinal Oncology 7 (5): 30–46. (May 2015). doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ “Micro spies from the brain to the periphery: new clues from studies on microRNAs in neuropsychiatric disorders”. Frontiers in Cellular Neuroscience 8: 75. (2014). doi:10.3389/fncel.2014.00075. PMC 3949217. PMID 24653674.
- ^ “The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders”. Frontiers in Psychiatry 3: 39. (2012). doi:10.3389/fpsyt.2012.00039. PMC 3336189. PMID 22539927.
- ^ “MicroRNA and Posttranscriptional Dysregulation in Psychiatry”. Biological Psychiatry 78 (4): 231–9. (Aug 2015). doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ “A history of research on yeasts 7: enzymic adaptation and regulation”. Yeast 21: 703–746. (2004). doi:10.1002/yea.1113.
- ^ “Segmental patterning of the vertebrate embryonic axis”. Nat Rev Genet 9 (5): 370–82. (May 2008). doi:10.1038/nrg2320. PMID 18414404.
- ^ Gilbert SF (2003). Developmental biology, 7th ed., Sunderland, Mass: Sinauer Associates, 65–6. ISBN 0-87893-258-5.
- ^ “Control of yeast GAL genes by MIG1 repressor: a transcriptional cascade in the glucose response”. EMBO J. 10 (11): 3373–7. (1991). PMC 453065. PMID 1915298.
- ^ “Control of gene expression during T cell activation: alternate regulation of mRNA transcription and mRNA stability”. BMC Genomics 6: 75. (2005). doi:10.1186/1471-2164-6-75. PMC 1156890. PMID 15907206.
- ^ “The balance sheet for transcription: an analysis of nuclear RNA metabolism in mammalian cells”. FASEB Journal 14 (2): 242–54. (Feb 2000). PMID 10657981.
- ^ “Genome-wide analyses show that nuclear and cytoplasmic RNA levels are differentially affected by dioxin”. Biochimica et Biophysica Acta 1759 (8-9): 388–402. (2006). doi:10.1016/j.bbaexp.2006.07.005. PMID 16962184.
関連文献
[編集]- Latchman, David S. (2005). Gene regulation: a eukaryotic perspective. Psychology Press. ISBN 978-0-415-36510-9
関連項目
[編集]- Artificial transcription factors (転写因子を模倣する低分子)
- Cellular model
- Conserved non-coding DNA sequence
- エンハンサー
- Gene structure
- Spatiotemporal gene expression
外部リンク
[編集]- Plant Transcription Factor Database and Plant Transcriptional Regulation Data and Analysis Platform
- Regulation of Gene Expression (MeSH) - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- ChIPBase - ChIP-seqデータから得られた、ノンコーディングRNAとタンパク質コード遺伝子の転写制御ネットワークのデータベース
