「デオキシリボ核酸」の版間の差分
内部リンク除去 |
→top: 誤訳: 複合糖質(多糖) → 複合多糖 (complex carbohydrates) |
||
| (4人の利用者による、間の5版が非表示) | |||
| 1行目: | 1行目: | ||
{{redirect|DNA |
{{redirect|DNA}} |
||
[[File:DNA Structure+Key+Labelled.pn NoBB.png|thumb|right|upright=1.55|<small>(左)</small> DNA[[二重らせん]]の構造 ([[B型DNA|B-DNA]])。構造内の[[原子]]は[[元素]]ごとに色分けされている。<small>(右)</small> 二組の[[塩基対]]の詳細構造。]][[File:Phosphate backbone.jpg|thumb|糖リン酸主鎖と塩基からなるDNAの構造]] |
|||
[[画像:DNA animation.gif|thumb|right|DNA二重らせん構造の一部を、[[三次元]]立体構造で表現した図。]] |
|||
{{Genetics sidebar}} |
|||
'''デオキシリボ核酸'''(デオキシリボかくさん、{{lang-en-short|<u>d</u>eoxyribo<u>n</u>ucleic <u>a</u>cid}}、'''DNA''')は、[[核酸]]の一種で、二重らせん構造。[[地球]]上の多くの[[生物]]において[[遺伝]]情報の継承と[[発現]]を担う[[高分子]][[生体物質]]である<ref name="Tamra41-1" />。 |
|||
'''デオキシリボ核酸'''(デオキシリボかくさん、{{Lang-en-short|deoxyribonucleic acid}}、'''DNA'''<ref>{{MerriamWebsterDictionary|deoxyribonucleic acid|accessdate=2023-12-13}}</ref>)は、2本の[[ポリヌクレオチド]]鎖が互いに巻きついて[[二重らせん]]を形成している[[ポリマー]]である。このポリマーは、すべての既知の[[生物]]と多くの[[ウイルス]]の発生、機能、成長、および[[生殖]]のための[[遺伝学|遺伝的]]命令を伝達する。DNAは[[リボ核酸]]({{Lang-en-short|ribonucleic acid}}、RNA)とともに[[核酸]]と総称される。核酸は[[タンパク質]]、[[脂質]]、[[多糖|複合多糖]]と並んで、すべての既知の[[生命|生命体]]にとって不可欠な4大[[生体高分子]]のひとつである。 |
|||
== 構造 == |
|||
=== 構成物質と二重らせん構造 === |
|||
[[画像:AGCT DNA mini.png|thumb|200px|相補的塩基対:AとT、GとCが水素結合でつながる。]] |
|||
DNAの二本鎖は、[[ヌクレオチド]]と呼ばれるより単純な[[モノマー|単量体]]単位から構成されていることから、ポリヌクレオチドと呼ばれる<ref>{{Cite book |vauthors= Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P |title= Molecular Biology of the Cell |edition= 6th |publisher= Garland |year= 2014 |url= http://www.garlandscience.com/product/isbn/9780815344322 |page= Chapter 4: DNA, Chromosomes and Genomes |isbn= 978-0-8153-4432-2 |url-status=live |archive-url= https://web.archive.org/web/20140714210549/http://www.garlandscience.com/product/isbn/9780815344322 |archive-date= 14 July 2014 |df= dmy-all }}</ref><ref>{{cite web |vauthors=Purcell A |title=DNA |url=http://basicbiology.net/micro/genetics/dna |website=Basic Biology |url-status=live |archive-url=https://web.archive.org/web/20170105045651/http://basicbiology.net/micro/genetics/dna/ |archive-date=5 January 2017 |access-date=2015-07-28}}</ref>。各ヌクレオチドは、4つの窒素含有[[核酸塩基]]([[シトシン]]: C、[[グアニン]]: G、[[アデニン]]: A、[[チミン]]: T)のうちの1つ、デオキシリボースと呼ばれる[[単糖|糖]]、および[[リン酸エステル|リン酸基]]で構成されている。あるヌクレオチドの糖と、次のヌクレオチドのリン酸が[[共有結合]]([[ホスホジエステル結合]]と呼ばれる)によって鎖状に結合し、糖-リン酸が交互に繰り返される[[主鎖]]が形成される。二本のポリヌクレオチド鎖の窒素塩基は、[[塩基対|塩基対合]]則(AとT、CとG)に従って[[水素結合]]で結合し、二本鎖DNAを形成する。窒素塩基は、単環の[[ピリミジン]]と二重環の[[プリン (化学)|プリン]]という2つのグループに分類される。DNAでは、チミンとシトシンがピリミジン、アデニンとグアニンがプリンである。 |
|||
DNAは'''[[デオキシリボース]]'''(五炭[[糖]])と'''[[リン酸]]'''、'''[[塩基]]''' から構成される核酸である。塩基は[[プリン塩基]]である[[アデニン]](A)と[[グアニン]](G)、[[ピリミジン塩基]]である[[シトシン]](C)と[[チミン]](T)の4種類がある<ref name="Tamra38">[[#田村(2010)|田村(2010)、p.38-40、I細胞生物学の基礎、5.情報高分子(2):ヌクレオチドと核酸、5-1 核酸を構成するヌクレオチド]]</ref>。2-[[デオキシリボース]]の1'位に塩基が結合したものをデオキシ'''ヌクレオシド'''、この[[ヌクレオシド]]のデオキシリボースの5'位にリン酸が結合したものをデオキシ'''ヌクレオチド'''と呼ぶ<ref name="Tamra38" />。 |
|||
二本鎖DNAの両鎖は同一の[[セントラルドグマ|生物学的情報]]を保存している。この情報は2本の鎖が分離するときに[[DNA複製|複製]]される。DNAの大部分([[ヒト]]では98%以上)は[[ノンコーディングDNA|ノンコーディング]]であり、これらの部分は[[一次構造|タンパク質配列]]のパターンとしては機能しない。DNAの2本の鎖は互いに反対方向に走っているため、[[逆平行 (生化学)|逆平行]]になっている。それぞれの糖には4種類の核酸塩基(または塩基)のうちの1つが結合している。遺伝情報を[[遺伝コード|コード]](符号化)するのは、主鎖に沿ったこれら4種類の[[塩基配列|核酸塩基の配列]]である。[[リボ核酸|RNA]](リボ核酸)鎖はDNA鎖を鋳型として[[転写 (生物学)|転写]]と呼ばれる過程で作られ、その際にDNA塩基は対応する塩基と交換されるが、チミン(T)の場合は例外で、RNAは[[ウラシル]](U)と交換する<ref>{{Cite web|url=https://www.genome.gov/genetics-glossary/Uracil|title=Uracil|website=Genome.gov|language=en|access-date=21 November 2019}}</ref>。これらのRNA鎖は[[翻訳 (生物学)|翻訳]]と呼ばれる過程で、{{Ill2|遺伝暗号|en|Genetic code|preserve=1}}に基づいてタンパク質の[[アミノ酸]]配列を決定する。 |
|||
[[ヌクレオチド]]は核酸の最小単位であるが、DNAはデオキシヌクレオチドの[[重合体|ポリマー]]である。核酸が構成物質として用いる[[糖]]を'''構成糖'''と呼ぶが、構成糖に[[リボース]]を用いる核酸は[[リボ核酸]] (RNA) という<ref name=Tamra38 />。ヌクレオチド分子は、糖の3’位OH基と[[リン酸]]のOH基から[[水]]が取れる形で[[ホスホジエステル結合]]を形成して結合し、これが連続的に鎖状の分子構造をとる<ref name=Tamra41-1>[[#田村(2010)|田村(2010)、p.41、I細胞生物学の基礎、5.情報高分子(2):ヌクレオチドと核酸、5-2 核酸の鎖状分子形成]]</ref>。ヌクレオチドが100個以上連結したものをポリヌクレオチド(またはポリヌクレオチド鎖)と言うが、これがDNAの1本鎖の構造である<ref name=Tamra41-1 />。DNAには方向性という概念が存在し、[[DNA複製|複製]]の際、DNAポリメラーゼは5'→3'末端の方向でDNAを合成する。RNAの[[転写 (生物学)|転写]]もこの方向性に従う<ref name=Tamra41-1 />。 |
|||
真核細胞では、DNAは[[染色体]]と呼ばれる長い構造体に組織化されている。これらの染色体は、通常の[[細胞分裂]]の前にDNA複製過程で複製され、それぞれの娘細胞に完全な染色体の集合を提供する。[[真核生物]]([[動物]]、[[植物]]、[[真菌類]]、[[原生生物]])はDNAの大部分を[[核DNA]]として[[細胞核]]内に保存し、一部を[[ミトコンドリアDNA]]として[[ミトコンドリア]]内、あるいは{{Ill2|葉緑体DNA|en|Chloroplast DNA}}として[[葉緑体]]内に保存している<ref>{{cite book | vauthors = Russell P | title= iGenetics |url= https://archive.org/details/igenetics0000russ_v6o1 |url-access= registration |publisher= Benjamin Cummings |location= New York |year= 2001 |isbn= 0-8053-4553-1}}</ref>。対照的に、[[原核生物]]([[細菌]]と[[古細菌]])はDNAを[[細胞質]]内の{{Ill2|環状染色体|en|Circular chromosome}}にのみ保存している。真核生物の染色体内では、[[ヒストン]]などの[[クロマチン]]タンパク質がDNAを小さくまとめて組織化している。これらの緻密な構造は、DNAと他のタンパク質との相互作用を導き、DNAのどの部分が転写されるかを制御するのに役立っている。{{TOC limit|3}} |
|||
二重鎖DNAでは、2本のポリヌクレオチド鎖が反平行に配向し、右巻きのらせん形態をとる('''[[二重らせん]]構造''')。2本のポリヌクレオチド鎖は、相補的な塩基 (A/T, G/C)対の[[水素結合]]を介して結合している。'''塩基の相補性'''とは、A、T、G、Cの4種の塩基うち、1種を決めればそれと水素結合で結ばれるもう1種も決まる性質である。A/T間の水素結合は2個、C/G間は3個であり、安定性が異なる。例外的に、特殊な配列が左巻きらせん構造をとる場合があり、これは[[Z-DNA|Z型DNA]]と呼ばれる。 |
|||
== 特性 == |
|||
この相補的二本鎖構造の意義は、片方を保存用(センス鎖)に残し、もう片方は、遺伝情報を必要な分だけ[[mRNA]]に伝達する転写用([[アンチセンス鎖]])とに分けることである。また、二本鎖の片方をそのまま受け継がせるため、正確なDNAの[[DNA複製|複製]]を容易に行うことができるため、遺伝情報を伝えていく上で決定的に重要である。[[DNA損傷]]の修復にも役立つ(詳しくは[[二重らせん]])。 |
|||
[[File:DNA chemical structure.svg|thumb|upright=1.35|DNAの化学構造 (点線は[[水素結合]])。4つの塩基と主鎖を構成するリン酸およびデオキシリボースを色分けしてある。二重らせんの各末端には、一方の鎖に露出した5'リン酸が、他方の鎖に露出した3'ヒドロキシ基 (-OH) がある。5'→3'方向は、左鎖では下を向き、右鎖では上を向く。]] |
|||
DNAは[[ヌクレオチド]]と呼ばれる反復単位からなる長い[[ポリマー]]である<ref>{{cite book | vauthors = Saenger W |title= Principles of Nucleic Acid Structure |publisher= Springer-Verlag |location= New York |year= 1984 |isbn= 0-387-90762-9}}</ref><ref name="Alberts">{{cite book | vauthors = Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Peter W | title = Molecular Biology of the Cell | edition = Fourth | publisher = Garland Science | year = 2002 | location = New York and London | isbn = 0-8153-3218-1 | oclc = 145080076 | url = https://www.ncbi.nlm.nih.gov/books/NBK21054/ | url-status=live | archive-url = https://web.archive.org/web/20161101022040/https://www.ncbi.nlm.nih.gov/books/NBK21054/ | archive-date = 1 November 2016 | df = dmy-all }}</ref>。DNAの構造はその長さに沿って動的であり、密なループを作ったり、他の形状に巻きつくことができる<ref>{{cite journal | vauthors = Irobalieva RN, Fogg JM, Catanese DJ, Catanese DJ, Sutthibutpong T, Chen M, Barker AK, Ludtke SJ, Harris SA, Schmid MF, Chiu W, Zechiedrich L | title = Structural diversity of supercoiled DNA | journal = Nature Communications | volume = 6 | pages = 8440 | date = October 2015 | pmid = 26455586 | pmc = 4608029 | doi = 10.1038/ncomms9440 | bibcode = 2015NatCo...6.8440I |issn=2041-1723 }}</ref>。どの[[生物種]]においても、DNAは[[水素結合]]で結合した2本のらせん状の鎖で構成されている。両方の鎖とも、同じ軸にらせん状に巻かれ、ピッチも同じで{{convert|34|Å|nm|lk=on}}である。一対の鎖の半径は{{cvt|10|Å|nm}}である<ref name="autogenerated2">{{cite journal | vauthors = Watson JD, Crick FH | title = Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid | journal = Nature | volume = 171 | issue = 4356 | pages = 737–38 | date = April 1953 | pmid = 13054692 | doi = 10.1038/171737a0 | url = http://www.nature.com/nature/dna50/watsoncrick.pdf | bibcode = 1953Natur.171..737W | s2cid = 4253007 | url-status=live | archive-url = https://web.archive.org/web/20070204110320/http://www.nature.com/nature/dna50/watsoncrick.pdf | archive-date = 4 February 2007 | df = dmy-all |issn=0028-0836 }}</ref>。別の研究によると、別の溶液中で測定した場合、DNA鎖の幅は{{cvt|22|-|26|Å|nm}}、1ヌクレオチド単位の長さは{{cvt|3.3|Å|nm}}であった<ref>{{cite journal | vauthors = Mandelkern M, Elias JG, Eden D, Crothers DM | title = The dimensions of DNA in solution | journal = Journal of Molecular Biology | volume = 152 | issue = 1 | pages = 153–61 | date = October 1981 | pmid = 7338906 | doi = 10.1016/0022-2836(81)90099-1|issn=0022-2836 }}</ref>。ほとんどのDNAの浮力密度は1.7 g/cm<sup>3</sup>である<ref>{{cite journal |last1=Arrighi |first1=Frances E. |last2=Mandel |first2=Manley |last3=Bergendahl |first3=Janet |last4=Hsu |first4=T. C. |title=Buoyant densities of DNA of mammals |journal=Biochemical Genetics |date=June 1970 |volume=4 |issue=3 |pages=367–376 |doi=10.1007/BF00485753}}</ref>。 |
|||
DNAの長さは様々である。長さの単位は、二本鎖の場合 '''bp'''(base pair: [[塩基対]])、一本鎖の場合 '''nt'''(nucleotide: 塩基、ヌクレオチド)である。 |
|||
通常、DNAは一本の鎖として存在するのではなく、一対の鎖がしっかりと結合して存在する<ref name="autogenerated2" /><ref name="berg">{{cite book | vauthors = Berg J, Tymoczko J, Stryer L | date = 2002 | title = Biochemistry | publisher = W.H. Freeman and Company | isbn = 0-7167-4955-6 }}</ref>。この2本の長い鎖は互いに巻きついて[[二重らせん]]を形成している。ヌクレオチドには、DNA分子の[[主鎖]]の一部(鎖を構成する)と[[核酸塩基]](らせん内部でもう一方のDNA鎖と相互作用する)の両方が含まれている。糖と結合した核酸塩基は[[ヌクレオシド]]({{Lang-en-short|nucleoside}})と呼ばれ、これに対し糖と1つ以上のリン酸基と結合した塩基は[[ヌクレオチド]]({{Lang-en-short|nucleotide}})と呼ばれる。(DNAのように)複数のヌクレオチドが結合した生体高分子をポリヌクレオチドと呼ぶ<ref name="IUPAC">{{cite journal | author = IUPAC-IUB Commission on Biochemical Nomenclature (CBN) | title = Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents. Recommendations 1970 | journal = The Biochemical Journal | volume = 120 | issue = 3 | pages = 449–54 | date = December 1970 | pmid = 5499957 | pmc = 1179624 | doi = 10.1042/bj1200449 | url = http://www.chem.qmul.ac.uk/iupac/misc/naabb.html | archive-url = https://web.archive.org/web/20070205191106/http://www.chem.qmul.ac.uk/iupac/misc/naabb.html | url-status=dead | archive-date = 5 February 2007 |issn=0306-3283 }}</ref>。 |
|||
=== 立体構造 === |
|||
細胞内のDNAには、[[原核生物]]や[[ミトコンドリアDNA]]のような環状と、[[真核生物]]一般に見られる線状がある<ref name=Tamra42>[[#田村(2010)|田村(2010)、p.42-45、I細胞生物学の基礎、5.情報高分子(2):ヌクレオチドと核酸、5-4 核酸の性質]]</ref>。自然界のDNAはらせん巻き数が理論値(1回転あたり10.4塩基)よりもほんの少し小さい。線状DNAには問題は無いが、環状DNAではこの差による不安定を解消するために環にねじれが生じ、これをDNAの超らせん(または負の超らせん)という<ref name=Tamra42 />。 |
|||
DNA鎖の主鎖は[[リン酸塩|リン酸]]基と[[炭水化物|糖]]基が交互に結合してできている<ref name="Ghosh">{{cite journal | vauthors = Ghosh A, Bansal M | title = A glossary of DNA structures from A to Z | journal = Acta Crystallographica Section D | volume = 59 | issue = Pt 4 | pages = 620–26 | date = April 2003 | pmid = 12657780 | doi = 10.1107/S0907444903003251|issn=0907-4449 }}</ref>。DNAの糖は[[デオキシリボース|2-デオキシリボース]]で、[[ペントース]]([[炭素]]数5、五炭糖)の一種である。糖と糖は、隣接する糖環の3番目と5番目の炭素[[原子]]間に[[ホスホジエステル結合]]を形成するリン酸基によって結合している。これらの炭素はそれぞれ、{{Ill2|方向性 (分子生物学)#3'末端|en|Directionality (molecular biology)#3′-end|label=3'末端}}(three prime end)、{{Ill2|方向性 (分子生物学)#5'末端|en|Directionality (molecular biology)#5′-end|label=5'末端}}(five prime end)と呼ばれる。プライム記号(')は、デオキシリボースが[[グリコシド結合]]を形成する塩基の炭素原子と区別するために使われる<ref name="berg" />。 |
|||
== DNAの化学的性質 == |
|||
* 2本のポリヌクレオチドを結びつける水素結合は不安定なため、沸騰水の中では離れて1本鎖になる。しかしゆっくり冷ますとポリヌクレオチドは相補性から再び結合して元に戻る。このようにDNAが1本鎖になる事を「DNAの変性」、元に復元する事をアニールという。変性が50%起こる[[温度]]はT<sub>m</sub>と記され、A/T対が多いほど低い<ref name=Tamra42 />。T<sub>m</sub>は一価の陽イオン濃度が低い場合や、水素結合を遊離させやすい[[尿素]]や[[ホルムアミド]]などが存在すると下がる<ref name=Tamra42 />。 |
|||
* 穏やかな方法で単離されたDNAは白色のフェルト状繊維で、そのナトリウム塩の水溶液は粘性が高く、[[流動複屈折]]を示す。これは熱、酸、アルカリに容易に変性し、粘度は低下し、乾燥すると粉末となり、もはや繊維状になり得ない。この変化から分子量は数百万から2万〜3万程度に下がってしまう。この化学組成はアルカリに対しては安定性が高いが、酸には弱く、容易に[[プリン (化学)|プリン]]を遊離する。この変化に伴い、[[デソキシペントース]]の[[アルデヒド基]]が遊離し、[[シッフ試薬]]を赤紫に変色させる。この[[呈色反応]]を[[フォイルゲン反応]]と呼び、これを利用して、DNAを含む核や分裂中の染色体を赤紫に着色して観察できる。 |
|||
* DNAの吸光度は塩基によって紫外線260nmを吸収極大としている<ref name=Tamra42 />。この値は、塩基が接近しているほど小さい。塩基が極めて整然と、かつ接近している二本鎖DNAよりも、不規則に配列しているときの一本鎖DNAのほうが光を吸収する力は強い。例えば、A<sub>260</sub>=1,00である二本鎖DNAと同濃度の一本鎖DNAについて、A<sub>260</sub>=1、37である(詳しくは[[DNAの巻き戻し]]参照)。 |
|||
* 変性したDNA溶液から、未変性状態のDNA状態と同じDNAを作ることができる。 |
|||
* 異なる分子種から得た一本鎖の試料を混ぜて[[再結合DNA]]を形成させる手法を[[ハイブリダイゼーション|ハイブリッド形成]]という。 |
|||
* DNAの半減期は521年、DNAの復元に必要な長さのDNA断片が残るのは約100万年前([[更新世]]の[[カラブリアン]]後半)までとそれぞれと推定されている<ref>{{Cite web|title=Mystery of DNA decay unravelled|url=https://www.murdoch.edu.au/news/articles/mystery-of-dna-decay-unravelled|website=web.archive.org|date=2020-10-17|accessdate=2020-10-17}}</ref>。 |
|||
このようにDNA鎖には通常、リボースの5'炭素に結合したリン酸基(5'ホスホリル)を持つ末端と、リボースの3'炭素に結合した遊離[[ヒドロキシ基]](3'ヒドロキシ)を持つ末端がある。糖-リン酸骨格に沿った3’と5'炭素の配向は、各DNA鎖に{{ill2|方向性 (分子生物学)|en|Directionality (molecular biology)|label=方向性}}(極性とも呼ばれる)を与える。{{Ill2|核酸二重らせん|en|Nucleic acid double helix|label=核酸の二重らせん}}では、一方の鎖のヌクレオチドの方向ともう一方の鎖のヌクレオチドの方向は反対で、[[逆平行 (生化学)|逆平行]]になっている。DNA鎖の非対称末端については、5'末端方向と3'末端方向という方向性を有し、5'末端はリン酸基を有し、3'末端はヒドロキシ基を有すると呼ばれる。DNAとRNAの大きな違いの一つは糖で、DNAの2-デオキシリボースがRNAではペントース糖の[[リボース]]に置き換えられている<ref name="berg" />。 |
|||
== 細胞内でのDNAの存在形態 == |
|||
[[細菌]]のゲノムは通常[[環状DNA]]であり、細胞内で[[核様体]]と呼ばれる構造体を形成する。[[核様体]]は[[真核細胞]]の[[核膜]]に対応する膜構造はもたない。 |
|||
[[File:DNA animation.gif|thumb|upright|DNAの部分拡大図。塩基は2本のらせん状の鎖の間に水平に配置されている ([[:ファイル:DNA orbit animated.gif|アニメーション版]])<ref>{{Cite web| vauthors = Edwards KJ, Brown DG, Spink N, Skelly JV, Neidle S |title=RCSB PDB – 1D65: Molecular structure of the B-DNA dodecamer d(CGCAAATTTGCG)2. An examination of propeller twist and minor-groove water structure at 2.2 A resolution.|url=https://www.rcsb.org/structure/1D65|access-date=2023-03-27|website=www.rcsb.org|language=en-US}}</ref>。]] |
|||
[[真核細胞]]のゲノムDNAは、[[ヒストン]]というタンパク質群と結合して、[[ヌクレオソーム]]と呼ばれる構造をつくる。[[ヌクレオソーム]]は[[クロマチン]]の基本単位であり、分裂期にはさらに折り畳まれて[[染色体]]を構築する。 |
|||
DNA二重らせんは、ヌクレオチド間の[[水素結合]]と、{{Ill2|芳香族性|en|Aromaticity|preserve=1}}核酸塩基間の塩基[[スタッキング相互作用]]という、主に2つの力によって安定化されている<ref name="Yakovchuk2006">{{cite journal | vauthors = Yakovchuk P, Protozanova E, Frank-Kamenetskii MD | title = Base-stacking and base-pairing contributions into thermal stability of the DNA double helix | journal = Nucleic Acids Research | volume = 34 | issue = 2 | pages = 564–74 | year = 2006 | pmid = 16449200 | pmc = 1360284 | doi = 10.1093/nar/gkj454 |issn=0305-1048 }}</ref>。DNAに含まれる4つの塩基は、[[アデニン]]({{mono|A}})、[[シトシン]]({{mono|C}})、[[グアニン]]({{mono|G}})、[[チミン]]({{mono|T}})である。これらの4つの塩基は、[[アデノシン一リン酸]]で示したように、糖-リン酸に結合して完全なヌクレオチドを形成する。アデニンはチミンと対になり、グアニンはシトシンと対になり、それぞれ {{mono|A-T}} と {{mono|G-C}} の[[塩基対]]を形成する<ref>{{cite book | vauthors = Tropp BE | title = Molecular Biology | edition = 4th | year = 2012 | publisher = Jones and Barlett Learning | location = Sudbury, Mass. | isbn = 978-0-7637-8663-2 }}</ref><ref>{{cite web | url = https://www.mun.ca/biology/scarr/Watson-Crick_Model.html | title = Watson-Crick Structure of DNA | year = 1953 | vauthors = Carr S | publisher = Memorial University of Newfoundland | access-date=13 July 2016 | url-status=live | archive-url = https://web.archive.org/web/20160719095721/http://www.mun.ca/biology/scarr/Watson-Crick_Model.html | archive-date = 19 July 2016 | df = dmy-all }}</ref>。 |
|||
[[古細菌]]のゲノムも[[細菌]]と同様に[[核様体]]をつくるが、[[真核細胞]]の[[ヒストン]]に似たタンパク質ももっている。 |
|||
=== 核酸塩基の分類 === |
|||
また[[細胞小器官|オルガネラ]]でも[[ミトコンドリア]]や[[葉緑体]]は独自のDNAを持つ。このことがオルガネラの由来に関する[[膜進化説]]に対する[[細胞内共生説]]の証拠であるとされている。形状は環状のものもあれば、そうでないものもある。 |
|||
核酸塩基は、5員および6員の縮合[[複素環式化合物]]である[[プリン (化学)|プリン]] {{mono|A}} と {{mono|G}} と、6員環の[[ピリミジン]] {{mono|C}} と {{mono|T}} の2種類に分類される<ref name="berg" />。第5のピリミジン核酸塩基である[[ウラシル]]({{mono|U}})は通常、RNA内でチミンの代わりを担い、その環上に[[メチル基]]を持たない点でチミンと異なる。RNAとDNAに加えて、多くの人工{{Ill2|核酸類似体|en|Nucleic acid analogue}}が核酸の特性を研究するため、あるいは[[バイオテクノロジー]]で使用するために作成されてきた<ref>{{cite journal | vauthors = Verma S, Eckstein F | title = Modified oligonucleotides: synthesis and strategy for users | journal = Annual Review of Biochemistry | volume = 67 | pages = 99–134 | year = 1998 | pmid = 9759484 |issn=0066-4154 | doi = 10.1146/annurev.biochem.67.1.99 | doi-access = free }}</ref>。 |
|||
== |
=== 非標準塩基 === |
||
DNAには修飾塩基が存在する。このうち最初に認識されたのは[[5-メチルシトシン]]で、1925年に、[[結核菌]]({{Snamei|Mycobacterium tuberculosis}})の[[ゲノム]]から発見された<ref name="Johnson1925">{{cite journal | vauthors = Johnson TB, Coghill RD | year = 1925 | title = Pyrimidines. CIII. The discovery of 5-methylcytosine in tuberculinic acid, the nucleic acid of the tubercle bacillus. | journal = Journal of the American Chemical Society | volume = 47 | pages = 2838–44 | doi=10.1021/ja01688a030|issn=0002-7863}}</ref>。細菌ウイルス([[ファージ|バクテリオファージ]])にこうした非標準塩基({{Lang-en-short|noncanonical base}})が存在する理由は、細菌に存在する[[制限酵素]]を避けるためである。この酵素系は、少なくとも部分的には、細菌をウイルス感染から保護する分子免疫系として働く<ref name="pmid27319741">{{cite journal |vauthors=Weigele P, Raleigh EA |title=Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses |journal=Chemical Reviews |volume=116 |issue=20 |pages=12655–12687 |date=October 2016 |pmid=27319741 |doi=10.1021/acs.chemrev.6b00114 |doi-access=free |issn=0009-2665 }}</ref>。より一般的な修飾DNA塩基であるシトシンとアデニンの修飾は、動植物における遺伝子発現の[[エピジェネティック]]制御(後成的調整)において、重要な役割を果たしている<ref name="pmid30619465">{{cite journal |vauthors=Kumar S, Chinnusamy V, Mohapatra T |title=Epigenetics of Modified DNA Bases: 5-Methylcytosine and Beyond |journal=Frontiers in Genetics |volume=9 |pages=640 |date=2018 |pmid=30619465 |pmc=6305559 |doi=10.3389/fgene.2018.00640 |issn=1664-8021 |doi-access=free }}</ref>。 |
|||
=== 塩基配列 === |
|||
DNAのヌクレオチドの並び方を[[塩基配列]]と言う。本来は「ヌクレオチド配列」と言うべきだが、実際の差異はそれぞれの塩基部分のみであるためこのように呼ばれる。別な呼び方では「遺伝暗号」 (genetic code) という専門的な呼称もある<ref name=Take14>[[#武村(2012)|武村(2012)、p.14-24、第1章 エピジェネティクスを理解するための基礎知識、1-1 DNAとセントラルドグマ]]</ref>。塩基配列は[[タンパク質]]の[[アミノ酸]]配列に対応しており、3つの塩基の組み合わせが20種類のアミノ酸1つずつに対応しており、mRNAに配列の情報を[[転写 (生物学)|転写]]し、細胞内の[[リボソーム]]でmRNAの3つの塩基が並ぶ情報([[コドン]])が[[翻訳 (生物学)|翻訳]]されてアミノ酸が鎖状に繋がってタンパク質が合成される。この連鎖は全生物に共通の原理であるため[[セントラルドグマ]]と呼ばれる<ref name=Take14 />。 |
|||
DNAには多くの非標準塩基が存在することが知られている<ref name="pmid28941008">{{cite journal | vauthors = Carell T, Kurz MQ, Müller M, Rossa M, Spada F | title = Non-canonical Bases in the Genome: The Regulatory Information Layer in DNA | journal = Angewandte Chemie | volume = 57 | issue = 16 | pages = 4296–4312 | date = April 2018 | pmid = 28941008 | doi = 10.1002/anie.201708228 }}</ref>。これらのほとんどは、ウラシルを含む標準塩基({{Lang-en-short|canonical base}})が修飾されたものである。 |
|||
ただし、一般的に広まっている「DNAは生命の設計図」という表現は、専門家からの批判が多い。イギリスの生物学者[[ブライアン・グッドウィン]]は「生物を遺伝子の性質に還元することはできない。生物は、それが生きている状態を特徴づけるようなダイナミックな系として理解されなければならない」<ref>{{Cite |和書 |author = 矢沢サイエンスオフィス |title = 大科学論争 |date = 1998| pages = 50|publisher = 学習研究社| series =最新科学論シリーズ |isbn=4-05-601993-2|ref = harv }}</ref>、医学博士の荻原清文は「遺伝子はあくまでもタンパク質の設計図にすぎません。すなわち、遺伝子から読み取られるタンパク質が脳細胞の形や配置のしかたを決めることはあっても、脳ができるときに1つ1つの脳細胞がお互いにどのように結合するかということまでは遺伝子は決められないのです」<ref>{{Cite |和書 |author = 荻原清文|title = 好きになる免疫学|date = 2004| pages = 152|publisher = 講談社 |isbn=4-06-153435-1|ref = harv }}</ref>、などと述べている。実際、ヒトの場合DNA中でタンパク質合成の設計にあずかる部分は全体の1.5%に過ぎない<ref name=Take14 />。 |
|||
* 修飾'''アデニン''' |
|||
** N6-カルバモイル-メチルアデニン |
|||
** N6-メチルアデニン |
|||
* 修飾'''グアニン''' |
|||
** 7-デアザグアニン |
|||
** 7-メチルグアニン |
|||
* 修飾'''シトシン''' |
|||
** N4-メチルシトシン |
|||
** 5-カルボキシルシトシン |
|||
** 5-ホルミルシトシン |
|||
** 5-グリコシルヒドロキシメチルシトシン |
|||
** 5-ヒドロキシシトシン |
|||
** 5-メチルシトシン |
|||
* 修飾'''チミジン''' |
|||
** α-グルタミルチミジン<!-- α-Glutamythymidine --> |
|||
** α-プトレシニルチミン |
|||
* '''ウラシル'''および修飾物 |
|||
** 塩基J |
|||
** ウラシル |
|||
** 5-ジヒドロキシペンタウラシル |
|||
** 5-ヒドロキシメチルデオキシウラシル |
|||
* その他 |
|||
** デオキシアルケオシン<!-- Deoxyarchaeosine --> |
|||
** 2,6-ジアミノプリン(2-アミノアデニン) |
|||
=== |
=== 主溝と副溝 === |
||
[[File:DNA-ligand-by-Abalone.png|thumb|DNAの主溝と副溝。<small>(左)</small> 副溝に侵入した[[ヘキスト染色]]色素33258が見える。<small>(右)</small> 副溝の結合部位を見る。]] |
|||
DNAの塩基配列をmRNAに転写させる遺伝子の発現は、ヒストンに巻きついて折りたたまれた状態のままでは不可能である。転写の前段階に、特定の化学物質がヒストンの[[リジン]]残基と結びついて[[アセチル化]]を起こし、元々帯びていたプラスの[[電荷]]を弱める。するとマイナスに帯電したDNAとの結びつきが弱まり<ref name=Take54>[[#武村(2012)|武村(2012)、p.54-66、第2章 エピジェネティクスを理解するための基礎知識、1-3 DNAは衣服をまとい装飾品で飾りたてる]]</ref>、ヒストンから離れて遺伝暗号部分を含む特定のDNAのらせん2本鎖がむき出しになる<ref name=Take34>[[#武村(2012)|武村(2012)、p.34-44、第1章 エピジェネティクスを理解するための基礎知識、1-3 DNAは衣服をまとい装飾品で飾りたてる]]</ref>。 |
|||
二本のらせん鎖がDNAの[[主鎖]]を形成している。もう一つの二重らせんが、その鎖と鎖の間にある空隙、あるいは溝をたどって見いだされる。これらの空隙は塩基対に隣接しており、[[結合部位]]となる可能性がある。鎖は互いに対称に配置されていないため、溝の大きさは不均等である。主溝(しゅこう)の幅は{{convert|22|Å|nm}}で、副溝(ふくこう)の幅は{{cvt|12|Å|nm}}である<ref>{{cite journal | vauthors = Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson RE | title = Crystal structure analysis of a complete turn of B-DNA | journal = Nature | volume = 287 | issue = 5784 | pages = 755–58 | date = October 1980 | pmid = 7432492 | doi = 10.1038/287755a0 | bibcode = 1980Natur.287..755W | s2cid = 4315465 }}</ref>。主溝の方が幅が広いため、塩基の端は副溝よりも主溝の方が近づきやすい。その結果、二本鎖DNAの特異的配列に結合できる[[転写因子]]などのタンパク質は、通常、主溝に露出した塩基の側面に接触する傾向がある<ref name="Pabo1984">{{cite journal | vauthors = Pabo CO, Sauer RT | title = Protein-DNA recognition | journal = Annual Review of Biochemistry | volume = 53 | pages = 293–321 | year = 1984 | pmid = 6236744 | doi = 10.1146/annurev.bi.53.070184.001453 }}</ref>。このような状況は細胞内のDNAの異常な[[コンホメーション]](立体配座)によって異なるが、主溝と副溝はDNAを通常の[[B型DNA|B型]]に巻き戻した場合に見られる幅の違いを反映するよう常に命名されている。 |
|||
DNA上のある遺伝暗号が発現を開始する際には、まずその部分のすぐそばにある「[[プロモーター]]」と呼ばれる塩基配列の転写調節領域に、「[[基本転写因子]]」とよばれる複数のタンパク質が集まって来る。この中にある活性酵素の[[ヘリカーゼ]]が作用すると、DNAの二重らせんは解離して2本の1本鎖となる。この部分に[[RNAポリメラーゼII]]という酵素がとりついて、mRNAの合成を行う<ref name=Take45>[[#武村(2012)|武村(2012)、p.45-52、第1章 エピジェネティクスを理解するための基礎知識、1-4 遺伝子の転写と高次クロマチン構造]]</ref>。遺伝暗号は2本鎖がほどけた2本の1本鎖のうちのどちらか片方に載っていることになるが、その1本鎖の相補的なもう片方の部分に別の遺伝暗号が載っている場合もある。この場合にも発現すべき遺伝暗号は基本転写因子によって制御される。 |
|||
=== 塩基対合 === |
|||
上記のような基本転写因子がプロモーター部分に集まる仕組みは、遺伝暗号とは全く離れた位置にある塩基配列の箇所によって促進され調整される。その部分は「[[エンハンサー]]」と呼ばれ、ここに「[[アクチベーター]]」と呼ばれるタンパク質が結合することが基本転写因子の活性化を制御する信号となる<ref name=Take45 />。このようにDNA上のある箇所の(タンパク質を合成するための)遺伝子としての発現は、タンパク質の合成には直接関与していない塩基配列の部分を介した複雑な機構によって支配されている。また、基本転写因子が近づくことはクロマチン構造がほどけていなければ難しいので、束の非常に固く縮こまった部分([[ヘテロクロマチン]])にある遺伝子の発現はほとんど起こらない。そのような例としては[[X染色体の不活性化]]があげられる<ref name=Take45 />。 |
|||
{{further|塩基対}} |
|||
<div class="thumb tright" style="background:#f9f9f9; border:1px solid #ccc; margin:0.5em;"> |
|||
{| border="0" border="0" cellpadding="2" cellspacing="0" style="width:230px; font-size:85%; border:1px solid #ccc; margin:0.3em;" |
|||
|- |
|||
|[[File:Base pair GC.svg|282px]] |
|||
|} |
|||
{| border="0" border="0" cellpadding="2" cellspacing="0" style="width:230px; font-size:85%; border:1px solid #ccc; margin:0.3em;" |
|||
|- |
|||
|[[File:Base pair AT.svg|282px]] |
|||
|} |
|||
<div style="border: none; width:282px;"><div class="thumbcaption"><small>(上)</small> 3つの[[水素結合]]を持つ'''{{mono|GC}}'''塩基対。<small>(下)</small> 2つの水素結合を持つ'''{{mono|AT}}'''塩基対。破線は塩基対間の非共有水素結合を示す。</div></div></div> |
|||
DNAの二重らせんでは、一方の鎖上にあるそれぞれの核酸塩基が、もう一方の鎖上のただ一種類の核酸塩基と結合する。これは[[相補性 (分子生物学)|相補的塩基対形成]]({{Lang-en-short|complementary base pairing}})と呼ばれる。プリンとピリミジンは対合して[[水素結合]]を形成し、アデニンとチミンは2本、シトシンとグアニンは3本の水素結合を形成する。このように、二重らせんを挟んで(6炭素環から6炭素環へ)2つのヌクレオチドが結合対を形成する配置は、[[ワトソン・クリック型塩基対|ワトソン・クリック塩基対]]と呼ばれる。[[GC含量|{{mono|GC}}含量]]の高いDNAは{{mono|GC}}含量の低いDNAよりも安定である。[[フーグスティーン型塩基対|フーグスティーン塩基対]]({{Lang-en-short|Hoogsteen base pair}}、6炭素環と5炭素環の水素結合)は、塩基対形成のまれな変種である<ref name="pmid23818176">{{cite journal |vauthors=Nikolova EN, Zhou H, Gottardo FL, Alvey HS, Kimsey IJ, Al-Hashimi HM |title=A historical account of Hoogsteen base-pairs in duplex DNA |journal=Biopolymers |volume=99 |issue=12 |pages=955–68 |year=2013 |pmid=23818176 |pmc=3844552 |doi=10.1002/bip.22334 }}</ref>。[[共有結合]]と異なり、水素結合は比較的簡単に切断したり再結合したりすることができる。そのため二重らせんを構成するDNAの二本鎖は、機械的な力や[[温度|高温]]によって[[線ファスナー|ファスナー]]のように引き離すことができる<ref>{{cite journal | vauthors = Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub HE | title = Mechanical stability of single DNA molecules | journal = Biophysical Journal | volume = 78 | issue = 4 | pages = 1997–2007 | date = April 2000 | pmid = 10733978 | pmc = 1300792 | doi = 10.1016/S0006-3495(00)76747-6 | bibcode = 2000BpJ....78.1997C }}</ref>。この塩基対の相補性の結果、DNAらせんの二本鎖配列のすべての情報がそれぞれの鎖に複製され、これはDNA複製に不可欠である。相補的な塩基対間のこの可逆的で特異的な相互作用は、生物におけるDNAのすべての機能にとって重要である<ref name="Alberts" />。 |
|||
== DNAとRNA == |
|||
DNAと[[リボ核酸|RNA]]はともに[[ヌクレオチド]]の重合体である[[核酸]]であるが、両者の生体内の役割は明確に異なっている。DNAは主に核の中で情報の蓄積・保存、RNAはその情報の一時的な処理を担い、DNAと比べて、必要に応じて合成・分解される頻度は顕著である。DNAとRNAの化学構造の違いの意味することの第一は「RNAはDNAに比べて不安定」である。両者の安定の度合いの違いが、{{要出典|DNAは静的でRNAは動的な印象を与える|date=2023年7月}}。 |
|||
=== |
==== ssDNAとdsDNA ==== |
||
{{Anchors|ssDNA}} |
|||
DNAとRNAの化学構造の違いの第一は、構成糖が、RNAは[[リボース]]で、リボースから2'位の[[水酸基]]で酸素が一つ少ない2'-[[デオキシリボース]]であることである。これにより、構成糖の[[立体配座]]が異なる。DNAではリボースがC2'-エンド形構造を取ることが多いが、RNAでは2'位のヒドロキシ基の存在により立体障害が生じ、リボースがC3'-エンド型構造を取る。このためDNAはB型らせん構造を取りやすく、RNAは[[A-DNA|A型らせん]]構造を取りやすくなるという違いが生じる。この結果RNAのらせん構造は主溝が深く狭くなり、副溝が浅く広くなる。 |
|||
上述したように、ほとんどのDNA分子は実際には2本のポリマー鎖であり、非共有結合によってらせん状に結合している。この二本鎖DNA構造({{Lang-en-short|double-stranded DNA}}、dsDNA)は、主に鎖内塩基[[スタッキング相互作用]]({{mono|G,C}}スタックが最も強い)によって維持されている。この2本の鎖は、融解(melting)と呼ばれる過程を経て分離し、2本の一本鎖DNA分子({{Lang-en-short|single-stranded DNA}}、ssDNA)を形成することがある。融解は、高温、低塩、高[[水素イオン指数|pH]]の条件下で起こる(低pHもDNAを融解させるが、DNAは酸-脱プリン化により不安定であるため、低pHはほとんど行われない)。 |
|||
{{see also|二重らせん}} |
|||
dsDNA型の安定性は、{{mono|GC}}含有({{mono|G,C}}塩基対の割合)だけでなく、配列(スタッキングは配列特異的であるため)および長さ(分子が長いほど安定)にも依存する。安定性はさまざまな方法で測定できる。一般的な方法は{{Ill2|核酸の熱力学|en|Nucleic acid thermodynamics|label=融解温度}}(''T<sub>m</sub>''値とも呼ばれる)であり、二本鎖分子の50%が一本鎖分子に変換される温度である。融解温度はDNAのイオン強度と濃度に依存する。したがって、{{mono|GC}}塩基対の割合とDNA二重らせんの全長の両方が、DNAの二本鎖間の結合の強さを決定する。{{mono|GC}}含量が高く長いDNAらせんは相互作用が強い鎖が多く、{{mono|AT}}含量が高く短いDNAらせんは相互作用が弱い鎖が多い<ref>{{cite journal | vauthors = Chalikian TV, Völker J, Plum GE, Breslauer KJ | title = A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 96 | issue = 14 | pages = 7853–58 | date = July 1999 | pmid = 10393911 | pmc = 22151 | doi = 10.1073/pnas.96.14.7853 | bibcode = 1999PNAS...96.7853C | doi-access = free }}</ref>。生物学では、DNA二重らせんのうち分離しやすい部分、たとえば一部の[[プロモーター]]に含まれる {{mono|TATAAT}} [[プリブノーボックス|プリブノー・ボックス]]などは、鎖を引き離しやすくするために{{mono|AT}}含量が高くなる傾向がある<ref>{{cite journal | vauthors = deHaseth PL, Helmann JD | title = Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA | journal = Molecular Microbiology | volume = 16 | issue = 5 | pages = 817–24 | date = June 1995 | pmid = 7476180 | doi = 10.1111/j.1365-2958.1995.tb02309.x | s2cid = 24479358 }}</ref>。 |
|||
1本鎖RNAでは2'位のヒドロキシ基が比較的柔軟な構造を取り反応性もあるため、DNAと比較すると不安定である。水酸基の酸素には[[孤立電子対]]が2つあるため負の電荷を帯びており、例えば、近接したリン酸のリンは周囲を[[電気陰性度]]の高い酸素原子に囲まれて水酸基の酸素原子から[[求核反応|求核攻撃]]を受けやすく、攻撃により[[ホスホジエステル結合]]が切れ、リン酸とリボースの骨格が開裂する可能性があるなどDNAと比べて不安定である。この特性から、翻訳の役割を終えたmRNAを直ちに分解することが可能になる(バクテリアでは数分、動物細胞でも数時間後には分解される)。[[安定RNA]]では1本鎖に水素結合を形成し、らせん構造となるなど、多様な二次構造、三次構造を取り、安定性を増している。<!--(安定RNAの具体的な構造および安定RNAとmRNA以外のRNAはどうなのか一言お願いします。)--> |
|||
実験室では、水素結合の半分を切断するのに必要な融解温度''T<sub>m</sub>''を求めることにより、この相互作用の強さを測定することができる。DNA二重らせん内の塩基対がすべて融解すると鎖は分離し、溶液中に完全に独立した2つの分子として存在する。これらの一本鎖DNA分子には単一の共通形状は存在しないが、いくつかのコンホメーションは他のものよりも安定している<ref>{{cite journal | vauthors = Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J | title = Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern | journal = Biochemistry | volume = 43 | issue = 51 | pages = 15996–6010 | date = December 2004 | pmid = 15609994 | doi = 10.1021/bi048221v | url = http://www.boc.uu.se/boc14www/thesis/johan2005/Paper%20V/Paper%20V.pdf | url-status=live | archive-url = https://web.archive.org/web/20070610205112/http://www.boc.uu.se/boc14www/thesis/johan2005/Paper%20V/Paper%20V.pdf | archive-date = 10 June 2007 | df = dmy-all }}</ref>。 |
|||
糖に結合している塩基にも違いがあり、DNAはA、C、G、Tであるが、RNAはTがU([[ウラシル]])に替わっている。ただし、DNA上にもUが稀に生じることがあり、また、塩基にTではなくUを用いるDNA([[U-DNA]])を持つ生物も存在する。圧倒的大多数の生物でDNAの構成塩基にUではなくTが用いられるのは、同じピリミジン塩基であるCは自然の状態でも脱アミノ化することでUに置き換わることがあるからである<ref name=Take90>[[#武村(2012)|武村(2012)、p.90-94、第3章 DNAに生じる塩基配列以外の変化、3-1 塩基の成り立ちとメチル化]]</ref>。そのため、U-DNAは頻繁に塩基配列が変化し、またそれを防ぐためには、損傷してUに変化したCと元々がUであるのと識別する必要があるという問題がある。TはUの2'にメチル基がついている構造をしている。メチル基は水素結合に係わるものの他の原子には殆ど反応しない。また、Uに比較してCからは容易に生じず、Cの損傷によって生じたUを容易に検出できる。 |
|||
以上より、DNAではUではなくTが用いられているが、UはTよりエネルギー的に有利であるため、RNAではUが用いられている。 |
|||
=== |
=== 含有量 === |
||
[[File:Human karyotype with bands and sub-bands.png|thumb|ヒトの[[核型|核型図]] (カリオグラム)。22本の{{Ill2|相同染色体|en|Homologous chromosome|preserve=1}}と、<small>(右下)</small> 女性型 (XX) と男性型 (XY) の{{Ill2|性染色体|en|Sex chromosome|preserve=1}}、<small>(左下)</small> ミトコンドリアゲノム (縮尺が左下隅にある)。それぞれの染色体対 (および{{Ill2|ヒトミトコンドリア遺伝学|en|Human mitochondrial genetics|label=ミトコンドリアゲノム}}) の左側にある青い目盛りは、その長さを数百万DNA塩基対で示している。{{further|核型}}]] |
|||
DNAとRNAの物理化学的性質について。DNAとRNAはともに[[紫外線]]である波長260nm付近に[[吸収極大]]を持ち、230nm付近に[[吸収極小]]を持つ。この吸光度はタンパク質の280nmよりもずっと大きいが、これはDNAとRNAの塩基は[[プリン (化学)|プリン]]または[[ピリミジン]]に由来するためである。ただし、二重らせん構造のDNAの場合、溶液を加熱するとその吸光度は増す(濃色効果)。これは、DNAは規則正しい二重らせん構造ゆえ、全体の吸光度は個々の塩基の吸光度の総和より小さい(淡色効果)が、熱によって水素結合が切れ、二重らせん構造が解け(核酸の変性)、個々の塩基が自由になり、独自に光を吸収するためである。また、DNAとRNAはアルカリ溶液中で挙動が異なる。RNAは弱塩基でも容易に加水分解するが、DNAは安定して存在する。 |
|||
ヒトの場合、細胞1個あたり、女性の[[二倍体]][[核ゲノム]]の総長は6.37ギガ[[塩基対]](Gbp)に及び、長さは208.23 cm、質量は6.51 pgである<ref name="pmid30813969">{{cite journal| vauthors=Piovesan A, Pelleri MC, Antonaros F, Strippoli P, Caracausi M, Vitale L| title=On the length, weight and GC content of the human genome. | journal=BMC Res Notes | year= 2019 | volume= 12 | issue= 1 | pages= 106 | pmid=30813969 | doi=10.1186/s13104-019-4137-z | pmc=6391780 | doi-access=free }}</ref>。男性の値はそれぞれ、6.27 Gbp、205.00 cm、6.41 pgである<ref name="pmid30813969" />。各DNAポリマーは、[[1番染色体 (ヒト)|1番染色体]]のように数億ものヌクレオチドを含むことがある。1番染色体は約2億2千万塩基対からなるヒト最大の[[染色体]]で、まっすぐに伸ばすと85 mmの長さになる<ref name="Gregory_2006" />。 |
|||
[[真核生物]]には、[[核DNA]]のほかに[[ミトコンドリアDNA]](mtDNA)もあり、ミトコンドリアで使われる特定のタンパク質をコードしている。mtDNAは通常、核DNAに比べて比較的小さい。たとえば、{{Ill2|ヒトミトコンドリア遺伝学|en|Human mitochondrial genetics|label=ヒトのミトコンドリアDNA}}は閉じた環状分子を形成し、それぞれの分子は16,569個<ref name="Anderson_1981">{{cite journal | vauthors = Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG | display-authors = 6 | title = Sequence and organization of the human mitochondrial genome | journal = Nature | volume = 290 | issue = 5806 | pages = 457–465 | date = April 1981 | pmid = 7219534 | doi = 10.1038/290457a0 | s2cid = 4355527 | bibcode = 1981Natur.290..457A }}</ref><ref>{{Cite web |url=http://chemistry.umeche.maine.edu/CHY431/MitoDNA.html |title=Untitled |access-date=2012-06-13 |archive-url=https://web.archive.org/web/20110813123936/http://chemistry.umeche.maine.edu/CHY431/MitoDNA.html |archive-date=2011-08-13 |url-status=dead }}</ref>のDNA塩基対を含み<ref name="Satoh1991">{{cite journal | vauthors = Satoh M, Kuroiwa T | title = Organization of multiple nucleoids and DNA molecules in mitochondria of a human cell | journal = Experimental Cell Research | volume = 196 | issue = 1 | pages = 137–140 | date = September 1991 | pmid = 1715276 | doi = 10.1016/0014-4827(91)90467-9 }}</ref>、そうした各分子には通常、ミトコンドリア遺伝子の完全な集合が含まれる。ヒトの各ミトコンドリアには、このようなmtDNA分子が平均して約5個含まれている<ref name="Satoh1991" />。各ヒト[[細胞]]は約100個のミトコンドリアを含むので、ヒト細胞あたりのmtDNA分子の総数は約500個となる<ref name="Satoh1991" />。ただし、細胞あたりのミトコンドリアの量も細胞の種類によって異なり、[[卵細胞]]には10万個のミトコンドリアが含まれることがあり、ミトコンドリアゲノム(細胞のDNAの最大90%を構成する)の最大150万コピーに相当する<ref name="pmid28721182">{{cite journal | vauthors = Zhang D, Keilty D, Zhang ZF, Chian RC | title = Mitochondria in oocyte aging: current understanding | journal = Facts, Views & Vision in ObGyn | volume = 9 | issue = 1 | pages = 29–38 | date = March 2017 | pmid = 28721182 | pmc = 5506767 | doi = }}</ref>。 |
|||
== DNAの含有量 == |
|||
細胞の分化に伴いDNAの一部が欠落する場合を除き、核内のDNA含有量は生理的条件に左右されない。すなわち一般的な体細胞<ref group="†">通常のコケは配偶体で半数体である。</ref>は[[倍数性|二倍体]]で、卵・精子等は半数体(一倍体<ref name=Tamra173>[[#田村(2010)|田村(2010)、p.173、IV細胞の増殖、20.減数分裂、20-1 減数分裂の概要]]</ref>)である。つまり卵・精子の核のDNA含有量は、その生物の体細胞のほぼ半分(厳密には、y染色体がx染色体より小さい場合、精子のDNA量はx染色体を持つ場合半分より多く、y染色体を持つ場合半分より少ない)である。DNA含有量は個々の生物で特有であり、一つの種類で、二倍体ならばどの種類の細胞であろうと値は一定である。脊椎動物の場合[[両生類]]では特に高い。[[哺乳類]]では種類ごとの含有量の差が小さく、6-7×10<sup>-12</sup>gぐらいであり、[[鳥類]]はその半分ぐらいである。 |
|||
この現象は、[[DNA複製|複製]]のためにあり、[[体細胞分裂]]ごとにDNAは2倍に増加して、2個の[[娘細胞]]に等分される。 |
|||
=== センスとアンチセンス === |
|||
DNAの合成は[[染色体]]が出現する分裂期ではなく、静止核の時期である[[間期 (細胞分裂)|間期]]の[[S期]]に行われ、分裂期は合成されたDNAを娘細胞に等分する時期に当たる。詳細は[[細胞周期]]参照。またDNAは分裂間期から分裂期までの間、転写をせず、安定な状態で、娘細胞の中に入れられる。 |
|||
{{further|センス (分子生物学)}} |
|||
ある[[DNAシークエンシング|DNA配列]]が、タンパク質に翻訳される[[メッセンジャーRNA]]のコピーと同じである場合、「センス配列」({{Lang-en-short|sense sequence}})と呼ばれる<ref>[http://www.chem.qmul.ac.uk/iubmb/newsletter/misc/DNA.html Designation of the two strands of DNA] {{Webarchive|url=https://web.archive.org/web/20080424015915/http://www.chem.qmul.ac.uk/iubmb/newsletter/misc/DNA.html |date=24 April 2008 }} JCBN/NC-IUB Newsletter 1989. Retrieved 7 May 2008</ref>。反対側の鎖の配列は「アンチセンス配列」({{Lang-en-short|antisense sequence}})と呼ばれる。センス配列とアンチセンス配列は、同じDNA鎖の異なる部分に存在することがある(すなわち、両方の鎖がセンス配列とアンチセンス配列の両方を含む)。原核生物でも真核生物でもアンチセンスRNA配列が作られるが、これらのRNAの機能は完全には解明されていない<ref>{{cite journal | vauthors = Hüttenhofer A, Schattner P, Polacek N | title = Non-coding RNAs: hope or hype? | journal = Trends in Genetics | volume = 21 | issue = 5 | pages = 289–97 | date = May 2005 | pmid = 15851066 | doi = 10.1016/j.tig.2005.03.007 }}</ref>。一つの提案は、アンチセンスRNAがRNA-RNA塩基対形成を通じて[[遺伝子発現の調節]]に関与しているというものである<ref>{{cite journal | vauthors = Munroe SH | title = Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns | journal = Journal of Cellular Biochemistry | volume = 93 | issue = 4 | pages = 664–71 | date = November 2004 | pmid = 15389973 | doi = 10.1002/jcb.20252 | s2cid = 23748148 }}</ref>。 |
|||
== DNAの合成 == |
|||
; [[デノボ合成]] |
|||
: 食物から摂った糖や[[アミノ酸]]などを元に肝臓で合成する。 |
|||
; [[サルベージ経路|サルベージ合成]] |
|||
: 食品から摂取されて分解経路に入ったヌクレオチドを再利用する。 |
|||
原核生物や真核生物のDNA配列、そして[[プラスミド]]や[[ウイルス]]ではより多くのDNA配列が、{{Ill2|オーバーラップ遺伝子|en|Overlapping gene}}({{Lang-en-short|overlapping gene}})を持つことによってセンス鎖とアンチセンス鎖の区別をあいまいにしている<ref>{{cite journal | vauthors = Makalowska I, Lin CF, Makalowski W | title = Overlapping genes in vertebrate genomes | journal = Computational Biology and Chemistry | volume = 29 | issue = 1 | pages = 1–12 | date = February 2005 | pmid = 15680581 | doi = 10.1016/j.compbiolchem.2004.12.006 }}</ref>。このような場合、DNA配列の中には、一方の鎖に沿って読まれると一方のタンパク質をコードし、もう一方の鎖に沿って逆方向に読まれるともう一方のタンパク質をコードするという、二重の役割を果たすものがある。[[細菌]]では、この重畳が遺伝子転写の調節に関与している可能性がある<ref>{{cite journal | vauthors = Johnson ZI, Chisholm SW | title = Properties of overlapping genes are conserved across microbial genomes | journal = Genome Research | volume = 14 | issue = 11 | pages = 2268–72 | date = November 2004 | pmid = 15520290 | pmc = 525685 | doi = 10.1101/gr.2433104 }}</ref>。一方、ウイルスでは、オーバーラップ遺伝子によって、小さなウイルスゲノム内にコードできる情報量を増加させる<ref>{{cite journal | vauthors = Lamb RA, Horvath CM | title = Diversity of coding strategies in influenza viruses | journal = Trends in Genetics | volume = 7 | issue = 8 | pages = 261–66 | date = August 1991 | pmid = 1771674 | doi = 10.1016/0168-9525(91)90326-L | pmc = 7173306 }}</ref>。 |
|||
=== DNAの材料 === |
|||
[[ヌクレオチド]]及びその結合体である[[ポリヌクレオチド]]、DNA、[[リボ核酸|RNA]]は生物を原料とするほとんどの食品に微量含まれており、[[魚]]の[[白子 (精巣)|白子]]や[[動物]]の[[睾丸]]などでは含有率が高い。DNAを摂取すると、体内でいったんヌクレオチドに分解された後、ヌクレオシド3リン酸となり、RNA、DNAを効率的に合成する材料となる。 |
|||
=== スーパーコイル === |
|||
工業的に効率的に分離するための原料として[[サケ]]の[[白子 (精巣)|白子]]や[[ホタテガイ]]の[[生殖巣]]などが利用されている。 |
|||
{{further|DNA超らせん}} |
|||
DNAは、[[DNAスーパーコイル]]({{Lang-en-short|DNA supercoiling}}、DNA超らせん)と呼ばれる過程で、[[ロープ]]のようにねじれることがある。DNAが「弛緩した」状態では、鎖は通常10.4塩基対ごとに二重らせんの軸の周りを一周するが、DNAがねじれると鎖はよりきつく、あるいはより緩く巻かれる<ref>{{cite journal | vauthors = Benham CJ, Mielke SP | s2cid = 1427671 | title = DNA mechanics | journal = Annual Review of Biomedical Engineering | volume = 7 | pages = 21–53 | year = 2005 | pmid = 16004565 | doi = 10.1146/annurev.bioeng.6.062403.132016 | url = http://pdfs.semanticscholar.org/ab63/d57290ebf9bc3536fd3f2257a2b509076fc1.pdf | archive-url = https://web.archive.org/web/20190301225243/http://pdfs.semanticscholar.org/ab63/d57290ebf9bc3536fd3f2257a2b509076fc1.pdf | url-status = dead | archive-date = 1 March 2019 }}</ref>。DNAがらせんの方向にねじれている場合、これは正のスーパーコイルと呼ばれ、塩基同士はより近くに配置される。もし反対方向にねじれているなら、これは負のスーパーコイルと呼ばれ、塩基同士はより離れやすくなる。自然界では、ほとんどのDNAは、[[DNAトポイソメラーゼ|トポイソメラーゼ]]と呼ばれる[[酵素]]によって導入される、わずかに負のスーパーコイルを持っている<ref name="Champoux">{{cite journal | vauthors = Champoux JJ | s2cid = 18144189 | title = DNA topoisomerases: structure, function, and mechanism | journal = Annual Review of Biochemistry | volume = 70 | pages = 369–413 | year = 2001 | pmid = 11395412 | doi = 10.1146/annurev.biochem.70.1.369 | url = http://pdfs.semanticscholar.org/983e/e70eabbeccac71bf6a634d1d538225c64c71.pdf }}</ref>。これらの酵素は、[[転写 (生物学)|転写]]や[[DNA複製]]などの過程でDNA鎖に生じるねじれ応力を緩和するためにも必要である<ref name="Wang">{{cite journal | vauthors = Wang JC | title = Cellular roles of DNA topoisomerases: a molecular perspective | journal = Nature Reviews Molecular Cell Biology | volume = 3 | issue = 6 | pages = 430–40 | date = June 2002 | pmid = 12042765 | doi = 10.1038/nrm831 | s2cid = 205496065 }}</ref>。 |
|||
== 遺伝情報の担い手としてのDNA == |
|||
[[画像:Dna-split.png|thumb|right|DNAの複製を描いた図。]] |
|||
{{Main|DNA複製}} |
|||
全ての生物で、細胞分裂の際の[[母細胞]]から[[娘細胞]]への遺伝情報の受け渡しは、DNAの複製によって行われる。DNA の複製は[[DNAポリメラーゼ]]によって行われる。 |
|||
=== 代替DNA構造 === |
|||
DNAが親から子へ伝わるときにDNAに[[変異]]が起こり、新しい[[形質]]が付加されることがあり、これが種の保存にとって重要になることがある。 |
|||
{{further|{{ill2|核酸の分子構造: デオキシリボ核酸の構造|en|Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid}}|{{ill2|DNAの分子モデル|en|Molecular models of DNA}}|核酸構造}} |
|||
[[File:Dnaconformations.png|thumb|right|[[A型DNA|A-DNA]]、[[B型DNA|B-DNA]]、[[Z型DNA|Z-DNA]]の構造 (左から右へ)]] |
|||
DNAは、[[A型DNA|A-DNA]](A型DNA)、[[B型DNA|B-DNA]](B型DNA)、[[Z型DNA|Z-DNA]](Z型DNA)などの多くの起こりうる[[コンホメーション]]で存在するが、機能的な生物で直接観察されているのはB-DNAとZ-DNAに限られる<ref name="Ghosh" />。DNAが取るコンホメーションは、水和レベル、DNA配列、スーパーコイルの量と方向、塩基の化学修飾、金属[[イオン]]の種類と濃度、溶液中の[[ポリアミン]]の有無に依存する<ref>{{cite journal | vauthors = Basu HS, Feuerstein BG, Zarling DA, Shafer RH, Marton LJ | title = Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies | journal = Journal of Biomolecular Structure & Dynamics | volume = 6 | issue = 2 | pages = 299–309 | date = October 1988 | pmid = 2482766 | doi = 10.1080/07391102.1988.10507714 }}</ref>。 |
|||
[[細菌]]など[[細胞分裂|分裂]]によって増殖する生物は、条件が良ければ指数的に増殖する。その際、複製のミスによって[[薬剤耐性]]のような新たな形質を獲得し、それまで生息できなかった条件で生き残ることができるようになる。 |
|||
A-DNA、およびB-DNAの{{Ill2|X線散乱法|en|X-ray scattering technique|label=X線回折パターン}}について最初に発表された報告では、[[パターソン関数]]に基づく解析が使用され、DNAの配向繊維に限られた構造情報しか得られなかった<ref>*{{cite journal |vauthors=Franklin RE, Gosling RG |title=The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content |journal=Acta Crystallogr |volume=6 |issue=8–9 |pages=673–77 |date=6 March 1953 |doi=10.1107/S0365110X53001939 |url=http://journals.iucr.org/q/issues/1953/08-09/00/a00979/a00979.pdf |url-status=live |archive-url=https://web.archive.org/web/20160109043915/http://journals.iucr.org/q/issues/1953/08-09/00/a00979/a00979.pdf |archive-date=9 January 2016 |doi-access=free }} |
|||
[[有性生殖]]をする生物において、DNAは[[減数分裂]]時の[[染色体]]の組み換えや、[[配偶子]]の染色体の組み合わせにより、次世代の形質に多様性が生まれる。 |
|||
*{{cite journal |vauthors=Franklin RE, Gosling RG |title=The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function |journal=Acta Crystallogr |volume=6 |issue=8–9 |pages=678–85 |year=1953|doi=10.1107/S0365110X53001940|url=http://journals.iucr.org/q/issues/1953/08-09/00/a00980/a00980.pdf |archive-url=https://web.archive.org/web/20170629084321/http://journals.iucr.org/q/issues/1953/08-09/00/a00980/a00980.pdf |archive-date=2017-06-29 |url-status=live |doi-access=free }}</ref><ref name="NatFranGos">{{cite journal | vauthors = Franklin RE, Gosling RG | title = Molecular configuration in sodium thymonucleate | journal = Nature | volume = 171 | issue = 4356 | pages = 740–41 | date = April 1953 | pmid = 13054694 | doi = 10.1038/171740a0 | url = http://www.nature.com/nature/dna50/franklingosling.pdf | bibcode = 1953Natur.171..740F | s2cid = 4268222 | url-status=live | archive-url = https://web.archive.org/web/20110103160712/http://www.nature.com/nature/dna50/franklingosling.pdf | archive-date = 3 January 2011 | df = dmy-all }}</ref>。1953年、ウィルキンスらによって、高水和DNA繊維の ''[[in vivo]]''(生体内'')''B-DNA X線回折散乱パターンについて、[[ベッセル関数]]の2乗という観点から別の解析法が提案された<ref name="NatWilk">{{cite journal | vauthors = Wilkins MH, Stokes AR, Wilson HR | title = Molecular structure of deoxypentose nucleic acids | journal = Nature | volume = 171 | issue = 4356 | pages = 738–40 | date = April 1953 | pmid = 13054693 | doi = 10.1038/171738a0 | url = http://www.nature.com/nature/dna50/wilkins.pdf | bibcode = 1953Natur.171..738W | s2cid = 4280080 | url-status=live | archive-url = https://web.archive.org/web/20110513234223/http://www.nature.com/nature/dna50/wilkins.pdf | archive-date = 13 May 2011 | df = dmy-all }}</ref>。同じジャーナルで、[[ジェームズ・ワトソン]]と[[フランシス・クリック]]が、DNAのX線回折パターンの[[分子モデリング]]解析を発表し、その構造が二重らせんであることを提案した<ref name="autogenerated2" />。 |
|||
B-DNAは細胞内で見られる条件下で最もありふれているが<ref>{{cite journal | vauthors = Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL | title = Polymorphism of DNA double helices | journal = Journal of Molecular Biology | volume = 143 | issue = 1 | pages = 49–72 | date = October 1980 | pmid = 7441761 | doi = 10.1016/0022-2836(80)90124-2 }}</ref>、これは明確に定義されたコンホメーションではなく、細胞内で見られる高水和レベルで生じる関連するDNAコンホメーションの一群である<ref>{{cite journal|vauthors=Baianu IC|s2cid=189888972|year=1980|title=Structural Order and Partial Disorder in Biological systems|url=http://cogprints.org/3822/|journal=Bull. Math. Biol.|volume=42|issue=4|pages=137–41|doi=10.1007/BF02462372}}</ref>。それらに対応するX線回折とX線散乱のパターンは、かなりの程度の無秩序を伴う分子{{Ill2|準結晶性|en|Paracrystallinity|label=準結晶}}に特徴的である<ref>{{cite book | vauthors = Hosemann R, Bagchi RN | title = Direct analysis of diffraction by matter | publisher = North-Holland Publishers | location = Amsterdam – New York | year = 1962 }}</ref><ref>{{cite journal|vauthors=Baianu IC|title=X-ray scattering by partially disordered membrane systems|journal=Acta Crystallogr A|volume=34|issue=5|pages=751–53|year=1978|doi=10.1107/S0567739478001540|bibcode=1978AcCrA..34..751B|url=http://journals.iucr.org/a/issues/1978/05/00/a15615/a15615.pdf|access-date=29 August 2019|archive-date=14 March 2020|archive-url=https://web.archive.org/web/20200314050140/http://journals.iucr.org/a/issues/1978/05/00/a15615/a15615.pdf|url-status=dead}}</ref>。 |
|||
== 3本鎖DNAの存在について == |
|||
これまで2本鎖、もしくは1本鎖のみと考えられていたDNAであるが、近年3本鎖DNAの存在が示唆されてきている<ref name=Tamra41-2>[[#田村(2010)|田村(2010)、p.41、I細胞生物学の基礎、5.情報高分子(2):ヌクレオチドと核酸、5-3 DNAの二重鎖と塩基対の相補性]]</ref><ref>reviewed in Right 2004; myong et al., 2006</ref>。 |
|||
B-DNAと比較すると、A-DNAは浅く広い副溝と狭く深い主溝を持つ、より幅の広い[[螺旋|右巻き]]らせんである。A型は、非生理学的条件下では、部分的に脱水したDNA試料中に生じるが、細胞内ではDNA鎖とRNA鎖の混成ペアリング<!-- hybrid pairings -->や、酵素-DNA複合体に生じることがある<ref>{{cite journal | vauthors = Wahl MC, Sundaralingam M | title = Crystal structures of A-DNA duplexes | journal = Biopolymers | volume = 44 | issue = 1 | pages = 45–63 | year = 1997 | pmid = 9097733 | doi = 10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-# }}</ref><ref>{{cite journal | vauthors = Lu XJ, Shakked Z, Olson WK | title = A-form conformational motifs in ligand-bound DNA structures | journal = Journal of Molecular Biology | volume = 300 | issue = 4 | pages = 819–40 | date = July 2000 | pmid = 10891271 | doi = 10.1006/jmbi.2000.3690 }}</ref>。塩基が[[メチル化]]で化学修飾されたDNAセグメントは、より大きなコンホメーション変化を起こし、[[Z型DNA|Z-DNA]]を取ることがある。この場合、鎖はらせん軸を中心に左巻きのらせんを描き、より一般的なB型とは正反対となる<ref>{{cite journal | vauthors = Rothenburg S, Koch-Nolte F, Haag F | title = DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles | journal = Immunological Reviews | volume = 184 | pages = 286–98 | date = December 2001 | pmid = 12086319 | doi = 10.1034/j.1600-065x.2001.1840125.x | s2cid = 20589136 }}</ref>。このような特異な構造は、特異的なZ-DNA結合タンパク質によって認識され、転写制御に関与している可能性がある<ref>{{cite journal | vauthors = Oh DB, Kim YG, Rich A | title = Z-DNA-binding proteins can act as potent effectors of gene expression in vivo | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 99 | issue = 26 | pages = 16666–71 | date = December 2002 | pmid = 12486233 | pmc = 139201 | doi = 10.1073/pnas.262672699 | bibcode = 2002PNAS...9916666O | doi-access = free }}</ref>。 |
|||
通常、DNAは[[真核生物]]の細胞内では2本鎖の状態で存在している。そのDNAのGC含量にもよるが、DNAは60℃前後で水素結合が壊れて1本鎖となる([[Tm値]])。逆に温度が下がり、0℃を下回るあたり(Bm値。若干の幅がある)で[[細胞質]]内の[[リン酸]]基を中心に3つの塩基が同じ高さに来ることがある。 |
|||
=== 代替DNA化学 === |
|||
この場合、事実上3本のDNA鎖が並列に存在することとなり、DNAは3本鎖となる。リン酸を必要とするため、単純なDNA溶液のみでの実験を行っても、[[in vitro]](試験管内などの人工的に構成された条件下)での証明が難しい。今後はより再現性を高めた研究が進むものと期待されている。 |
|||
[[宇宙生物学|宇宙生物学者]]たちは長年にわたり、現在知られている生命とは根本的に異なる生化学的および分子学的プロセスを用いる、地球上の微生物生物圏({{Ill2|影の生物圏|en|Shadow biosphere}})の存在を提案してきた。その提案の一つは、[[ヒ素の生化学|DNA中のリンの代わりにヒ素を使用する]]生命体の存在であった。2010年、[[GFAJ-1]]という[[細菌]]におけるその可能性が報告されたが<ref name="arsenic extremophile">{{cite news | vauthors = Palmer J |title=Arsenic-loving bacteria may help in hunt for alien life |date=2 December 2010 |url=https://www.bbc.co.uk/news/science-environment-11886943 |work=BBC News |access-date=2 December 2010 |url-status=live |archive-url=https://web.archive.org/web/20101203045804/http://www.bbc.co.uk/news/science-environment-11886943 |archive-date=3 December 2010 }}</ref><ref name="Space">{{cite news | vauthors = Bortman H |title=Arsenic-Eating Bacteria Opens New Possibilities for Alien Life |date=2 December 2010 |url=http://www.space.com/scienceastronomy/arsenic-bacteria-alien-life-101202.html |website=Space.com |access-date=2 December 2010 |url-status=live |archive-url=https://web.archive.org/web/20101204235915/http://www.space.com/scienceastronomy/arsenic-bacteria-alien-life-101202.html |archive-date=4 December 2010 }}</ref>、この研究は論争を呼び<ref name="Space" /><ref>{{cite journal | vauthors = Katsnelson A |title=Arsenic-eating microbe may redefine chemistry of life |date=2 December 2010 |url=http://www.nature.com/news/2010/101202/full/news.2010.645.html |journal=Nature News |doi=10.1038/news.2010.645 |url-status=live |archive-url=https://web.archive.org/web/20120212155007/http://www.nature.com/news/2010/101202/full/news.2010.645.html |archive-date=12 February 2012 }}</ref>、細菌がDNA骨格や他の生体分子へのヒ素の取り込みを積極的に妨げていることを示唆する証拠が示された<ref name="Nature">{{cite journal | vauthors = Cressey D |s2cid=87341731 |title='Arsenic-life' Bacterium Prefers Phosphorus after all |date=3 October 2012 |journal=Nature News |doi=10.1038/nature.2012.11520}}</ref>。 |
|||
=== 四重鎖構造 === |
|||
3本鎖となったDNAにおいても、そのねじれは2本鎖の場合と変わらず、約10.5塩基ごとに1周である。3本鎖になることにより、2本鎖の場合のDNAの一次構造の保持への負担はより軽くなると思われがちである。しかし、実際に保持エネルギーを計測すると3本鎖DNAの方がエネルギーが大きく、遥かに不安定であることが実験的に証明されている<ref>reviewed in Documentation 2006; leime et al., 2007</ref>。 |
|||
{{further|グアニン四重鎖}} |
|||
[[File:Parallel telomere quadruple.png|thumb|right|[[テロメア]]の反復によって形成されたDNA[[グアニン四重鎖|四重鎖]]。DNA骨格のループ構造は、典型的なDNAらせんとは大きく異なる。中央の緑色の球はカリウムイオンを表す<ref>{{Cite web|title=Structure and packing of human telomeric DNA|url=http://ndbserver.rutgers.edu/service/ndb/atlas/summary?searchTarget=UD0017|access-date=2023-05-18|website=ndbserver.rutgers.edu}}</ref>。]] |
|||
{{Ill2|線状染色体|en|Linear chromosome}}の末端には、[[テロメア]]と呼ばれる特殊なDNA領域がある。テロメアの主な役割は、通常DNAを複製する酵素は染色体の3'末端の端部をコピーできないため、細胞が[[テロメラーゼ]]という酵素を使用して染色体末端を複製できるようにすることである<ref name="Greider">{{cite journal | vauthors = Greider CW, Blackburn EH | title = Identification of a specific telomere terminal transferase activity in Tetrahymena extracts | journal = Cell | volume = 43 | issue = 2 Pt 1 | pages = 405–13 | date = December 1985 | pmid = 3907856 | doi = 10.1016/0092-8674(85)90170-9 | doi-access = free }}</ref>。これらの特殊な染色体キャップはDNA末端を保護し、細胞の[[DNA修復]]系がそれらを修正すべき損傷として扱うことを防ぐのにも役立つ<ref name="Nugent">{{cite journal | vauthors = Nugent CI, Lundblad V | title = The telomerase reverse transcriptase: components and regulation | journal = Genes & Development | volume = 12 | issue = 8 | pages = 1073–85 | date = April 1998 | pmid = 9553037 | doi = 10.1101/gad.12.8.1073 | doi-access = free }}</ref>。[[ヒトの細胞の一覧|ヒト細胞]]ではテロメアは通常、単純な {{Mono|TTAGGG}} 配列が数千回繰り返された一本鎖DNAである<ref>{{cite journal | vauthors = Wright WE, Tesmer VM, Huffman KE, Levene SD, Shay JW | title = Normal human chromosomes have long G-rich telomeric overhangs at one end | journal = Genes & Development | volume = 11 | issue = 21 | pages = 2801–09 | date = November 1997 | pmid = 9353250 | pmc = 316649 | doi = 10.1101/gad.11.21.2801 }}</ref>。 |
|||
なお、一部の[[担子菌類]]では、自然界で正常に存在している状態で3本鎖のDNAを有するものが見つかっている。これらのDNAが転写・複製される場合、3本が同時にほどけるのではなく、1本ずつ順番にほどけて複製される。そのため、これらの生物がDNA複製を行う際生体内のDNA量を計測すると、ある1点で急激に増加するのではなく段階的に増加していることがわかる。 |
|||
これらのグアニンに富んだ配列は他のDNA分子に見られる通常の塩基対ではなく、4塩基単位が積み重なった構造を形成することによって染色体末端を安定化させる可能性がある。ここでは4つのグアニン塩基が、{{Ill2|グアニンテトラッド|en|Guanine tetrad}}({{Lang-en-short|guanine tetrad}})と呼ばれる平面を形成している。そして、これらの4塩基単位の平面が積み重なり、安定した[[グアニン四重鎖]]構造を形成する<ref name="Burge">{{cite journal | vauthors = Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S | title = Quadruplex DNA: sequence, topology and structure | journal = Nucleic Acids Research | volume = 34 | issue = 19 | pages = 5402–15 | year = 2006 | pmid = 17012276 | pmc = 1636468 | doi = 10.1093/nar/gkl655 }}</ref>。これらの構造は、塩基の端同士の水素結合と、各4塩基単位の中心にある金属イオンの[[キレート|キレート化]]によって安定化している<ref>{{cite journal | vauthors = Parkinson GN, Lee MP, Neidle S | title = Crystal structure of parallel quadruplexes from human telomeric DNA | journal = Nature | volume = 417 | issue = 6891 | pages = 876–80 | date = June 2002 | pmid = 12050675 | doi = 10.1038/nature755 | bibcode = 2002Natur.417..876P | s2cid = 4422211 }}</ref>。他の構造を形成することも可能で、中央にある4塩基の集まりは、塩基の周囲に折りたたまれた単鎖か、それぞれが中央の構造に1塩基ずつ寄与するいくつかの異なる平行鎖のいずれかから形成される。 |
|||
== DNAの利用 == |
|||
; [[DNA鑑定]] |
|||
: DNAの[[反復領域]]の違いをもとに、血液その他から人物の特定などを行う。犯罪[[捜査]]や[[親子]]鑑定に利用される。 |
|||
; [[医療]] |
|||
: [[遺伝子治療]]や[[オーダメイド医療]]という、一人ひとりの個性に合った治療が可能になる。 |
|||
; [[工業]] |
|||
: DNAの[[二重らせん構造]]を使って、微細な有機分子を捉える[[フィルター]]{{要曖昧さ回避|date=2023年7月}}が開発されている{{要出典|date=2023年7月}}。 |
|||
;[[環境DNA]]調査 |
|||
: [[海]]・[[湖沼]]・[[河川]]の水や[[土壌]]に含まれる動物の[[糞]]などに由来するDNAを分析することで、その水域・地域における希少生物などの有無を確認したり、生息数を推計したりできる<ref>[https://www.nikkei.com/article/DGKKZO28836920Q8A330C1TJM000/ 環境DNAとは]『日本経済新聞』朝刊2018年4月2日(科学技術面)「水中のDNAで生息調査」用語解説</ref><ref>[http://www.yomiuri.co.jp/osaka/feature/CO032251/20180202-OYTAT50000.html 【平成時代 DNAの30年】読み解く(5)魚の種類1杯の水で]『読売新聞』朝刊2018年2月2日</ref>。 |
|||
; [[ナノテクノロジー]] |
|||
: 生物学的な役割とは直接関係しないナノ構造材として用いられることもある([[DNAナノテクノロジー]])。 |
|||
このような積層構造に加えて、テロメアはテロメアループ(Tループ)と呼ばれる大きなループ構造も形成する。ここでは、一本鎖DNAがテロメア結合タンパク質によって安定化された大きな円を描くように巻きついている<ref>{{cite journal | vauthors = Griffith JD, Comeau L, Rosenfield S, Stansel RM, Bianchi A, Moss H, de Lange T | s2cid = 721901 | title = Mammalian telomeres end in a large duplex loop | journal = Cell | volume = 97 | issue = 4 | pages = 503–14 | date = May 1999 | pmid = 10338214 | doi = 10.1016/S0092-8674(00)80760-6 | citeseerx = 10.1.1.335.2649 }}</ref>。Tループの最先端では一本鎖テロメアDNAがテロメア鎖によって二本鎖DNAの領域に保持され、二重らせんDNAを分離し、二本鎖の一方と塩基対を形成する。この{{Ill2|三重鎖DNA|en|Triple-stranded DNA|label=三重鎖構造|preserve=1}}は、置換ループあるいは[[Dループ]]と呼ばれる<ref name="Burge" />。 |
|||
== DNA小史 == |
|||
* [[1869年]]: [[フリードリッヒ・ミーシェル|フリードリッヒ・ミーシャー]]([[スイス]])が[[白血球]]の核中からリン酸塩からなる化学物質(のちにDNAとされる)の抽出に成功。1871年にヌクレインという名で発表したが、彼はその役割を細胞内におけるリンの貯蔵と考えていた(後にリヒャルト・アルトマン(ドイツ)によってヌクレインは核酸と改称される)。 |
|||
<div class="thumb tright" style="background:#f9f9f9; border:1px solid #ccc; margin:0.5em;"> |
|||
* [[1885年]]: A.コッセルがアデニンを発見。86年にグアニン、93年にチミン、94年にシトシンも発見。 |
|||
{| border="0" cellpadding="2" cellspacing="0" style="width:200px; font-size:85%; border:1px solid #ccc; margin:0.3em;" |
|||
* [[1909年]]: [[フィーバス・レヴィーン]]がリボースを構成糖とする核酸・RNAを発見。 |
|||
|[[File:Branch-dna-single.svg|95px]] |
|||
* [[1929年]]: 上述のフィーバス・レヴィーンがDNAの構成糖はデオキシリボースで、核酸にはDNAとRNAの2種類あることを発見。 |
|||
|[[File:Branch-DNA-multiple.svg|95px]] |
|||
* [[1944年]]: [[オズワルド・アベリー]]らによって[[肺炎双球菌]]を用いて DNA が[[形質転換]]の原因物質であることが証明される。これはDNAが遺伝子本体であることを強く示唆したものであると理解された(当時、遺伝子の正体がDNAかタンパク質か論争が起こっていた)。 |
|||
|- |
|||
* [[1952年]]: A.D.ハーシーとM.チェイスは、[[バクテリオファージ]]を用いて、DNAが遺伝物質であることを直接に確認([[ハーシーとチェイスの実験]])。DNA が遺伝物質であることが決定的になる。 |
|||
| align="center" |単一分岐 |
|||
* [[1953年]]: [[ジェームズ・ワトソン|J.ワトソン]]、[[フランシス・クリック|F.クリック]]が[[ロザリンド・フランクリン]]や[[モーリス・ウィルキンス]]の研究データの提供によって DNA の二重らせん構造を明らかにした。 |
|||
| align="center" |多重分岐 |
|||
* [[1956年]]: A.コーンバーグによってDNAポリメラーゼが発見される。コーンバーグはDNAポリメラーゼの精製にも成功している。 |
|||
|} |
|||
* [[1957年]]: M.メセルソンとF.W.スタールによって DNA の[[半保存的複製]]が明らかにされる。 |
|||
<div style="border: none; width:200px;font-size: 90%;"><div class="thumbcaption">{{Ill2|分枝DNA法|en|Branched DNA assay|label=分枝DNA}}は、複数の枝を含むネットワークを形成することがある</div></div></div> |
|||
* [[1962年]]: J.ワトソン、Fクリックとモーリス・ウィルキンスが、ノーベル賞を受賞した。ロザリンド・フランクリンは、その前に亡くなった。 |
|||
* [[1967年]]: [[岡崎令治]]らによって[[岡崎フラグメント]]が発見される。 |
|||
=== 分岐DNA === |
|||
* [[1970年]]: H.スミスによって[[制限酵素]] [[HindIII]]が分離される。 |
|||
{{further|{{ill2|分岐DNA法|en|Branched DNA}}|DNAナノテクノロジー}} |
|||
* [[1971年]]: ポール・バーグによって史上初の組み替えDNA実験を行った。発ガンウイルスの1種SV40のDNAを、ある種のバクテリオファージに組み替えることに成功。その後、実験室から漏れ出した大腸菌の危険性を指摘され、4年後アシロマ会議を主催する。 |
|||
* [[1975年]]: 上述のバーグの呼びかけで[[アシロマ会議]]開催。{{Main|遺伝子#歴史|遺伝子}} |
|||
DNAでは、相補的であるべき二本鎖DNAの末端部に相補的でない領域が存在すると「{{Ill2|粘着末端および平滑末端|en|Sticky and blunt ends|label=ほつれ}}」を生じる。しかし第三のDNA鎖が導入され、既存の二本鎖のほつれ領域と混成できる隣接領域を含む場合、分岐DNA({{Lang-en-short|branched DNA}})が生じる可能性がある。分岐DNAの最も単純な例は3本のDNA鎖のみであるが、さらなる鎖と複数の分岐を含む複合体も可能である<ref>{{cite journal | vauthors = Seeman NC | title = DNA enables nanoscale control of the structure of matter | journal = Quarterly Reviews of Biophysics | volume = 38 | issue = 4 | pages = 363–71 | date = November 2005 | pmid = 16515737 | pmc = 3478329 | doi = 10.1017/S0033583505004087 }}</ref>。分岐DNAは、幾何学的形状を構築するために[[ナノテクノロジー]]で使用することができる。以下の[[DNA#技術における用途|技術における用途]]の節も参照のこと。 |
|||
=== 人工塩基 === |
|||
{{Main|{{ill2|核酸アナログ|en|Nucleic acid analogue}}}} |
|||
いくつかの人工塩基が合成され、[[ハチモジDNA]]({{Lang-en-short|Hachimoji DNA}})と呼ばれる8塩基の{{Ill2|核酸アナログ|en|Nucleic acid analogue}}に組み込むことに成功した。S、B、P、Zと命名されたこれらの人工塩基は、予測可能な方法で互いに結合し(S-BとP-Z)、DNAの二重らせん構造を維持し、RNAに転写することができる。これらの人工塩基の存在は、地球上で進化してきた4つの天然の核酸塩基には特別なものは何もないことを示すものと考えられる<ref>{{cite journal | vauthors = Warren M |title=Four new DNA letters double life's alphabet | journal = Nature |date=21 February 2019 | doi = 10.1038/d41586-019-00650-8 | pmid = 30809059 | volume=566 | issue = 7745 | page=436| doi-access = free | bibcode = 2019Natur.566..436W }}</ref><ref>{{cite journal| vauthors = Hoshika S, Leal NA, Kim MJ, Kim MS, Karalkar NB, Kim HJ, Bates AM, Watkins NE, SantaLucia HA, Meyer AJ, DasGupta S, Piccirilli JA, Ellington AD, SantaLucia J, Georgiadis MM, Benner SA | display-authors = 6 |title=Hachimoji DNA and RNA: A genetic system with eight building blocks (paywall)|journal=[[:en:Science (journal)|Science]] |volume=363 |issue=6429 |pages=884–887 |date=22 February 2019 | doi = 10.1126/science.aat0971 | pmid = 30792304 | pmc=6413494 | bibcode=2019Sci...363..884H}}</ref>。一方、DNAは[[リボ核酸|RNA]]と密接な関係にあり、RNAはDNAの転写産物としてだけではなく、細胞内で多くの仕事をこなす[[分子機械]]でもある。そのためには、RNAは適切な構造に折り畳まれなければならない。すべての可能な立体構造を作るためには、対応するRNAに少なくとも4つの塩基が必要であることが示されている<ref>{{cite journal | vauthors = Burghardt B, Hartmann AK | title = RNA secondary structure design | journal = Physical Review E | volume = 75 | issue = 2 | pages = 021920 | date = February 2007 | doi = 10.1103/PhysRevE.75.021920 | pmid = 17358380 | url = https://link.aps.org/doi/10.1103/PhysRevE.75.021920| arxiv = physics/0609135 | bibcode = 2007PhRvE..75b1920B | s2cid = 17574854 }}</ref>。一方、それ以上の数も可能であるが、これは{{Ill2|最小努力の原理|en|Principle of least effort|label=最小努力の自然原理}}に反することになる。 |
|||
===酸性度=== |
|||
DNAの[[リン酸基]]はリン酸と同様の[[酸性]]特性を与えることから、{{Ill2|強酸度|en|Acid strength|label=強酸}}とみなすことができる。DNAは、通常の細胞内pHでは完全にイオン化し、[[陽子]]を放出してリン酸基は負電荷を帯びる。これらの負電荷は、DNAを加水分解しうる[[求核剤|求核物質]]をはねつけて、[[加水分解]]による分解からDNAを保護する<ref name="Reusch">{{cite web | vauthors = Reusch W |title=Nucleic Acids |url=https://www2.chemistry.msu.edu/faculty/reusch/VirtTxtJml/nucacids.htm |publisher=Michigan State University |access-date=30 June 2022}}</ref>。 |
|||
[[File:Estrazione DNA (cropped).jpg|thumb|オレンジから抽出した不純なDNA]] |
|||
=== 巨視的外観 === |
|||
細胞から抽出された純粋なDNAは、白い糸状の凝集塊を形成する<ref>{{cite web |title=How To Extract DNA From Anything Living |url=https://learn.genetics.utah.edu/content/labs/extraction/howto/ |publisher=University of Utah |access-date=30 June 2022}}</ref>。{{Clear}} |
|||
== 化学修飾とDNAパッケージングの変化 == |
|||
<div class="thumb tright" style="background:#f9f9f9; border:1px solid #ccc; margin:0.5em;"> |
|||
{| border="0" cellpadding="2" cellspacing="0" style="width:300px; font-size:85%; border:1px solid #ccc; margin:0.3em;" |
|||
|- |
|||
|[[File:Cytosin.svg|75px]] |
|||
|[[File:5-Methylcytosine.svg|95px]] |
|||
|[[File:Thymin.svg|97px]] |
|||
|- |
|||
| align="center" |[[シトシン]] |
|||
| align="center" |[[5-メチルシトシン]] |
|||
| align="center" |[[チミン]] |
|||
|} |
|||
<div style="border: none; width:300px;font-size: 90%;"><div class="thumbcaption">シトシンがメチル化された5-メチルシトシンは、[[脱アミノ化]]によりチミンに変換される |
|||
</div></div></div> |
|||
=== 塩基修飾とDNAパッケージング === |
|||
{{further|DNAメチル化|クロマチンリモデリング}} |
|||
[[遺伝子発現|遺伝子の発現]]は、DNAが染色体の中で[[クロマチン]]と呼ばれる階層的な構造にどのようにパッケージングされているかに影響される。塩基修飾はパッケージングに関与する可能性があり、遺伝子発現が低いかまったくない領域は通常、[[シトシン]]塩基の[[メチル化]]が高レベルで見られる。DNAパッケージングとその遺伝子発現への影響は、クロマチン構造においてDNAが巻きついている[[ヒストン]]タンパク質コアの共有結合修飾や、[[クロマチンリモデリング|クロマチン・リモデリング]]複合体によるリモデリングでも起こりうる。さらに、DNAメチル化とヒストン修飾の間には{{Ill2|クロストーク (生物学)|en|Crosstalk (biology)|label=クロストーク}}があるため、クロマチンと遺伝子発現に協調的に影響を与える可能性がある<ref>{{cite journal | vauthors = Hu Q, Rosenfeld MG | title = Epigenetic regulation of human embryonic stem cells | journal = Frontiers in Genetics | volume = 3 | pages = 238 | year = 2012 | pmid = 23133442 | pmc = 3488762 | doi = 10.3389/fgene.2012.00238 | doi-access = free }}</ref>。 |
|||
たとえば、シトシンのメチル化は[[5-メチルシトシン]]を生成し、これは[[X染色体の不活性化]]に重要である<ref>{{cite journal | vauthors = Klose RJ, Bird AP | title = Genomic DNA methylation: the mark and its mediators | journal = Trends in Biochemical Sciences | volume = 31 | issue = 2 | pages = 89–97 | date = February 2006 | pmid = 16403636 | doi = 10.1016/j.tibs.2005.12.008 }}</ref>。メチル化の平均レベルは生物によって異なり、[[カエノラブディティス・エレガンス]]({{Snamei|Caenorhabditis elegans}})という線虫はシトシンのメチル化を欠くが、[[脊椎動物]]はメチル化のレベルが高く、DNAの最大1%が5-メチルシトシンを含む<ref>{{cite journal | vauthors = Bird A | title = DNA methylation patterns and epigenetic memory | journal = Genes & Development | volume = 16 | issue = 1 | pages = 6–21 | date = January 2002 | pmid = 11782440 | doi = 10.1101/gad.947102 | doi-access = free }}</ref>。5-メチルシトシンは重要であるにもかかわらず、[[脱アミノ化]]してチミン塩基に変換されることがあるため、メチル化シトシンは特に[[変異]]を起こしやすい<ref>{{cite journal | vauthors = Walsh CP, Xu GL | title = Cytosine methylation and DNA repair | volume = 301 | pages = 283–315 | year = 2006 | pmid = 16570853 | doi = 10.1007/3-540-31390-7_11 | isbn = 3-540-29114-8 | journal = Current Topics in Microbiology and Immunology }}</ref>。その他の塩基修飾としては、細菌におけるアデニンのメチル化、[[脳]]における[[5-ヒドロキシメチルシトシン]]の存在<ref>{{cite journal | vauthors = Kriaucionis S, Heintz N | title = The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain | journal = Science | volume = 324 | issue = 5929 | pages = 929–30 | date = May 2009 | pmid = 19372393 | pmc = 3263819 | doi = 10.1126/science.1169786 | bibcode = 2009Sci...324..929K }}</ref>、および[[キネトプラスト類]]における{{Ill2|塩基J|en|Base J|label=}}を生成するためのウラシルの[[グリコシル化]]などがある<ref>{{cite journal | vauthors = Ratel D, Ravanat JL, Berger F, Wion D | title = N6-methyladenine: the other methylated base of DNA | journal = BioEssays | volume = 28 | issue = 3 | pages = 309–15 | date = March 2006 | pmid = 16479578 | pmc = 2754416 | doi = 10.1002/bies.20342 }}</ref><ref>{{cite journal | vauthors = Gommers-Ampt JH, Van Leeuwen F, de Beer AL, Vliegenthart JF, Dizdaroglu M, Kowalak JA, Crain PF, Borst P | s2cid = 24801094 | title = beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei | journal = Cell | volume = 75 | issue = 6 | pages = 1129–36 | date = December 1993 | pmid = 8261512 | doi = 10.1016/0092-8674(93)90322-H | hdl = 1874/5219 | hdl-access = free }}</ref>。 |
|||
=== 損傷 === |
|||
{{further|{{ill2|DNA損傷 (自然発生)|en|DNA damage (naturally occurring)}}|変異|{{ill2|老化におけるDNA損傷理論|en|DNA damage theory of aging}}}} |
|||
[[File:Benzopyrene DNA adduct 1JDG.png|thumb|right|[[喫煙|タバコの煙]]に含まれる主な[[変異原]]である[[ベンゾピレン|ベンゾ[''a'']ピレン]]の{{Ill2|CYP1A1|en|CYP1A1|label=代謝活性型}}とDNAの[[共有結合]]{{Ill2|付加体|aa|Adduct}}<ref>Created from [http://www.rcsb.org/pdb/cgi/explore.cgi?pdbId=1JDG PDB 1JDG] {{Webarchive|url=https://web.archive.org/web/20080922150848/http://www.rcsb.org/pdb/cgi/explore.cgi?pdbId=1JDG |date=22 September 2008 }}</ref>]] |
|||
DNAは、[[DNAシークエンシング|DNA配列]]を変化させるさまざまな種類の[[変異原]]によって損傷を受ける可能性がある。変異原には、[[酸化剤]]や[[アルキル化剤]]などの化学物質のほか、[[紫外線]]や[[X線]]などの高エネルギー[[電磁放射線]]も含まれる。どのようなDNA損傷が生じるかは変異原の種類によって異なる。たとえば、紫外線はピリミジン塩基間の[[架橋]]である{{Ill2|ピリミジン二量体|en|Pyrimidine dimer|label=チミン二量体|preserve=1}}を生成することによって、DNAに損傷を与える可能性がある<ref>{{cite journal | vauthors = Douki T, Reynaud-Angelin A, Cadet J, Sage E | title = Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation | journal = Biochemistry | volume = 42 | issue = 30 | pages = 9221–26 | date = August 2003 | pmid = 12885257 | doi = 10.1021/bi034593c }}</ref>。一方、[[フリーラジカル]]や[[過酸化水素]]のような酸化剤は、塩基修飾、特にグアノシンの修飾や、二本鎖切断など、さまざまな形の損傷を引き起こす<ref>{{cite journal | vauthors = Cadet J, Delatour T, Douki T, Gasparutto D, Pouget JP, Ravanat JL, Sauvaigo S | title = Hydroxyl radicals and DNA base damage | journal = Mutation Research | volume = 424 | issue = 1–2 | pages = 9–21 | date = March 1999 | pmid = 10064846 | doi = 10.1016/S0027-5107(99)00004-4 }}</ref>。典型的なヒト細胞には、酸化的損傷を受けた塩基が約15万個所ある<ref>{{cite journal | vauthors = Beckman KB, Ames BN | title = Oxidative decay of DNA | journal = The Journal of Biological Chemistry | volume = 272 | issue = 32 | pages = 19633–36 | date = August 1997 | pmid = 9289489 | doi = 10.1074/jbc.272.32.19633 | doi-access = free }}</ref>。これらの酸化的損傷のうち最も危険なのは修復が困難な二本鎖切断であり、[[点変異]]、DNA配列からの{{Ill2|挿入 (遺伝学)|en|Insertion (genetics)|label=挿入}}や[[欠失]]、あるいは[[染色体転座]]を引き起こす可能性がある<ref>{{cite journal | vauthors = Valerie K, Povirk LF | title = Regulation and mechanisms of mammalian double-strand break repair | journal = Oncogene | volume = 22 | issue = 37 | pages = 5792–812 | date = September 2003 | pmid = 12947387 | doi = 10.1038/sj.onc.1206679 | doi-access = free }}</ref>。これらの変異は[[癌]](がん)を引き起こす可能性がある。DNA修復機構には本質的な限界があるため、人間が長生きすれば、いずれは誰も癌を発症することになる<ref name="Weinberg">{{cite news | url = https://www.nytimes.com/2010/12/28/health/28cancer.html | title = Unearthing Prehistoric Tumors, and Debate | newspaper = [[:en:The New York Times|The New York Times]] | date = 28 December 2010 | vauthors = Johnson G | quote = If we lived long enough, sooner or later we all would get cancer. | url-status=live | archive-url = https://web.archive.org/web/20170624233156/http://www.nytimes.com/2010/12/28/health/28cancer.html | archive-date = 24 June 2017 | df = dmy-all }}</ref><ref>{{cite book |vauthors= Alberts B, Johnson A, Lewis J |title= Molecular biology of the cell |publisher= Garland Science |location= New York |year= 2002 |edition= 4th |chapter= The Preventable Causes of Cancer |isbn= 0-8153-4072-9 |chapter-url= https://www.ncbi.nlm.nih.gov/books/NBK26897/ |quote= A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop. |display-authors= etal |url-status=live |archive-url= https://web.archive.org/web/20160102193148/http://www.ncbi.nlm.nih.gov/books/NBK26897/ |archive-date= 2 January 2016 |df= dmy-all }}</ref>。活性酸素種や細胞水の加水分解活性などを産生する正常な細胞プロセスに起因する、{{Ill2|DNA損傷 (自然発生)|en|DNA damage (naturally occurring)|label=自然発生的なDNA損傷}}も頻繁に起こる。これらの損傷の大部分は修復されるが、どの細胞においても、修復過程の作用にもかかわらず、DNA損傷の一部が残ることがある。これらの残存DNA損傷は、哺乳類の[[有糸分裂]]後組織において加齢とともに蓄積する。この蓄積は老化の重要な根本原因であると考えられている<ref>{{cite book | veditors = Kimura H, Suzuki A | title = New Research on DNA Damage | date = 2008 | publisher = Nova Science Publishers | location = New York | isbn = 978-1-60456-581-2 | vauthors = Bernstein H, Payne CM, Bernstein C, Garewal H, Dvorak K | chapter = Cancer and aging as consequences of un-repaired DNA damage | chapter-url = https://www.novapublishers.com/catalog/product_info.php?products_id=43247 | pages = 1–47 | url-status=live | archive-url = https://web.archive.org/web/20141025091740/https://www.novapublishers.com/catalog/product_info.php?products_id=43247 | archive-date = 25 October 2014 | df = dmy-all }}</ref><ref>{{cite journal | vauthors = Hoeijmakers JH | title = DNA damage, aging, and cancer | journal = The New England Journal of Medicine | volume = 361 | issue = 15 | pages = 1475–85 | date = October 2009 | pmid = 19812404 | doi = 10.1056/NEJMra0804615 }}</ref><ref>{{cite journal | vauthors = Freitas AA, de Magalhães JP | title = A review and appraisal of the DNA damage theory of ageing | journal = Mutation Research | volume = 728 | issue = 1–2 | pages = 12–22 | year = 2011 | pmid = 21600302 | doi = 10.1016/j.mrrev.2011.05.001 }}</ref>。 |
|||
変異原の多くは隣接する2つの塩基対の間に侵入し、これは{{Ill2|インターカレーション (生化学)|en|Intercalation (biochemistry)|label=インターカレーション}}({{Lang-en-short|intercalation}})と呼ばれる過程である。ほとんどのインターカレーター(侵入物質)は{{Ill2|芳香族性|en|Aromaticity|label=芳香族|preserve=1}}の平面分子であり、たとえば[[臭化エチジウム]]、[[アクリジン]]、[[ダウノルビシン]]、[[ドキソルビシン]]などである。インターカレーターが塩基対の間に侵入するためには、塩基が離れなければならず、二重らせんがほどけることでDNA鎖に歪みが生じる。これは転写とDNA複製の両方を阻害し、毒性と変異を引き起こす<ref>{{cite journal | vauthors = Ferguson LR, Denny WA | title = The genetic toxicology of acridines | journal = Mutation Research | volume = 258 | issue = 2 | pages = 123–60 | date = September 1991 | pmid = 1881402 | doi = 10.1016/0165-1110(91)90006-H }}</ref>。その結果、DNAインターカレーターは[[発がん性物質|発癌性]]を生じ、またサリドマイドの場合は{{Ill2|奇形学|en|Teratology|label=催奇形性}}を生じる可能性がある<ref>{{cite journal | vauthors = Stephens TD, Bunde CJ, Fillmore BJ | title = Mechanism of action in thalidomide teratogenesis | journal = Biochemical Pharmacology | volume = 59 | issue = 12 | pages = 1489–99 | date = June 2000 | pmid = 10799645 | doi = 10.1016/S0006-2952(99)00388-3 }}</ref>。また、[[ベンゾピレン|ベンゾ[''a'']ピレンジオールエポキシド]]や[[アフラトキシン]]のように、DNA付加体を形成し、複製誤りを引き起こすものもある<ref>{{cite journal | vauthors = Jeffrey AM | title = DNA modification by chemical carcinogens | journal = Pharmacology & Therapeutics | volume = 28 | issue = 2 | pages = 237–72 | year = 1985 | pmid = 3936066 | doi = 10.1016/0163-7258(85)90013-0 }}</ref>。それにもかかわらず、DNAの転写や複製を阻害する能力があるため、他の類似毒素も、急速に増殖する[[癌]]細胞を阻害する[[化学療法 (悪性腫瘍)|化学療法]]に使用されている<ref>{{cite journal | vauthors = Braña MF, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A | title = Intercalators as anticancer drugs | journal = Current Pharmaceutical Design | volume = 7 | issue = 17 | pages = 1745–80 | date = November 2001 | pmid = 11562309 | doi = 10.2174/1381612013397113 }}</ref>。 |
|||
== 生物学的機能 == |
|||
[[File:Eukaryote DNA-en.svg|thumb|upright=1.45|真核生物の染色体内における[[核DNA]]の位置]] |
|||
DNAは通常、[[真核生物]]では線状[[染色体]]として存在し、[[原核生物]]では{{Ill2|環状染色体|en|Circular chromosome}}として存在する。細胞内の染色体の集合が[[ゲノム]]を構成し、[[ヒトゲノム]]では46本の染色体に約30億塩基対のDNAが配置されている<ref name="Venter_2001" />。DNAが伝達する情報は、[[遺伝子]]と呼ばれるDNA断片の[[DNA配列|配列]]に含まれている。遺伝子による遺伝情報の伝達すなわち[[遺伝]]は、相補的な塩基対形成によって達成される。たとえば、転写において細胞が遺伝子の情報を使用する際、DNAと正しいRNAヌクレオチドとの間に引力が作用することで、DNA配列が相補的なRNA配列に複製される。通常、[[翻訳 (生物学)|翻訳]]と呼ばれる過程で、このRNAコピーは一致する[[一次構造|タンパク質配列]]を作るために使用されるが、これもRNAヌクレオチド間の同様な相互作用に依存している。あるいは、細胞は[[DNA複製]]と呼ばれる過程で、その遺伝情報を複製することができる。これらの機能の詳細については他の記事で取り上げており、ここではゲノムの機能を仲介するDNAと他の分子との相互作用に焦点を当てる。 |
|||
=== 遺伝子とゲノム === |
|||
{{further|細胞核|クロマチン|染色体|遺伝子|ノンコーディングDNA}} |
|||
ゲノムDNAは、{{Ill2|DNA凝縮|en|DNA condensation}}と呼ばれる過程を通じて、細胞の小さな体積に収まるようにきつく整然と詰め込まれている。真核生物の場合、DNAは[[細胞核]]に存在し、[[ミトコンドリア]]や[[葉緑体]]にも少量が存在する。原核生物では、DNAは[[核様体]](ヌクレオイド)と呼ばれる細胞質内の不規則な形をした構造体に保持されている<ref>{{cite journal | vauthors = Thanbichler M, Wang SC, Shapiro L | title = The bacterial nucleoid: a highly organized and dynamic structure | journal = Journal of Cellular Biochemistry | volume = 96 | issue = 3 | pages = 506–21 | date = October 2005 | pmid = 15988757 | doi = 10.1002/jcb.20519 | doi-access = free }}</ref>。ゲノムの遺伝情報は遺伝子内に保持されており、生物におけるこの情報の完全な集合をその[[遺伝子型|遺伝型]]({{Lang-en-short|genotype}})と呼ぶ。遺伝子は[[遺伝]]の単位であり、生物の特定の[[形質]]に影響を与えるDNAの領域である。遺伝子には、転写可能な[[オープンリーディングフレーム]]と、オープンリーディングフレームの転写を制御する[[プロモーター]]や[[エンハンサー]]などの{{Ill2|制御配列|en|Regulatory sequence}}が含まれている。 |
|||
多くの[[生物種]]では、[[ゲノム]]配列全体のごく一部のみタンパク質を[[遺伝コード|コード]]している。たとえば、ヒトゲノムのうちタンパク質をコードする[[エクソン]]はわずか約1.5%しかなく、ヒトDNAの50%以上は非コード[[反復配列]]で構成されている<ref>{{cite journal | vauthors = Wolfsberg TG, McEntyre J, Schuler GD | title = Guide to the draft human genome | journal = Nature | volume = 409 | issue = 6822 | pages = 824–26 | date = February 2001 | pmid = 11236998 | doi = 10.1038/35057000 | bibcode = 2001Natur.409..824W | url = https://zenodo.org/record/1233093 | doi-access = free }}</ref>。真核生物のゲノムに非常に多くの[[ノンコーディングDNA|非コードDNA]]が存在する理由と、{{Ill2|ゲノムの大きさ|en|Genome size|label=ゲノムの大きさ}}({{Ill2|C値|en|C-value|label=''C値''}})が生物種によって著しく異なる理由は、「{{Ill2|C値の謎|en|C-value enigma}}」として知られる長年の難問である<ref>{{cite journal | vauthors = Gregory TR | title = The C-value enigma in plants and animals: a review of parallels and an appeal for partnership | journal = Annals of Botany | volume = 95 | issue = 1 | pages = 133–46 | date = January 2005 | pmid = 15596463 | doi = 10.1093/aob/mci009 | pmc = 4246714 }}</ref>。しかし、タンパク質をコードしないDNA配列の中には、[[遺伝子発現の調節]]に関与する機能的な[[ノンコーディングRNA|非コードRNA]]分子をコードしているものもある<ref name="Birney_2007" />。 |
|||
[[File:T7 RNA polymerase.jpg|thumb|{{Ill2|T7 RNAポリメラーゼ|en|T7 RNA polymerase}}<small>(青)</small> は、DNA鋳型 <small>(橙)</small> からmRNA <small>(緑)</small> を生成する<ref>{{Cite web| vauthors = Yin YW, Steitz TA |title=RCSB PDB – 1MSW: Structural basis for the transition from initiation to elongation transcription in T7 RNA polymerase|url=https://www.rcsb.org/structure/1MSW|access-date=2023-03-27|website=www.rcsb.org|language=en-US}}</ref>。]] |
|||
非コードDNA配列の中には染色体の構造的役割を果たすものがある。[[テロメア]]と[[セントロメア]]には通常、ほとんど遺伝子が存在しないが、染色体の機能と安定性にとって重要である<ref name="Nugent" /><ref>{{cite journal | vauthors = Pidoux AL, Allshire RC | title = The role of heterochromatin in centromere function | journal = Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences | volume = 360 | issue = 1455 | pages = 569–79 | date = March 2005 | pmid = 15905142 | pmc = 1569473 | doi = 10.1098/rstb.2004.1611 }}</ref>。ヒトに多く存在する非コードDNAは{{Ill2|偽遺伝子|en|Pseudogene|preserve=1}}であり、変異によって機能しなくなった遺伝子の複製である<ref>{{cite journal | vauthors = Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M | title = Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22 | journal = Genome Research | volume = 12 | issue = 2 | pages = 272–80 | date = February 2002 | pmid = 11827946 | pmc = 155275 | doi = 10.1101/gr.207102 }}</ref>。これらの配列は、遺伝子の[[遺伝子重複|重複]]や{{Ill2|分岐進化|en|Divergent evolution|label=分岐}}の過程を通じて、新しい遺伝子を生み出すための[[ゲノム|遺伝物質]]の原料として役に立つこともあるが、通常は単なる分子の[[化石|遺物]]である<ref>{{cite journal | vauthors = Harrison PM, Gerstein M | title = Studying genomes through the aeons: protein families, pseudogenes and proteome evolution | journal = Journal of Molecular Biology | volume = 318 | issue = 5 | pages = 1155–74 | date = May 2002 | pmid = 12083509 | doi = 10.1016/S0022-2836(02)00109-2 }}</ref>。 |
|||
=== 転写と翻訳 === |
|||
{{further|{{ill2|遺伝情報|en|Genetic code|preserve=1}}|転写 (生物学)|タンパク質生合成}} |
|||
遺伝子は遺伝情報を含むDNA配列で、生物の[[表現型]]に影響を与えることがある。遺伝子内では、DNA鎖に沿った塩基配列が[[伝令RNA|メッセンジャーRNA]]配列を規定し、それが1つか複数のタンパク質配列を規定する。遺伝子のヌクレオチド配列とタンパク質の[[アミノ酸]]配列との関係は、{{ill2|遺伝暗号|en|Genetic code|preserve=1}}と総称される[[翻訳 (生物学)|翻訳]]規則によって決定される。遺伝暗号は、コドン(''codon'')と呼ばれる3文字の「単語」からなり(例:ACT、CAG、TTT)、ヌクレオチドが3個連続した配列に基づいている。 |
|||
転写の際、遺伝子のコドンが[[RNAポリメラーゼ]]によってメッセンジャーRNAにコピーされる。次に、このRNAコピーは[[リボソーム]]によって解読され、リボソームはメッセンジャーRNAをアミノ酸を運ぶ[[転移RNA|トランスファーRNA]]に塩基対合させることによってRNA配列を読み取る。4種類の塩基を表す3文字が組み合わさって、64通りのコドンの可能性が存在する(4<sup>3</sup> 通りの組み合わせ)。これらのコドンは20種類の[[タンパク質を構成するアミノ酸|標準アミノ酸]]をコードしており、ほとんどのアミノ酸は複数のコドンに対応付けられる。また、コード領域の終わりを示す3つの「[[終止コドン|終止]]コドン」(ナンセンスコドンとも呼ばれる)もある。これらは、TAG、TAA、TGAコドンである(mRNAではUAG、UAA、UGA)。 |
|||
[[File:DNA replication en.svg|thumb|DNA[[複製フォーク]]の模式図。DNA二重らせんは[[ヘリカーゼ]]と[[DNAトポイソメラーゼ|トポイソメラーゼ]]によってほどかれる。次に、一つのDNAポリメラーゼが[[リーディング鎖]]の複製を作る。もう一つのDNAポリメラーゼが[[ラギング鎖]]に結合する。この酵素は、[[DNAリガーゼ]]がそれらを結合する前に、不連続なセグメント ([[岡崎フラグメント]]と呼ばれる) を作る。|440x440ピクセル]] |
|||
=== 複製 === |
|||
{{further|DNA複製}} |
|||
[[細胞分裂]]は生物が成長するために不可欠であるが、細胞が分裂する際には、2つの娘細胞が親と同じ遺伝情報を持つように、ゲノム中のDNAを複製しなければならない。DNAの二本鎖構造は[[DNA複製]]の単純な機構を提供する。ここでは二本鎖が分離され、次に[[DNAポリメラーゼ]]と呼ばれる[[酵素]]によってそれぞれの鎖の[[相補的DNA]]配列が再作成される。この酵素は、相補的塩基対の形成を通じて正しい塩基を見つけ、それを元の鎖に結合させることで相補鎖を作成する。DNAポリメラーゼはDNA鎖を5'から3'の方向にしか伸長できないため、二重らせんの逆平行鎖を複製するために異なる機構が使われる<ref>{{cite journal | vauthors = Albà M | title = Replicative DNA polymerases | journal = Genome Biology | volume = 2 | issue = 1 | pages = REVIEWS3002 | year = 2001 | pmid = 11178285 | pmc = 150442 | doi = 10.1186/gb-2001-2-1-reviews3002 | doi-access = free }}</ref>。このようにして、古い鎖の塩基が新しい鎖の塩基を決定し、細胞はそのDNAの完全な複製を得ることができる。 |
|||
=== 細胞外核酸 === |
|||
裸の細胞外DNA({{Lang-en-short|extracellular DNA}}、eDNA)は、そのほとんどが[[細胞死]]の際に放出されたもので、環境中にほぼ遍在している。土壌中の濃度は2 μg/Lと高く、自然の水性環境中では88 μg/Lに達することもある<ref name="Tani_2010">{{cite book | vauthors = Tani K, Nasu M | veditors = Kikuchi Y, Rykova EY | title = Extracellular Nucleic Acids |url=https://archive.org/details/extracellularnuc00kiku |url-access=limited |publisher=Springer |date=2010 |pages=[https://archive.org/details/extracellularnuc00kiku/page/n35 25]–38 |chapter=Roles of Extracellular DNA in Bacterial Ecosystems |isbn=978-3-642-12616-1}}</ref>。eDNAの働きとして、[[遺伝子の水平伝播]]への関与<ref name="Vlassov_2007">{{cite journal | vauthors = Vlassov VV, Laktionov PP, Rykova EY | title = Extracellular nucleic acids | journal = BioEssays | volume = 29 | issue = 7 | pages = 654–67 | date = July 2007 | pmid = 17563084 | doi = 10.1002/bies.20604 | s2cid = 32463239 }}</ref>、栄養素の供給<ref name="pmid11591672">{{cite journal | vauthors = Finkel SE, Kolter R | title = DNA as a nutrient: novel role for bacterial competence gene homologs | journal = Journal of Bacteriology | volume = 183 | issue = 21 | pages = 6288–93 | date = November 2001 | pmid = 11591672 | pmc = 100116 | doi = 10.1128/JB.183.21.6288-6293.2001 }}</ref>、あるいはイオンや抗生物質を取り込んだり用量を調整するための緩衝剤としての機能など、さまざまな可能性が提案されている<ref name="Mulcahy_2008">{{cite journal | vauthors = Mulcahy H, Charron-Mazenod L, Lewenza S | title = Extracellular DNA chelates cations and induces antibiotic resistance in Pseudomonas aeruginosa biofilms | journal = PLOS Pathogens | volume = 4 | issue = 11 | pages = e1000213 | date = November 2008 | pmid = 19023416 | pmc = 2581603 | doi = 10.1371/journal.ppat.1000213 | doi-access = free }}</ref>。eDNAは、いくつかの細菌種の[[バイオフィルム]]において、機能的な[[細胞外マトリックス]]成分として機能する。eDNAの働きには、バイオフィルム内の特定の細胞型の付着と分散を制御する認識因子として働く可能性や<ref name="Berne_2010">{{cite journal | vauthors = Berne C, Kysela DT, Brun YV | title = A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm | journal = Molecular Microbiology | volume = 77 | issue = 4 | pages = 815–29 | date = August 2010 | pmid = 20598083 | pmc = 2962764 | doi = 10.1111/j.1365-2958.2010.07267.x }}</ref>、バイオフィルム形成に寄与する可能性<ref name="Whitchurch_2002">{{cite journal | vauthors = Whitchurch CB, Tolker-Nielsen T, Ragas PC, Mattick JS | title = Extracellular DNA required for bacterial biofilm formation | journal = Science | volume = 295 | issue = 5559 | pages = 1487 | date = February 2002 | pmid = 11859186 | doi = 10.1126/science.295.5559.1487 }}</ref>、あるいはバイオフィルムの物理的強度と生物学的ストレスに対する抵抗性に寄与する可能性がある<ref name="Hu_2012">{{cite journal | vauthors = Hu W, Li L, Sharma S, Wang J, McHardy I, Lux R, Yang Z, He X, Gimzewski JK, Li Y, Shi W | title = DNA builds and strengthens the extracellular matrix in Myxococcus xanthus biofilms by interacting with exopolysaccharides | journal = PLOS ONE | volume = 7 | issue = 12 | pages = e51905 | year = 2012 | pmid = 23300576 | pmc = 3530553 | doi = 10.1371/journal.pone.0051905 | bibcode = 2012PLoSO...751905H | doi-access = free }}</ref>。 |
|||
{{Ill2|無細胞胎児DNA|en|Cell-free fetal DNA}}は母体の血液中に存在し、その塩基配列を決定することで発達中の[[胎児]]に関する多くの情報を得ることができる<ref name="Hui_2013">{{cite journal | vauthors = Hui L, Bianchi DW | title = Recent advances in the prenatal interrogation of the human fetal genome | journal = Trends in Genetics | volume = 29 | issue = 2 | pages = 84–91 | date = February 2013 | pmid = 23158400 | pmc = 4378900 | doi = 10.1016/j.tig.2012.10.013 }}</ref>。 |
|||
[[環境DNA]]として知られるeDNAは、水中、大気中、陸上における生物種の動きと存在を監視し、その地域の生物多様性を評価する[[生態学]]の調査ツールとして、自然科学の分野で利用が拡大している<ref>{{cite journal | vauthors = Foote AD, Thomsen PF, Sveegaard S, Wahlberg M, Kielgast J, Kyhn LA, Salling AB, Galatius A, Orlando L, Gilbert MT | display-authors = 6 | title = Investigating the potential use of environmental DNA (eDNA) for genetic monitoring of marine mammals | journal = PLOS ONE | volume = 7 | issue = 8 | pages = e41781 | year = 2012 | pmid = 22952587 | pmc = 3430683 | doi = 10.1371/journal.pone.0041781 | bibcode = 2012PLoSO...741781F | doi-access = free }}</ref><ref>{{Cite web |url=https://www.the-scientist.com/news-opinion/researchers-detect-land-animals-using-dna-in-nearby-water-bodies-67481 |title=Researchers Detect Land Animals Using DNA in Nearby Water Bodies |access-date=2020-05-24}}</ref>。 |
|||
=== 好中球細胞外トラップ === |
|||
{{Main|{{ill2|好中球細胞外トラップ|en|Neutrophil extracellular traps}}}} |
|||
好中球細胞外トラップ({{Lang-en-short|neutrophil extracellular trap}}、NET)は、主にDNAから構成される細胞外繊維のネットワークであり、白血球の一種である[[好中球]]が[[宿主]]細胞への損傷を最小限に抑えながら細胞外の病原体を殺滅することを可能にする。 |
|||
== タンパク質との相互作用 == |
|||
DNAの機能はすべて[[タンパク質]]との{{Ill2|タンパク質相互作用|en|Protein interactions|label=相互作用}}に依存している。これらのタンパク質相互作用は非特異的であることもあれば、タンパク質が単一のDNA配列に特異的に結合することもある。酵素もDNAに結合することができ、その中でも特に重要なものは、転写とDNA複製の際にDNA塩基配列をコピーするポリメラーゼである。 |
|||
=== DNA結合タンパク質 === |
|||
{{further|DNA結合タンパク質}} |
|||
[[File:Nucleosome1.png|thumb|260px|left|DNA <small>(橙色)</small> と[[ヒストン]] <small>(青色)</small> の相互作用を示す三次元図。これらのタンパク質の塩基性アミノ酸は、DNA上の酸性リン酸基と結合する。]] |
|||
DNAと結合する構造タンパク質は、非特異的DNA-タンパク質相互作用の例としてよく理解されている。染色体内でDNAは構造タンパク質と複合体を形成して保持されている。これらのタンパク質はDNAを[[クロマチン]](染色質)と呼ばれる緻密な構造に組織化する。真核生物では、この構造は[[ヒストン]]という小さな塩基性タンパク質の複合体にDNAが結合したものであるが、原核生物では複数種類のタンパク質が関与している<ref>{{cite journal | vauthors = Sandman K, Pereira SL, Reeve JN | s2cid = 21101836 | title = Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome | journal = Cellular and Molecular Life Sciences | volume = 54 | issue = 12 | pages = 1350–64 | date = December 1998 | pmid = 9893710 | doi = 10.1007/s000180050259 }}</ref><ref>{{cite journal | vauthors = Dame RT | title = The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin | journal = Molecular Microbiology | volume = 56 | issue = 4 | pages = 858–70 | date = May 2005 | pmid = 15853876 | doi = 10.1111/j.1365-2958.2005.04598.x | s2cid = 26965112 | doi-access = free }}</ref>。ヒストンは[[ヌクレオソーム]]と呼ばれる円盤状の複合体を形成し、その表面には二本鎖DNAが2周完全に巻きついている。これらの非特異的相互作用は、ヒストンの塩基性残基がDNAの酸性糖-リン酸骨格と[[イオン結合]]を形成することによって生じるもので、したがって、塩基配列とはほとんど無関係である<ref>{{cite journal | vauthors = Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ | title = Crystal structure of the nucleosome core particle at 2.8 A resolution | journal = Nature | volume = 389 | issue = 6648 | pages = 251–60 | date = September 1997 | pmid = 9305837 | doi = 10.1038/38444 | bibcode = 1997Natur.389..251L | s2cid = 4328827 }}</ref>。これらの塩基性アミノ酸残基の化学修飾には、[[メチル化]]、[[リン酸化]]、[[アセチル化]]などがある<ref>{{cite journal | vauthors = Jenuwein T, Allis CD | title = Translating the histone code | journal = Science | volume = 293 | issue = 5532 | pages = 1074–80 | date = August 2001 | pmid = 11498575 | doi = 10.1126/science.1063127 | s2cid = 1883924 | url = http://www.gs.washington.edu/academics/courses/braun/55104/readings/jenuwein.pdf | url-status=live | archive-url = https://web.archive.org/web/20170808142426/http://www.gs.washington.edu/academics/courses/braun/55104/readings/jenuwein.pdf | archive-date = 8 August 2017 | df = dmy-all }}</ref>。これらの化学的変化はDNAとヒストン間の相互作用の強度を変化させ、DNAを[[転写因子]]に近づきやすくしたり、あるいは近づきにくくし、転写速度を変化させる<ref>{{cite book | vauthors = Ito T | title = Protein Complexes that Modify Chromatin | chapter = Nucleosome Assembly and Remodeling | series = Current Topics in Microbiology and Immunology | volume = 274 | pages = 1–22 | year = 2003 | pmid = 12596902 | doi = 10.1007/978-3-642-55747-7_1 | isbn = 978-3-540-44208-0 }}</ref>。クロマチン内の他の非特異的[[DNA結合タンパク質]]には、曲がったDNAや歪んだDNAに結合する[[高移動度群タンパク質|高移動度郡タンパク質]]がある<ref>{{cite journal | vauthors = Thomas JO | title = HMG1 and 2: architectural DNA-binding proteins | journal = Biochemical Society Transactions | volume = 29 | issue = Pt 4 | pages = 395–401 | date = August 2001 | pmid = 11497996 | doi = 10.1042/BST0290395 }}</ref>。これらのタンパク質は、ヌクレオソームの配列を曲げたり、染色体を構成する大きな構造体を組み立てる際に重要である<ref>{{cite journal | vauthors = Grosschedl R, Giese K, Pagel J | title = HMG domain proteins: architectural elements in the assembly of nucleoprotein structures | journal = Trends in Genetics | volume = 10 | issue = 3 | pages = 94–100 | date = March 1994 | pmid = 8178371 | doi = 10.1016/0168-9525(94)90232-1 }}</ref>。 |
|||
DNA結合タンパク質のもう一つのグループとして、一本鎖DNAと特異的に結合するDNA結合タンパク質がある。ヒトの場合、[[複製タンパク質A]]がこの一群の中で最もよく理解されており、DNA複製、組換え、DNA修復など、二重らせんが分離するプロセスに関与している<ref>{{cite journal | vauthors = Iftode C, Daniely Y, Borowiec JA | title = Replication protein A (RPA): the eukaryotic SSB | journal = Critical Reviews in Biochemistry and Molecular Biology | volume = 34 | issue = 3 | pages = 141–80 | year = 1999 | pmid = 10473346 | doi = 10.1080/10409239991209255 }}</ref>。これらの結合タンパク質は一本鎖DNAを安定化させ、[[ステムループ]]を形成したり、[[ヌクレアーゼ]]による分解からDNAを保護していると考えられている。 |
|||
[[File:Lambda repressor 1LMB.png|thumb|upright=1.1|ラムダリプレッサー・[[ヘリックスターンヘリックス]]転写因子が、DNAターゲットに結合している<ref>{{Cite web| vauthors = Beamer LJ, Pabo CO |title=RCSB PDB – 1LMB: Refined 1.8 Å crystal structure of the lambda repressor-operator complex |url=https://www.rcsb.org/structure/1LMB|access-date=2023-03-27|website=www.rcsb.org|language=en-US}}</ref>。]] |
|||
対照的に、他のタンパク質は特定のDNA配列に結合するような進化をしてきた。最も研究が進んでいるのは、転写を制御するタンパク質であるさまざまな[[転写因子]]である。各転写因子はプロモーター近くの特定のDNA配列に結合し、遺伝子の転写を活性化または阻害する。転写因子は2つの方法でこれを行う。一つは、転写を担うRNAポリメラーゼに直接、あるいは他の媒介タンパク質を介して結合することである。これによって、ポリメラーゼはプロモーターに位置し、転写を開始することができる<ref>{{cite journal | vauthors = Myers LC, Kornberg RD | title = Mediator of transcriptional regulation | journal = Annual Review of Biochemistry | volume = 69 | pages = 729–49 | year = 2000 | pmid = 10966474 | doi = 10.1146/annurev.biochem.69.1.729 }}</ref>。あるいは、転写因子はプロモーターのヒストンを修飾する[[酵素]]と結合することができる。これによってDNA鋳型に対するポリメラーゼの近づきやすさを変化させる<ref>{{cite journal | vauthors = Spiegelman BM, Heinrich R | title = Biological control through regulated transcriptional coactivators | journal = Cell | volume = 119 | issue = 2 | pages = 157–67 | date = October 2004 | pmid = 15479634 | doi = 10.1016/j.cell.2004.09.037 | doi-access = free }}</ref>。 |
|||
これらのDNA[[生物学的標的|標的]]は生物のゲノム全体に存在する可能性があるため、一種類の転写因子の活性が変化すると、何千もの遺伝子に影響を及ぼす可能性がある<ref>{{cite journal | vauthors = Li Z, Van Calcar S, Qu C, Cavenee WK, Zhang MQ, Ren B | title = A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 100 | issue = 14 | pages = 8164–69 | date = July 2003 | pmid = 12808131 | pmc = 166200 | doi = 10.1073/pnas.1332764100 | bibcode = 2003PNAS..100.8164L | doi-access = free }}</ref>。その結果、これらのタンパク質はしばしば、環境変化への応答や[[細胞分化|細胞の分化]]・発達を制御する[[シグナル伝達]]プロセスの標的となる。これらの転写因子のDNAとの相互作用の特異性は、タンパク質がDNA塩基の端と何度も接触して、DNA配列を「読み取る」ことを可能にすることで生じる。これらの塩基相互作用のほとんどは塩基が最も接近しやすい主溝で起こる<ref name="Pabo1984" />。 |
|||
[[File:EcoRV 1RVA.png|thumb|upright=1.1|[[制限酵素]][[EcoRV]] <small>(緑色)</small> と基質DNA <small>(赤と青)</small> の複合体<ref>{{Cite web| vauthors = Kostrewa D, Winkler FK |title=RCSB PDB – 1RVA: Mg2+ binding to the active site of EcoRV endonuclease: a crystallographic study of complexes with substrate and product DNA at 2 Å resolution |url=https://www.rcsb.org/structure/1RVA|access-date=2023-03-27|website=www.rcsb.org|language=en-US}}</ref>]] |
|||
=== DNA修飾酵素 === |
|||
==== ヌクレアーゼとリガーゼ ==== |
|||
[[ヌクレアーゼ]]は、[[ホスホジエステル結合]]の[[加水分解]]を触媒することによってDNA鎖を切断する[[酵素]]である。DNA鎖の末端からヌクレオチドを加水分解するヌクレアーゼは[[エキソヌクレアーゼ]]と呼ばれ、一方[[エンドヌクレアーゼ]]は鎖内で切断する。[[分子生物学]]で最もよく使用されるヌクレアーゼは、特異的配列でDNAを切断する[[制限エンドヌクレアーゼ]]である。たとえば、上図に示したEcoRV酵素は、DNA鎖の6塩基配列 5′-GATATC-3′ を認識し、水平線で切断する。自然界でこれらの酵素は、[[制限修飾系]]の一部として[[細菌]]の細胞内に侵入した[[ファージ]]DNAを消化することにより、細菌をファージ感染から保護している<ref>{{cite journal | vauthors = Bickle TA, Krüger DH | title = Biology of DNA restriction | journal = Microbiological Reviews | volume = 57 | issue = 2 | pages = 434–50 | date = June 1993 | pmid = 8336674 | pmc = 372918 | doi = 10.1128/MMBR.57.2.434-450.1993 }}</ref>。技術分野では、これらの配列特異的ヌクレアーゼは{{Ill2|分子クローニング|en|Molecular cloning}}や[[DNAプロファイリング]]に使用されている。 |
|||
[[DNAリガーゼ]]と呼ばれる酵素は、切断または破損したDNA鎖を再結合させることができる<ref name="Doherty">{{cite journal | vauthors = Doherty AJ, Suh SW | title = Structural and mechanistic conservation in DNA ligases | journal = Nucleic Acids Research | volume = 28 | issue = 21 | pages = 4051–58 | date = November 2000 | pmid = 11058099 | pmc = 113121 | doi = 10.1093/nar/28.21.4051 }}</ref>。リガーゼは、[[ラギング鎖]]DNA複製において特に重要で、[[複製フォーク]]で作られた短いDNAセグメントをDNA鋳型の完全なコピーに結合する働きをする。これらはまた[[DNA修復]]や[[遺伝的組換え]]にも使用される<ref name="Doherty" />。 |
|||
==== トポイソメラーゼとヘリカーゼ ==== |
|||
[[DNAトポイソメラーゼ|トポイソメラーゼ]]はヌクレアーゼとリガーゼの両方の活性を持つ酵素である。これらのタンパク質はDNA[[DNAスーパーコイル|スーパーコイル]]の量を変化させる。これらの酵素の中には、DNAらせんを切断し、その一部分を回転させることでスーパーコイルのひずみを低減させ、その後DNAの切断部を封着するものもある<ref name="Champoux" />。別の種類の酵素は、DNAらせんを切断し、その切断部分に2本目のDNAを通過させてから、らせんを再結合することができる<ref>{{cite journal | vauthors = Schoeffler AJ, Berger JM | title = Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism | journal = Biochemical Society Transactions | volume = 33 | issue = Pt 6 | pages = 1465–70 | date = December 2005 | pmid = 16246147 | doi = 10.1042/BST20051465 }}</ref>。このようにトポイソメラーゼは、DNA複製や転写など、DNAが関与する多くの過程に必要な酵素である<ref name="Wang" />。 |
|||
[[ヘリカーゼ]]は[[分子モーター]]として働くタンパク質である。これらは、[[ヌクレオシド三リン酸]]、主に[[アデノシン三リン酸]](ATP)の[[化学エネルギー]]を利用して、塩基間の水素結合を切断し、DNA二重らせんをほどいて一本鎖にする<ref>{{cite journal | vauthors = Tuteja N, Tuteja R | title = Unraveling DNA helicases. Motif, structure, mechanism and function | journal = European Journal of Biochemistry | volume = 271 | issue = 10 | pages = 1849–63 | date = May 2004 | pmid = 15128295 | doi = 10.1111/j.1432-1033.2004.04094.x | url = http://repository.ias.ac.in/52775/1/40-pub.pdf | doi-access = free }}</ref>。これらの酵素は、酵素がDNA塩基に近接する必要があるほとんどの過程にとって不可欠である。 |
|||
==== ポリメラーゼ ==== |
|||
[[ポリメラーゼ]]は[[ヌクレオシド三リン酸]]からポリヌクレオチド鎖を合成する[[酵素]]である。その生成物の配列は、鋳型({{Lang-en-short|template}})と呼ばれる既存のポリヌクレオチド鎖に基づいて作られる。これらの酵素は、伸長するポリヌクレオチド鎖末端の3'[[ヒドロキシ基]]に繰り返しヌクレオチドを付加する機能を持つ。結果としてすべてのポリメラーゼは5'から3'の方向に働く<ref name="Joyce">{{cite journal | vauthors = Joyce CM, Steitz TA | title = Polymerase structures and function: variations on a theme? | journal = Journal of Bacteriology | volume = 177 | issue = 22 | pages = 6321–29 | date = November 1995 | pmid = 7592405 | pmc = 177480 | doi=10.1128/jb.177.22.6321-6329.1995}}</ref>。これらの酵素の[[活性部位]]では、入ってきたヌクレオシド三リン酸が鋳型と塩基対を形成する。これにより、ポリメラーゼは鋳型の相補鎖を正確に合成することができる。ポリメラーゼは、使用する鋳型の種類によって分類される。 |
|||
DNA複製は、DNA依存性[[DNAポリメラーゼ]]がDNAポリヌクレオチド鎖のコピーを作る。生物学的情報を保存するためには、各コピーの塩基配列が鋳型鎖の塩基配列と正確に相補的であることが不可欠である。多くのDNAポリメラーゼは[[校正 (生物学)|校正]]活性を持っている。これによりポリメラーゼは、ミスマッチしたヌクレオチド間での塩基対形成の欠如によって、合成反応の際にときおり起こる誤りを検出することができる。ミスマッチが検出されると、3'→5'[[エキソヌクレアーゼ]]活性が活性化され、誤った塩基が除去される<ref>{{cite journal | vauthors = Hubscher U, Maga G, Spadari S | s2cid = 26171993 | title = Eukaryotic DNA polymerases | journal = Annual Review of Biochemistry | volume = 71 | pages = 133–63 | year = 2002 | pmid = 12045093 | doi = 10.1146/annurev.biochem.71.090501.150041 | url = http://pdfs.semanticscholar.org/e941/98efed7eb8fa606b87d9a44c118c235a62e9.pdf | archive-url = https://web.archive.org/web/20210126170051/http://pdfs.semanticscholar.org/e941/98efed7eb8fa606b87d9a44c118c235a62e9.pdf | url-status = dead | archive-date = 26 January 2021 }}</ref>。ほとんどの生物でDNAポリメラーゼは、[[DNAクランプ]]や[[ヘリカーゼ]]などの複数のアクセサリー・サブユニットを含む、{{Ill2|レプリソーム|en|Replisome}}と呼ばれる大きな複合体の中で機能する<ref>{{cite journal | vauthors = Johnson A, O'Donnell M | title = Cellular DNA replicases: components and dynamics at the replication fork | journal = Annual Review of Biochemistry | volume = 74 | pages = 283–315 | year = 2005 | pmid = 15952889 | doi = 10.1146/annurev.biochem.73.011303.073859 }}</ref>。 |
|||
RNA依存性DNAポリメラーゼは、RNA鎖の塩基配列をDNAにコピーする特殊なポリメラーゼである。これらには、[[レトロウイルス科|レトロウイルス]]による細胞感染に関与する[[ウイルス|ウイルス性]]酵素である[[逆転写酵素]]や、テロメアの複製に必要な[[テロメラーゼ]]が含まれる<ref name="Greider" /><ref name="Tarrago-Litvak1994">{{cite journal | vauthors = Tarrago-Litvak L, Andréola ML, Nevinsky GA, Sarih-Cottin L, Litvak S | title = The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention | journal = FASEB Journal | volume = 8 | issue = 8 | pages = 497–503 | date = May 1994 | pmid = 7514143 | doi = 10.1096/fasebj.8.8.7514143 | s2cid = 39614573 | url = http://www.fasebj.org/doi/pdf/10.1096/fasebj.8.8.7514143 }}</ref>。たとえば、HIV逆転写酵素は、[[エイズウイルス]]の複製に関与する酵素である<ref name="Tarrago-Litvak1994" />。テロメラーゼは、その構造の一部として自身のRNA鋳型を含むという珍しいポリメラーゼである。これは染色体の末端に[[テロメア]]を合成する。テロメアは隣接する染色体末端が融合するのを防ぎ、染色体末端を損傷から保護する<ref name="Nugent" />。 |
|||
転写は、DNA鎖の配列をRNAにコピーするDNA依存性[[RNAポリメラーゼ]]によって行われる。遺伝子の転写を開始するために、RNAポリメラーゼはプロモーターと呼ばれるDNA配列に結合し、DNA鎖を分離する。その後、[[ターミネーター (遺伝学)|ターミネーター]]と呼ばれるDNAの領域に到達するまで、遺伝子配列を[[伝令RNA|メッセンジャーRNA]]転写物にコピーし、そこで停止してDNAから分離する。ヒトのDNA依存性DNAポリメラーゼと同様に、ヒトゲノムのほとんどの遺伝子を転写する酵素である[[RNAポリメラーゼII]]は、いくつか調節サブユニットとアクセサリーサブユニットを持つ大きな[[タンパク質複合体]]の一部として働いている<ref>{{cite journal | vauthors = Martinez E | s2cid = 24946189 | title = Multi-protein complexes in eukaryotic gene transcription | journal = Plant Molecular Biology | volume = 50 | issue = 6 | pages = 925–47 | date = December 2002 | pmid = 12516863 | doi = 10.1023/A:1021258713850 }}</ref>。 |
|||
== 遺伝子組換え == |
|||
<div class="thumb tright" style="background:#f9f9f9; border:1px solid #ccc; margin:0.5em;"> |
|||
{| border="0" border="0" cellpadding="0" cellspacing="0" style="width:250px; font-size:85%; border:1px solid #ccc; margin:0.3em;" |
|||
|- |
|||
|[[File:Holliday Junction.svg|220x220px]] |
|||
|[[File:Holliday junction coloured.png|220x220px]] |
|||
|- |
|||
| colspan="2" |[[遺伝的組換え]]における[[ホリデイジャンクション]]中間体の構造。4本のDNA鎖は、赤、青、緑、黄に色分けされている<ref name="Thorpe_2003">{{Cite web |vauthors=Thorpe JH, Gale BC, Teixeira SC, Cardin CJ |title=RCSB PDB – 1M6G: Structural Characterisation of the Holliday Junction TCGGTACCGA |url=https://www.rcsb.org/structure/1M6G |access-date=2023-03-27 |website=www.rcsb.org |language=en-US}}</ref> |
|||
|} |
|||
</div> |
|||
{{further|遺伝的組換え}} |
|||
[[File:Homologous Recombination.jpg|thumb|300px| 現在の減数分裂の組換えモデルは二本鎖切断またはギャップによって開始され、その後、相同染色体との対合とストランド侵入によって組換え修復プロセスが開始される。ギャップ修復は、隣接領域のクロスオーバー (CO) やノンクロスオーバー (NCO) をもたらす。CO組換えは、上図右側のダブルホリデイジャンクション ({{Lang-en-short|Double Holliday Junction}}、DHJ) モデルによって起こると考えられている。NCO組換えは、主に左側の合成依存差 ({{Lang-en-short|Synthesis Dependent Strand Annealing}}、SDSA) モデルによって起こると考えられている。ほとんどの組換え事象はSDSA型と考えられる。]] |
|||
DNAらせんは通常、他のDNAセグメントと相互作用することはなく、ヒトの細胞では、異なる染色体は{{Ill2|染色体テリトリー|en|Chromosome territories}}({{Lang-en-short|chromosome territories}})と呼ばれる核内の別々の領域を占めることさえある<ref>{{cite journal | vauthors = Cremer T, Cremer C | title = Chromosome territories, nuclear architecture and gene regulation in mammalian cells | journal = Nature Reviews Genetics | volume = 2 | issue = 4 | pages = 292–301 | date = April 2001 | pmid = 11283701 | doi = 10.1038/35066075 | s2cid = 8547149 }}</ref>。このように異なる染色体が物理的に分離していることは、DNAが安定した情報保管場所として機能するために重要である。なぜなら、染色体が相互作用する数少ない機会のひとつが、[[有性生殖]]の際に起こる[[乗換え (生物学)|染色体交差]]({{Lang-en-short|chromosomal crossover}})であり、その際に[[遺伝的組換え]]が起こるからである。染色体交差とは、DNAの2本のらせんが切断され、一部が入れ替わり、再び結合することである。 |
|||
組換えは、染色体が遺伝情報を交換して遺伝子の新しい組み合わせを作り出すことを可能にし、これにより[[自然選択説|自然選択]]の効率を高め、新しいタンパク質の急速な進化において重要である<ref>{{cite journal | vauthors = Pál C, Papp B, Lercher MJ | title = An integrated view of protein evolution | journal = Nature Reviews Genetics | volume = 7 | issue = 5 | pages = 337–48 | date = May 2006 | pmid = 16619049 | doi = 10.1038/nrg1838 | s2cid = 23225873 }}</ref>。遺伝的組換えはDNA修復、特に二本鎖切断に対する細胞の反応にも関与している可能性がある<ref>{{cite journal | vauthors = O'Driscoll M, Jeggo PA | title = The role of double-strand break repair – insights from human genetics | journal = Nature Reviews Genetics | volume = 7 | issue = 1 | pages = 45–54 | date = January 2006 | pmid = 16369571 | doi = 10.1038/nrg1746 | s2cid = 7779574 }}</ref>。 |
|||
染色体交差の最も一般的な形態は[[相同組換え]]で、関与する2つの染色体の配列は非常によく似ている。[[非相同組換え]]は、染色体転座や遺伝的異常を生じさせるため、細胞に損傷を与える可能性がある。組換え反応は、[[Rad51|RAD51]]のような{{Ill2|リコンビナーゼ|en|Recombinase}}として知られる酵素によって触媒される<ref>{{cite journal | vauthors = Vispé S, Defais M | title = Mammalian Rad51 protein: a RecA homologue with pleiotropic functions | journal = Biochimie | volume = 79 | issue = 9–10 | pages = 587–92 | date = October 1997 | pmid = 9466696 | doi = 10.1016/S0300-9084(97)82007-X }}</ref>。組換えの最初の段階は、[[エンドヌクレアーゼ]]かDNAの損傷によって引き起こされる二本鎖切断である<ref>{{cite journal | vauthors = Neale MJ, Keeney S | title = Clarifying the mechanics of DNA strand exchange in meiotic recombination | journal = Nature | volume = 442 | issue = 7099 | pages = 153–58 | date = July 2006 | pmid = 16838012 | doi = 10.1038/nature04885 | bibcode = 2006Natur.442..153N | pmc = 5607947 }}</ref>。その後、リコンビナーゼによって部分的に触媒される一連の段階によって、2つのらせんは少なくとも1つの[[ホリデイジャンクション]]によって結合され、次に、各らせん中の一本鎖セグメントが他方のらせんの相補鎖と二本鎖を形成する。ホリデイジャンクションは四面体の接合構造で、染色体対に沿って移動することができ、一方の鎖をもう一方の鎖と交換することができる。組換え反応は、結合部の切断と遊離したDNAの再結合によって停止する<ref>{{cite journal | vauthors = Dickman MJ, Ingleston SM, Sedelnikova SE, Rafferty JB, Lloyd RG, Grasby JA, Hornby DP | s2cid = 39505263 | title = The RuvABC resolvasome | journal = European Journal of Biochemistry | volume = 269 | issue = 22 | pages = 5492–501 | date = November 2002 | pmid = 12423347 | doi = 10.1046/j.1432-1033.2002.03250.x | doi-access = free }}</ref>。組換えの際に同じ{{ill2|方向性 (分子生物学)|en|Directionality (molecular biology)|label=方向性}}(極性)の鎖だけがDNAを交換する。切断には東西切断(east-west cleavage)と南北切断(north–south cleavage)の2種類がある。南北切断はDNAの両鎖を切断するが、東西切断はDNAの片鎖をそのまま残す。組換えの際にホリデイジャンクションが形成されることで、遺伝的多様性、染色体上での遺伝子の交換、および[[野生型]]ウイルスゲノムの発現が可能になる。 |
|||
== 進化 == |
|||
{{further|RNAワールド仮説}} |
|||
DNAにはあらゆる生命体が機能し、成長し、生殖するための遺伝情報が含まれている。しかし40億年の{{Ill2|生命の進化史年表|en|Timeline of the evolutionary history of life|label=生命の歴史}}の中で、DNAがいつからこの機能を果たしてきたかは不明である。最も初期の生命体はRNAを遺伝物質として使っていたのではないかという提案もある<ref name="autogenerated1">{{cite journal | vauthors = Joyce GF | title = The antiquity of RNA-based evolution | journal = Nature | volume = 418 | issue = 6894 | pages = 214–21 | date = July 2002 | pmid = 12110897 | doi = 10.1038/418214a | bibcode = 2002Natur.418..214J | s2cid = 4331004 }}</ref><ref>{{cite journal | vauthors = Orgel LE | title = Prebiotic chemistry and the origin of the RNA world | journal = Critical Reviews in Biochemistry and Molecular Biology | volume = 39 | issue = 2 | pages = 99–123 | year = 2004 | pmid = 15217990 | doi = 10.1080/10409230490460765 | citeseerx = 10.1.1.537.7679 | s2cid = 4939632 }}</ref>。RNAは遺伝情報の伝達と[[リボザイム]]の一部としての[[触媒作用]]の両方を行うことができるため、初期の[[細胞代謝]]において中心的な役割を果たしていた可能性がある<ref>{{cite journal | vauthors = Davenport RJ | s2cid = 85976762 | title = Ribozymes. Making copies in the RNA world | journal = Science | volume = 292 | issue = 5520 | pages = 1278a–1278 | date = May 2001 | pmid = 11360970 | doi = 10.1126/science.292.5520.1278a }}</ref>。核酸が触媒作用と遺伝学の両方に使われていたとする、この古代の[[RNAワールド]]は、4塩基に基づく現在の遺伝暗号の[[進化]]に影響を与えたかもしれない。このような生物における異なる塩基の数は、少ない塩基数による複製精度の向上と、多数の塩基によるリボザイムの触媒効率の向上との釣り合い関係によってきまった可能性もある<ref>{{cite journal | vauthors = Szathmáry E | title = What is the optimum size for the genetic alphabet? | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 89 | issue = 7 | pages = 2614–18 | date = April 1992 | pmid = 1372984 | pmc = 48712 | doi = 10.1073/pnas.89.7.2614 | bibcode = 1992PNAS...89.2614S | doi-access = free }}</ref>。しかしDNAは環境中で100万年未満しか存在できず、溶液中でゆっくりと短い断片に分解されるため、ほとんどの化石からDNAを回収することは不可能で、古代の遺伝子系の直接的な証拠はない<ref>{{cite journal | vauthors = Lindahl T | title = Instability and decay of the primary structure of DNA | journal = Nature | volume = 362 | issue = 6422 | pages = 709–15 | date = April 1993 | pmid = 8469282 | doi = 10.1038/362709a0 | bibcode = 1993Natur.362..709L | s2cid = 4283694 }}</ref>。より古いDNAが存在するという主張もなされており、特に2億5千万年前の塩の結晶から生存可能な細菌が分離されたという報告があるが<ref>{{cite journal | vauthors = Vreeland RH, Rosenzweig WD, Powers DW | title = Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal | journal = Nature | volume = 407 | issue = 6806 | pages = 897–900 | date = October 2000 | pmid = 11057666 | doi = 10.1038/35038060 | bibcode = 2000Natur.407..897V | s2cid = 9879073 }}</ref>、これらの主張には賛否がある<ref>{{cite journal | vauthors = Hebsgaard MB, Phillips MJ, Willerslev E | title = Geologically ancient DNA: fact or artefact? | journal = Trends in Microbiology | volume = 13 | issue = 5 | pages = 212–20 | date = May 2005 | pmid = 15866038 | doi = 10.1016/j.tim.2005.03.010 }}</ref><ref>{{cite journal | vauthors = Nickle DC, Learn GH, Rain MW, Mullins JI, Mittler JE | title = Curiously modern DNA for a "250 million-year-old" bacterium | journal = Journal of Molecular Evolution | volume = 54 | issue = 1 | pages = 134–37 | date = January 2002 | pmid = 11734907 | doi = 10.1007/s00239-001-0025-x | bibcode = 2002JMolE..54..134N | s2cid = 24740859 }}</ref>。 |
|||
DNAの構成要素([[アデニン]]、[[グアニン]]、および関連する[[有機分子]])は、地球外の[[宇宙空間]]で形成された可能性もある<ref name="Callahan">{{cite journal | vauthors = Callahan MP, Smith KE, Cleaves HJ, Ruzicka J, Stern JC, Glavin DP, House CH, Dworkin JP | title = Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 108 | issue = 34 | pages = 13995–98 | date = August 2011 | pmid = 21836052 | pmc = 3161613 | doi = 10.1073/pnas.1106493108 | bibcode = 2011PNAS..10813995C | doi-access = free }}</ref><ref name="Steigerwald">{{cite web | vauthors = Steigerwald J |title=NASA Researchers: DNA Building Blocks Can Be Made in Space |url=http://www.nasa.gov/topics/solarsystem/features/dna-meteorites.html |publisher=[[:en:NASA|NASA]] |date=8 August 2011 |access-date=10 August 2011 |url-status=live |archive-url=https://web.archive.org/web/20150623004556/http://www.nasa.gov/topics/solarsystem/features/dna-meteorites.html |archive-date=23 June 2015 }}</ref><ref name="DNA">{{cite web |author=ScienceDaily Staff |title=DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests |url=https://www.sciencedaily.com/releases/2011/08/110808220659.htm |date=9 August 2011 |website=[[:en:ScienceDaily|ScienceDaily]] |access-date=9 August 2011 |url-status=live |archive-url=https://web.archive.org/web/20110905105043/https://www.sciencedaily.com/releases/2011/08/110808220659.htm |archive-date=5 September 2011 }}</ref>。[[ウラシル]]、[[シトシン]]、[[チミン]]を含む、[[生命]]の複雑なDNAや[[リボ核酸|RNA]]の[[有機化合物]]もまた、[[隕石]]から発見された[[ピリミジン]]のような化学物質を出発点として、宇宙空間の模倣した条件下の実験室で合成されている。ピリミジンは、[[宇宙]]で発見された最も炭素を多く含む化学物質である[[多環芳香族炭化水素]](PAH)と同様、[[赤色巨星]]や星間[[宇宙塵]]やガス雲で形成された可能性がある<ref name="NASA-20150303">{{cite web | vauthors = Marlaire R |title=NASA Ames Reproduces the Building Blocks of Life in Laboratory |url=http://www.nasa.gov/content/nasa-ames-reproduces-the-building-blocks-of-life-in-laboratory |date=3 March 2015 |work=[[:en:NASA|NASA]] |access-date=5 March 2015 |url-status=live |archive-url=https://web.archive.org/web/20150305083306/http://www.nasa.gov/content/nasa-ames-reproduces-the-building-blocks-of-life-in-laboratory/ |archive-date=5 March 2015 }}</ref>。 |
|||
2021年2月、科学者たちは初めて、100万年以上前の[[マンモス]]象の[[腐肉|遺体]]からDNA[[DNAシークエンシング|配列]]を決定したことを報告した。これまでに塩基配列が決定された最古のDNAである<ref name="CNN-20210217">{{cite news | vauthors = Hunt K |title=World's oldest DNA sequenced from a mammoth that lived more than a million years ago |url=https://www.cnn.com/2021/02/17/world/mammoth-oldest-dna-million-years-ago-scn/index.html |date=17 February 2021 |work=[[:en:CNN|CNN News]] |access-date=17 February 2021 }}</ref><ref name="NAT-20210217">{{cite journal | vauthors = Callaway E | title = Million-year-old mammoth genomes shatter record for oldest ancient DNA – Permafrost-preserved teeth, up to 1.6 million years old, identify a new kind of mammoth in Siberia. |date=17 February 2021 |journal=[[:en:Nature (journal)|Nature]] |volume=590 |issue=7847 |pages=537–538 |doi=10.1038/d41586-021-00436-x |issn=0028-0836 |pmid=33597786 | bibcode = 2021Natur.590..537C |doi-access=free }}</ref>。 |
|||
== 技術における用途 == |
|||
=== 遺伝子工学 === |
|||
{{further|分子生物学|{{ill2|核酸法|en|Nucleic acid methods}}|遺伝子工学}} |
|||
{{Ill2|フェノール・クロロホルム抽出法|en|Phenol–chloroform extraction}}のように、生物からDNAを精製する方法や、{{Ill2|制限消化|en|Restriction digest}}や[[ポリメラーゼ連鎖反応]]のように実験室でDNAを操作する方法が開発された。現代の[[生物学]]や[[生化学]]では、組換えDNAの分野でこれらの技術を活用している。[[組換えDNA]]とは、他のDNA配列から組み立てられた人工のDNA配列である。これらは[[ウイルスベクター]]を利用して、[[プラスミド]]あるいは他の適切な型式で、生物に[[形質転換]]することができる<ref>{{cite journal | vauthors = Goff SP, Berg P | s2cid = 41788896 | title = Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells | journal = Cell | volume = 9 | issue = 4 PT 2 | pages = 695–705 | date = December 1976 | pmid = 189942 | doi = 10.1016/0092-8674(76)90133-1 }}</ref>。生産された[[遺伝子工学|遺伝子組換え]]生物は、[[組換えタンパク質]]のような製品を製造したり、{{Ill2|医学研究|en|Medical research}}で使用したり<ref>{{cite book | vauthors = Houdebine LM | title = Target Discovery and Validation Reviews and Protocols | chapter = Transgenic animal models in biomedical research | series = Methods in Molecular Biology | volume = 360 | pages = 163–202 | year = 2007 | pmid = 17172731 | doi = 10.1385/1-59745-165-7:163 | isbn = 978-1-59745-165-9 }}</ref>、[[農業]]で繁殖したりする<ref>{{cite journal | vauthors = Daniell H, Dhingra A | title = Multigene engineering: dawn of an exciting new era in biotechnology | journal = Current Opinion in Biotechnology | volume = 13 | issue = 2 | pages = 136–41 | date = April 2002 | pmid = 11950565 | pmc = 3481857 | doi = 10.1016/S0958-1669(02)00297-5 }}</ref><ref>{{cite journal | vauthors = Job D | title = Plant biotechnology in agriculture | journal = Biochimie | volume = 84 | issue = 11 | pages = 1105–10 | date = November 2002 | pmid = 12595138 | doi = 10.1016/S0300-9084(02)00013-5 }}</ref>。 |
|||
=== DNAプロファイリング === |
|||
{{further|DNAプロファイリング}} |
|||
[[法科学|法科学者]]は、{{Ill2|犯罪現場|en|Crime scene}}で発見された[[血液]]、[[精液]]、[[皮膚]]、[[唾液]]、または[[毛髪]]に含まれるDNAを利用して、加害者などの個人と一致するDNAを特定することができる<ref>{{Cite news|url=https://theconversation.com/from-the-crime-scene-to-the-courtroom-the-journey-of-a-dna-sample-82250|title=From the crime scene to the courtroom: the journey of a DNA sample| vauthors = Curtis C, Hereward J |date=29 August 2017 |work=The Conversation |access-date=22 October 2017 |archive-url=https://web.archive.org/web/20171022033110/http://theconversation.com/from-the-crime-scene-to-the-courtroom-the-journey-of-a-dna-sample-82250 |archive-date=22 October 2017 |url-status=live }}</ref>。この手法は正式には[[DNAプロファイリング]]({{Lang-en-short|DNA profiling}})と呼ばれ、DNA指紋法({{Lang-en-short|DNA fingerprinting}})とも呼ばれる。DNAプロファイリングでは、ショートタンデムリピート([[縦列型反復配列]])や{{Ill2|ミニサテライト|en|Minisatellite}}など、反復DNAの可変部分の長さを個人間で比較する。この方法は通常、一致するDNAを同定するための非常に信頼性の高い技術である<ref>{{cite journal | vauthors = Collins A, Morton NE | title = Likelihood ratios for DNA identification | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 91 | issue = 13 | pages = 6007–11 | date = June 1994 | pmid = 8016106 | pmc = 44126 | doi = 10.1073/pnas.91.13.6007 | bibcode = 1994PNAS...91.6007C | doi-access = free }}</ref>。ただし、現場が複数名のDNAで汚染されている場合、同定が複雑になることがある<ref>{{cite journal | vauthors = Weir BS, Triggs CM, Starling L, Stowell LI, Walsh KA, Buckleton J | title = Interpreting DNA mixtures | journal = Journal of Forensic Sciences | volume = 42 | issue = 2 | pages = 213–22 | date = March 1997 | doi = 10.1520/JFS14100J | pmid = 9068179 | s2cid = 14511630 | url = http://pdfs.semanticscholar.org/f47f/2c895d0b06b3dc72a4707b464126e6c820aa.pdf }}</ref>。DNAプロファイリングは1984年にイギリスの遺伝学者[[アレック・ジェフリーズ]]によって開発され<ref>{{cite journal | vauthors = Jeffreys AJ, Wilson V, Thein SL | title = Individual-specific 'fingerprints' of human DNA | journal = Nature | volume = 316 | issue = 6023 | pages = 76–79 | year = 1985 | pmid = 2989708 | doi = 10.1038/316076a0 | bibcode = 1985Natur.316...76J | s2cid = 4229883 | doi-access = free }}</ref>、1988年のエンダービー殺人事件で{{Ill2|コリン・ピッチフォーク|en|Colin Pitchfork}}を有罪にするために法科学で初めて使用された<ref>{{Cite web|date=2006-12-14|title=Colin Pitchfork|url=http://www.forensic.gov.uk/forensic_t/inside/news/list_casefiles.php?case=1|access-date=2023-03-27|archive-url=https://web.archive.org/web/20061214004903/http://www.forensic.gov.uk/forensic_t/inside/news/list_casefiles.php?case=1 |archive-date=14 December 2006 }}</ref>。 |
|||
法科学が発達し、血液、皮膚、唾液、毛髪などの微量サンプルで遺伝子照合ができるようになったことで、多くの事件が再調査されるようになった。当初の調査時には科学的に不可能であった証拠も、現在では発見されることがある。一部の地域において{{Ill2|二重の危険|en|Double jeopardy|label=二重の危険の原則|preserve=1}}({{Lang-en-short|double jeopardy law}})が撤廃されたこともあいまって、これまでの裁判で陪審を納得させるに十分な証拠が得られなかった事件でも再審が可能になることがある。重大犯罪で起訴された人々は、照合目的でDNAサンプルの提出を求められることがある。法科学的に得られたDNA照合に対する最も明白な抗弁は、証拠の相互汚染が起こったと主張することである。このため、重大犯罪の新事例に対し、細心の注意を払った厳格な取り扱い手順が導入されるようになった。 |
|||
DNAプロファイリングはまた、集団死傷事件の犠牲者<ref>{{cite web |url=http://massfatality.dna.gov/Introduction/ |title=DNA Identification in Mass Fatality Incidents |date=September 2006 |publisher=National Institute of Justice |url-status=dead |archive-url=https://web.archive.org/web/20061112000837/http://massfatality.dna.gov/Introduction/ |archive-date=12 November 2006 |access-date=2007-01-01}}</ref>、重大事故の遺体やその一部、集団戦没者墓地における犠牲者個人の身元を、家族との照合によって確認するためにも使用され、成功を収めている。 |
|||
DNAプロファイリングは、誰かが子供の生みの親または祖父母であるかどうかを判定するための[[親子鑑定|DNA親子鑑定]]にも使用され、親とされる人物が子供と生物学的に血縁関係がある場合、親である確率は通常99.99%である。通常の[[DNAシークエンシング|DNA配列決定]]法は出生後に行われるが、母親がまだ妊娠している間に親子関係を検査する新しい方法がある<ref>{{Cite news| vauthors = Pollack A |date=2012-06-19|title=Before Birth, Dad's ID|language=en-US|work=The New York Times|url=https://www.nytimes.com/2012/06/20/health/paternity-blood-tests-that-work-early-in-a-pregnancy.html|access-date=2023-03-27|issn=0362-4331|archive-url=https://web.archive.org/web/20170624231639/http://www.nytimes.com/2012/06/20/health/paternity-blood-tests-that-work-early-in-a-pregnancy.html|archive-date=2017-06-24|url-status=live}}</ref>。 |
|||
=== DNA酵素または触媒DNA === |
|||
{{further|{{ill2|デオキシリボザイム|en|Deoxyribozyme}}}} |
|||
{{ill2|デオキシリボザイム|en|Deoxyribozyme}}({{Lang-en-short|deoxyribozyme}})は、DNA酵素(DNAzymes)または[[触媒]]DNA(catalytic DNA)とも呼ばれ、1994年に初めて発見された<ref name="Breaker 223–229">{{cite journal | vauthors = Breaker RR, Joyce GF | title = A DNA enzyme that cleaves RNA | journal = Chemistry & Biology | volume = 1 | issue = 4 | pages = 223–29 | date = December 1994 | pmid = 9383394 | doi = 10.1016/1074-5521(94)90014-0 }}</ref>。これらの大部分は、''[[in vitro]]''選択法または{{Ill2|試験管内進化法|en|Systematic evolution of ligands by exponential enrichment}}({{Lang-en-short|Systematic Evolution of Ligands by Exponential Enrichment}}、SELEX)と呼ばれる組み合わせアプローチを使用して、ランダムなDNA配列の[[化学ライブラリー|大規模プール]]から単離された一本鎖DNA配列である。DNA酵素は、RNA-DNA切断、RNA-DNA{{Ill2|ライゲーション (分子生化学)|en|Ligation (molecular biology)|label=ライゲーション}}、アミノ酸のリン酸化-脱リン酸化、炭素-炭素結合形成など、さまざまな化学反応を触媒する。DNA酵素は、触媒反応の[[化学反応速度]]を、無触媒反応の最大1千億倍に向上させることができる<ref>{{cite journal | vauthors = Chandra M, Sachdeva A, Silverman SK | title = DNA-catalyzed sequence-specific hydrolysis of DNA | journal = Nature Chemical Biology | volume = 5 | issue = 10 | pages = 718–20 | date = October 2009 | pmid = 19684594 | pmc = 2746877 | doi = 10.1038/nchembio.201 }}</ref>。DNA酵素の中でもっとも広く研究されているのはRNA切断型で、さまざまな金属イオンの検出や治療薬の設計に使用されている。GR-5 DNA酵素(鉛特異的)<ref name="Breaker 223–229" />、CA1-3 DNA酵素(銅特異的)<ref>{{cite journal | vauthors = Carmi N, Shultz LA, Breaker RR | title = In vitro selection of self-cleaving DNAs | journal = Chemistry & Biology | volume = 3 | issue = 12 | pages = 1039–46 | date = December 1996 | pmid = 9000012 | doi = 10.1016/S1074-5521(96)90170-2 | doi-access = free }}</ref>、39E DNA酵素(ウラニル特異的)、NaA43 DNA酵素(ナトリウム特異的)<ref>{{cite journal | vauthors = Torabi SF, Wu P, McGhee CE, Chen L, Hwang K, Zheng N, Cheng J, Lu Y | title = In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 112 | issue = 19 | pages = 5903–08 | date = May 2015 | pmid = 25918425 | pmc = 4434688 | doi = 10.1073/pnas.1420361112 | bibcode = 2015PNAS..112.5903T | doi-access = free }}</ref>など、いくつかの金属特異的DNA酵素が報告されている。NaA43 DNA酵素は、ナトリウムに対して他の金属イオンよりも10,000倍以上選択的であると報告されており、細胞内でリアルタイムのナトリウムセンサーを作成するために使用された。 |
|||
=== バイオインフォマティクス === |
|||
{{further|バイオインフォマティクス}} |
|||
[[バイオインフォマティクス]]は、DNA[[核酸配列]]データを含む生物学的データの保存、[[データマイニング]]、検索、操作のための技術開発を含む学問分野である。これらの技術は、[[コンピュータサイエンス]]、特に{{Ill2|文字列検索|en|String-searching algorithm|label=文字列検索アルゴリズム|preserve=1}}、[[機械学習]]、{{Ill2|データベース理論|en|Database theory}}に広く応用されるようになった<ref>{{cite book | vauthors = Baldi P, Brunak S |author1-link=Pierre Baldi |title=Bioinformatics: The Machine Learning Approach |publisher= MIT Press |year=2001| isbn=978-0-262-02506-5 |oclc=45951728}}</ref>。文字列検索またはマッチングアルゴリズムは、より大きな文字列の中にある文字列の出現を検出する手法で、ヌクレオチドの特異的配列を検索するために開発された<ref>{{cite book | vauthors = Gusfield D | title = Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology | publisher = [[:en:Cambridge University Press|Cambridge University Press]] | date = 15 January 1997 | isbn = 978-0-521-58519-4 }}</ref>。DNA配列を他のDNA配列と[[シーケンスアラインメント|整列]]させることで、{{Ill2|配列相同性|en|Sequence homology|label=相同配列}}を同定し、それらを区別する特異的[[変異]]を突き止めることができる。これらの技術、特に[[多重配列アラインメント]]は、[[系統学|系統的]]関係やタンパク質機能を研究する際に使用される<ref>{{cite journal | vauthors = Sjölander K | title = Phylogenomic inference of protein molecular function: advances and challenges | journal = Bioinformatics | volume = 20 | issue = 2 | pages = 170–79 | date = January 2004 | pmid = 14734307 | doi = 10.1093/bioinformatics/bth021 | citeseerx = 10.1.1.412.943 }}</ref>。[[ヒトゲノムプロジェクト]]で作成されたような全ゲノムDNA配列の大規模なデータセットは、各染色体上の遺伝子や調節エレメントの位置を特定するアノテーションがなくては利用が困難である。タンパク質やRNAをコードする遺伝子に関連する特徴的なパターンを持つDNA配列領域は、{{Ill2|遺伝子探索|en|Gene prediction}}アルゴリズムによって同定することができ、これにより研究者は、特定の[[遺伝子産物]]が実験的に単離される前であっても、生物内での存在と可能性のある機能を予測することができる<ref name="Mount">{{cite book | vauthors = Mount DM |title=Bioinformatics: Sequence and Genome Analysis |edition= 2nd |publisher= Cold Spring Harbor Laboratory Press |year= 2004 |isbn= 0-87969-712-1|oclc= 55106399|location=Cold Spring Harbor, NY}}</ref>。また、ゲノム全体を比較することで、生物の進化の歴史に焦点を当てたり、複雑な進化の過程を研究することもできる。 |
|||
=== DNAナノテクノロジー === |
|||
[[File:DNA nanostructures.png|thumb|upright=1.8|左側のDNA構造 (模式図) は、右側の[[原子間力顕微鏡]]で視覚化された構造に自己集合する。[[DNAナノテクノロジー]]は、DNA分子の[[分子認識]]特性を利用してナノスケール構造を設計しようとする分野である<ref>{{cite journal | vauthors = Strong M | title = Protein nanomachines | journal = PLOS Biology | volume = 2 | issue = 3 | pages = E73 | date = March 2004 | pmid = 15024422 | pmc = 368168 | doi = 10.1371/journal.pbio.0020073 | s2cid = 13222080 | doi-access = free }}</ref>。]] |
|||
{{further|DNAナノテクノロジー}} |
|||
[[DNAナノテクノロジー]]は、DNAや他の核酸に特有の[[分子認識]]特性を利用して、有用な特性を備えた自己集合化能・分岐DNA複合体<!-- self-assembling branched DNA complexes -->を作り出す技術領域である<ref>{{cite journal | vauthors = Rothemund PW | s2cid = 4316391 | title = Folding DNA to create nanoscale shapes and patterns | journal = Nature | volume = 440 | issue = 7082 | pages = 297–302 | date = March 2006 | pmid = 16541064 | doi = 10.1038/nature04586 | bibcode = 2006Natur.440..297R | url = https://authors.library.caltech.edu/22244/3/nature04586-s2.pdf }}</ref>。DNAは生物学的情報の伝達手段としてではなく、構造材料として使用することもできる。その結果、2次元周期格子(タイルベースと[[DNAオリガミ|DNAオリガミ法]]の両方)や、[[多面体]]形状を持つ3次元構造の創造につながった<ref>{{cite journal | vauthors = Andersen ES, Dong M, Nielsen MM, Jahn K, Subramani R, Mamdouh W, Golas MM, Sander B, Stark H, Oliveira CL, Pedersen JS, Birkedal V, Besenbacher F, Gothelf KV, Kjems J | s2cid = 4430815 | title = Self-assembly of a nanoscale DNA box with a controllable lid | journal = Nature | volume = 459 | issue = 7243 | pages = 73–76 | date = May 2009 | pmid = 19424153 | doi = 10.1038/nature07971 | bibcode = 2009Natur.459...73A | hdl = 11858/00-001M-0000-0010-9362-B | hdl-access = free }}</ref>。{{Ill2|DNAマシン|en|DNA machine|label=ナノメカニカルデバイス}}や[[DNAコンピュータ|アルゴリズム的自己集合化]]も実証されており<ref>{{cite journal | vauthors = Ishitsuka Y, Ha T | title = DNA nanotechnology: a nanomachine goes live | journal = Nature Nanotechnology | volume = 4 | issue = 5 | pages = 281–82 | date = May 2009 | pmid = 19421208 | doi = 10.1038/nnano.2009.101 | bibcode = 2009NatNa...4..281I }}</ref>、これらのDNA構造は、[[金コロイド|金ナノ粒子]]や[[ストレプトアビジン]]タンパク質など、他の分子集合体の鋳型とするために使用されている<ref>{{cite journal | vauthors = Aldaye FA, Palmer AL, Sleiman HF | title = Assembling materials with DNA as the guide | journal = Science | volume = 321 | issue = 5897 | pages = 1795–99 | date = September 2008 | pmid = 18818351 | doi = 10.1126/science.1154533 | bibcode = 2008Sci...321.1795A | s2cid = 2755388 }}</ref>。DNAや他の核酸は、[[アプタマー]](さまざまなバイオテクノロジーや生物医学の用途に使われる、特定の標的分子に対する合成オリゴヌクレオチドリガンド)の基礎となっている<ref>{{cite journal | vauthors = Dunn MR, Jimenez RM, Chaput JC |title=Analysis of aptamer discovery and technology |journal=Nature Reviews Chemistry |date=2017 |volume=1 |issue=10 |doi=10.1038/s41570-017-0076 |url=https://www.nature.com/articles/s41570-017-0076 |access-date=30 June 2022}}</ref>。 |
|||
=== 系統学と人類学 === |
|||
{{further|系統学|遺伝子系図}} |
|||
DNAは時間の経過とともに変異を蓄積し、遺伝によって歴史的な情報を含んでおり、DNAの塩基配列を比較することで、遺伝学者は生物の進化の歴史、[[系統発生]]を推定することができる<ref>{{cite journal | vauthors = Wray GA | title = Dating branches on the tree of life using DNA | journal = Genome Biology | volume = 3 | issue = 1 | pages = REVIEWS0001 | year = 2002 | pmid = 11806830 | pmc = 150454 | doi = 10.1186/gb-2001-3-1-reviews0001 | doi-access = free }}</ref>。系統発生学は[[進化生物学]]における強力な道具である。生物種内のDNA配列を比較することで、[[集団遺伝学|集団遺伝学者]]は特定の集団の歴史を知ることができる。これは、{{Ill2|生態遺伝学|en|Ecological genetics}}から[[人類学]]に至るまで、さまざまな研究に利用できる。 |
|||
=== 情報ストレージ === |
|||
{{Main|{{ill2|DNAデジタルデータストレージ|en|DNA digital data storage}}}} |
|||
{{Ill2|データストレージ|en|Data storage|preserve=1|label=情報記録媒体}}としてのDNAは、電子機器に比べて{{Ill2|密度 (コンピュータ)|en|Density (computer storage)|label=記録密度}}がはるかに高いため、非常に大きな可能性を秘めている。しかしコストが高く、読み書きに時間がかかり([[メモリ遅延|メモリレイテンシ]])、{{Ill2|データ破壊|en|Data corruption|label=信頼性}}が十分でないことなどから、実用化には至っていない<ref name="pmid29744271">{{cite journal | vauthors = Panda D, Molla KA, Baig MJ, Swain A, Behera D, Dash M | title = DNA as a digital information storage device: hope or hype? | journal = 3 Biotech | volume = 8 | issue = 5 | pages = 239 | date = May 2018 | pmid = 29744271 | doi = 10.1007/s13205-018-1246-7 | pmc=5935598}}</ref><ref name="pmid30073589">{{cite journal | vauthors = Akram F, Haq IU, Ali H, Laghari AT | s2cid = 51905843 | title = Trends to store digital data in DNA: an overview | journal = Molecular Biology Reports | volume = 45 | issue = 5 | pages = 1479–1490 | date = October 2018 | pmid = 30073589 | doi = 10.1007/s11033-018-4280-y }}</ref>。 |
|||
== 歴史 == |
|||
{{anchors|History of DNA research}} |
|||
{{further|分子生物学の歴史}} |
|||
[[File:Maclyn McCarty with Francis Crick and James D Watson - 10.1371 journal.pbio.0030341.g001-O.jpg|thumb|[[マクリン・マッカーティ]]と握手する[[フランシス・クリック]]と[[ジェームズ・ワトソン]]]] |
|||
[[File:Pencil sketch of the DNA double helix by Francis Crick Wellcome L0051225.jpg|thumb|upright|フランシス・クリックによるDNA二重らせんの鉛筆スケッチ (1953年)]] |
|||
DNAが最初に単離されたのは、1869年、スイスの医師[[フリードリッヒ・ミーシェル]]によって、廃棄された手術用包帯の[[膿]](うみ)の中から微小な物質を発見した時にさかのぼる。細胞核に存在することから、彼はこれを「ヌクレイン({{Lang-en-short|nuclein}})」と命名した<ref>{{cite journal | vauthors = Miescher F | year = 1871 | url = https://books.google.com/books?id=YJRTAAAAcAAJ&pg=PA441 | title = Ueber die chemische Zusammensetzung der Eiterzellen | language = de | trans-title = On the chemical composition of pus cells | journal = Medicinisch-chemische Untersuchungen | volume = 4 | pages = 441–60 | quote = [p. 456] ''Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend.'' (Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ("Nuclein"))}}</ref><ref>{{cite journal | vauthors = Dahm R | s2cid = 915930 | title = Discovering DNA: Friedrich Miescher and the early years of nucleic acid research | journal = Human Genetics | volume = 122 | issue = 6 | pages = 565–81 | date = January 2008 | pmid = 17901982 | doi = 10.1007/s00439-007-0433-0 }}</ref>。1878年、[[アルブレヒト・コッセル]]が「ヌクレイン」の非タンパク質成分である核酸を単離し、その後、5つの標準[[核酸塩基]]<!-- primary nucleobases -->を単離した<ref>See: |
|||
*{{cite journal | vauthors = Kossel A | year = 1879 | url = https://books.google.com/books?id=4H5NAAAAYAAJ&pg=PA284 | title = Ueber Nucleïn der Hefe | language = de| trans-title = On nuclein in yeast | journal = Zeitschrift für physiologische Chemie | volume = 3 | pages = 284–91 }} |
|||
*{{cite journal | vauthors = Kossel A | year = 1880 | url = https://books.google.com/books?id=u4s1AQAAMAAJ&pg=PA290 | title = Ueber Nucleïn der Hefe II | language = de | trans-title = On nuclein in yeast, Part 2 | journal = Zeitschrift für physiologische Chemie | volume = 4 | pages = 290–95 }} |
|||
*{{cite journal | vauthors = Kossel A | year = 1881 | url = https://books.google.com/books?id=xYs1AQAAMAAJ&pg=PA267 | title = Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich | language = de | trans-title = On the distribution of hypoxanthins in the animal and plant kingdoms | journal = Zeitschrift für physiologische Chemie | volume = 5 | pages = 267–71 }} |
|||
*{{cite book | vauthors = Kossel A | title = Untersuchungen über die Nucleine und ihre Spaltungsprodukte | language = de | trans-title = Investigations into nuclein and its cleavage products | location = Strassburg, Germany | veditors = Trübne KJ | year = 1881 | pages = 19 }} |
|||
*{{cite journal | vauthors = Kossel A | year = 1882 | url = https://books.google.com/books?id=z4s1AQAAMAAJ&pg=PA422 | title = Ueber Xanthin und Hypoxanthin |trans-title= On xanthin and hypoxanthin | journal = Zeitschrift für physiologische Chemie | volume = 6 | pages = 422–31 }} |
|||
*Albrect Kossel (1883) [https://books.google.com/books?id=2os1AQAAMAAJ&pg=PA7 "Zur Chemie des Zellkerns"] {{webarchive|url=https://web.archive.org/web/20171117235430/https://books.google.com/books?id=2os1AQAAMAAJ&pg=PA7 |date=17 November 2017 }} (On the chemistry of the cell nucleus), ''Zeitschrift für physiologische Chemie'', '''7''': 7–22. |
|||
*{{cite journal | vauthors = Kossel A | year = 1886 | title = Weitere Beiträge zur Chemie des Zellkerns | language = de | trans-title = Further contributions to the chemistry of the cell nucleus | journal = Zeitschrift für Physiologische Chemie | volume = 10 | pages = 248–64 | url = http://vlp.mpiwg-berlin.mpg.de/library/data/lit16615/index_html?pn=1&ws=1.5 | quote = On p. 264, Kossel remarked presciently: Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge. (The study of the quantitative relations of the four nitrogenous bases—[and] of the dependence of their quantity on the physiological states of the cell—promises important insights into the fundamental physiological-chemical processes.) }}</ref><ref name="Yale_Jones_1953">{{cite journal | vauthors = Jones ME | title = Albrecht Kossel, a biographical sketch | journal = The Yale Journal of Biology and Medicine | volume = 26 | issue = 1 | pages = 80–97 | date = September 1953 | pmid = 13103145 | pmc = 2599350 }}</ref>。 |
|||
1909年、[[フィーバス・レヴィーン]]はRNA(当時は「酵母核酸({{Lang-en-short|yeast nucleic acid}})」と呼んだ)の塩基、糖、リン酸の[[ヌクレオチド]]単位を同定した<ref>{{cite journal | vauthors = Levene PA, Jacobs WA | year = 1909 | title = Über Inosinsäure | language = de | journal = Berichte der Deutschen Chemischen Gesellschaft | volume = 42 | pages = 1198–203 |url=https://babel.hathitrust.org/cgi/pt?id=iau.31858002459620&view=1up&seq=1054 | doi=10.1002/cber.190904201196}}</ref><ref>{{cite journal | vauthors = Levene PA, Jacobs WA | year = 1909 | title = Über die Hefe-Nucleinsäure | language = de | journal = Berichte der Deutschen Chemischen Gesellschaft | volume = 42 | issue = 2 | pages = 2474–78 | doi=10.1002/cber.190904202148| url = https://zenodo.org/record/2175598 }}</ref><ref>{{cite journal | vauthors = Levene P |title=The structure of yeast nucleic acid| journal=J Biol Chem |volume=40 |issue=2 |pages=415–24 |year=1919|doi=10.1016/S0021-9258(18)87254-4|doi-access=free }}</ref>。1929年、レヴィーンはDNA(当時は「胸腺核酸({{Lang-en-short|thymus nucleic acid}})」)内のデオキシリボース糖を同定した<ref>{{cite journal | vauthors = Cohen JS, Portugal FH | year = 1974 | title = The search for the chemical structure of DNA | journal = Connecticut Medicine | volume = 38 | issue = 10 | pages = 551–52, 554–57 | pmid = 4609088 | url = https://profiles.nlm.nih.gov/ps/access/CCAAHW.pdf }}</ref>。レヴィーンは、DNAはリン酸基によって結合された4つのヌクレオチド単位からなる紐で構成されていることを提案した({{Ill|テトラヌクレオチド仮説|en|Tetranucleotide hypothesis}})。レヴィーンは、この鎖は短く、塩基が一定の順序で繰り返されていると考えた。1927年、{{Ill2|ニコライ・コルツォフ|en|Nikolai Koltsov}}は、遺伝形質は「それぞれの鎖を鋳型として半保存的に複製される2本の鏡像鎖」からなる「巨大な遺伝分子」を介して遺伝すると提案した<ref>Koltsov proposed that a cell's genetic information was encoded in a long chain of amino acids. See: |
|||
*{{cite speech | vauthors = Koltsov HK | title = Физико-химические основы морфологии | trans-title = The physical-chemical basis of morphology | language = ru | event = 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists | location = Leningrad, U.S.S.R. | date = 12 December 1927 }} |
|||
*Reprinted in: {{cite journal | vauthors = Koltsov HK | title = Физико-химические основы морфологии | trans-title = The physical-chemical basis of morphology | language = ru| journal = Успехи экспериментальной биологии (Advances in Experimental Biology) series B | volume = 7 | issue = 1 | pages = ? | date = 1928 }} |
|||
*Reprinted in German as: {{cite journal | vauthors = Koltzoff NK | date = 1928 | title = Physikalisch-chemische Grundlagen der Morphologie | trans-title = The physical-chemical basis of morphology | language = de | journal = Biologisches Zentralblatt | volume = 48 | issue = 6 | pages = 345–69 }} |
|||
*In 1934, Koltsov contended that the proteins that contain a cell's genetic information replicate. See: {{cite journal | vauthors = Koltzoff N | title = The structure of the chromosomes in the salivary glands of Drosophila | journal = Science | volume = 80 | issue = 2075 | pages = 312–13 | date = October 1934 | pmid = 17769043 | doi = 10.1126/science.80.2075.312 | quote = From page 313: "I think that the size of the chromosomes in the salivary glands [of Drosophila] is determined through the multiplication of ''genonemes''. By this term I designate the axial thread of the chromosome, in which the geneticists locate the linear combination of genes; … In the normal chromosome there is usually only one genoneme; before cell-division this genoneme has become divided into two strands."| bibcode = 1934Sci....80..312K }}</ref><ref name="Soyfer">{{cite journal | vauthors = Soyfer VN | s2cid = 46277758 | title = The consequences of political dictatorship for Russian science | journal = Nature Reviews Genetics | volume = 2 | issue = 9 | pages = 723–29 | date = September 2001 | pmid = 11533721 | doi = 10.1038/35088598 }}</ref>。1928年、[[フレデリック・グリフィス]]は[[グリフィスの実験|実験]]によって、[[肺炎球菌]]({{Snamei|Pneumococcus}})のS型菌の[[形質]]が、死滅したS型菌と生きたR型菌とを混合することによって、R型菌に転換できることを発見した([[グリフィスの実験]])<ref>{{cite journal | vauthors = Griffith F | title = The Significance of Pneumococcal Types | journal = The Journal of Hygiene | volume = 27 | issue = 2 | pages = 113–59 | date = January 1928 | pmid = 20474956 | pmc = 2167760 | doi = 10.1017/S0022172400031879 }}</ref><ref>{{cite journal | vauthors = Lorenz MG, Wackernagel W | title = Bacterial gene transfer by natural genetic transformation in the environment | journal = Microbiological Reviews | volume = 58 | issue = 3 | pages = 563–602 | date = September 1994 | pmid = 7968924 | pmc = 372978 | doi = 10.1128/MMBR.58.3.563-602.1994 }}</ref>。この実験系は、DNAが遺伝情報を伝達していることを初めて明確に示唆した。 |
|||
1933年、[[ウニ]]の未受精卵を研究していた{{Ill2|ジャン・ブラッシェ|en|Jean Brachet}}(Jean Brachet)は、DNAは[[細胞核]]に存在し、[[リボ核酸|RNA]]は[[細胞質]]にのみ存在することを提案した。当時は、酵母核酸(RNA)は植物だけに、胸腺核酸(DNA)は動物だけに存在すると考えられていた。後者は細胞内pHを緩衝する機能を持つ[[四量体]]であると考えられていた<ref>{{cite journal | vauthors = Brachet J | year = 1933 | title = Recherches sur la synthese de l'acide thymonucleique pendant le developpement de l'oeuf d'Oursin | language = it | journal = Archives de Biologie | volume = 44 | pages = 519–76 }}</ref><ref>{{cite book | vauthors = Burian R | year = 1994 | chapter = Jean Brachet's Cytochemical Embryology: Connections with the Renovation of Biology in France? | veditors = Debru C, Gayon J, Picard JF | title = Les sciences biologiques et médicales en France 1920–1950 | volume = 2 | series = Cahiers pour I'histoire de la recherche | location = Paris | publisher = CNRS Editions | pages = 207–20 | chapter-url = http://www.histcnrs.fr/ColloqDijon/Burian-Brachet.pdf }}</ref>。 |
|||
1937年、[[ウィリアム・アストベリー]]は、DNAが規則正しい構造を持っていることを示すX線回折パターンを初めて作成した<ref>See: |
|||
*{{cite journal | vauthors = Astbury WT, Bell FO | year = 1938 | title = Some recent developments in the X-ray study of proteins and related structures | journal = Cold Spring Harbor Symposia on Quantitative Biology | volume = 6 | pages = 109–21 | url = http://www.leeds.ac.uk/heritage/Astbury/bibliography/CSHSQB_Astbury_and_Bell_1938.pdf | archive-url = https://web.archive.org/web/20140714204539/http://www.leeds.ac.uk/heritage/Astbury/bibliography/CSHSQB_Astbury_and_Bell_1938.pdf | archive-date = 14 July 2014 | doi=10.1101/sqb.1938.006.01.013}} |
|||
*{{cite journal | vauthors = Astbury WT | title = X-ray studies of nucleic acids | journal = Symposia of the Society for Experimental Biology | issue = 1 | pages = 66–76 | year = 1947 | pmid = 20257017 | url = http://scarc.library.oregonstate.edu/coll/pauling/dna/papers/astbury-xray.html | archive-url = https://web.archive.org/web/20140705132403/http://scarc.library.oregonstate.edu/coll/pauling/dna/papers/astbury-xray.html | archive-date=5 July 2014 }}</ref>。 |
|||
1943年、[[オズワルド・アベリー]]は、共同研究者である{{Ill2|コリン・ムンロ・マクロード|en|Colin Munro MacLeod|label=コリン・マクロード}}、[[マクリン・マッカーティ]]とともに、DNAが[[グリフィスの実験|形質転換原理]]であることを突き止め、グリフィスの提案を支持した([[アベリー-マクロード-マッカーティの実験]])<ref>{{cite journal | vauthors = Avery OT, Macleod CM, McCarty M | title = Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III | journal = The Journal of Experimental Medicine | volume = 79 | issue = 2 | pages = 137–158 | date = February 1944 | pmid = 19871359 | pmc = 2135445 | doi = 10.1084/jem.79.2.137 }}</ref>。[[エルヴィン・シャルガフ]]は、現在「シャルガフの法則」として知られる見解を発表し、どの生物種のDNAにおいても、グアニンの量はシトシンと等しく、アデニンの量はチミンと等しくなければならないと述べた<ref name="chargaff_1950">{{cite journal | vauthors = Chargaff E | title = Chemical specificity of nucleic acids and mechanism of their enzymatic degradation | journal = Experientia | volume = 6 | issue = 6 | pages = 201–209 | date = June 1950 | pmid = 15421335 | doi = 10.1007/BF02173653 | s2cid = 2522535 }}</ref><ref name="kresge_2005">{{cite journal | vauthors = Kresge N, Simoni RD, Hill RL |title=Chargaff's Rules: the Work of Erwin Chargaff |journal=Journal of Biological Chemistry |date=June 2005 |volume=280 |issue=24 |pages=172–174 |doi=10.1016/S0021-9258(20)61522-8|doi-access=free }}</ref>。 |
|||
[[File:TheEaglePub-Cambridge-BluePlaque.jpg|thumb|left|{{Ill2|ザ・イーグル (ケンブリッジ)|en|The Eagle, Cambridge|label=ザ・イーグル}}パブの外に掲げられたクリックとワトソンを記念する[[ブルー・プラーク]]]] |
|||
1951年末、[[フランシス・クリック]]は、英国[[ケンブリッジ大学]]の[[キャヴェンディッシュ研究所]]で[[ジェームズ・ワトソン]]とともに研究を始めた。[[遺伝]]におけるDNAの役割は、1952年に[[アルフレッド・ハーシー]]と[[マーサ・チェイス]]が行った一連の実験([[ハーシーとチェイスの実験|ハーシー-チェイス実験]])で、DNAが{{Ill2|腸内細菌ファージT2|en|Enterobacteria phage T2}}の[[遺伝物質]]であることを示して確認された<ref>{{cite journal | vauthors = Hershey AD, Chase M | title = Independent functions of viral protein and nucleic acid in growth of bacteriophage | journal = The Journal of General Physiology | volume = 36 | issue = 1 | pages = 39–56 | date = May 1952 | pmid = 12981234 | pmc = 2147348 | doi = 10.1085/jgp.36.1.39 }}</ref>。 |
|||
1952年5月、[[ロザリンド・フランクリン]]の指導下で研究をしていた大学院生、{{Ill2|レイモンド・ゴスリング|en|Raymond Gosling}}は、高水和レベルでのDNA [[X線回折]]像を撮影し、「{{Ill2|Photo 51|en|Photo 51}}」とラベルを付けた<ref>{{Cite web|title=Pictures and Illustrations: Crystallographic photo of Sodium Thymonucleate, Type B. "Photo 51." May 1952|url=http://scarc.library.oregonstate.edu/coll/pauling/dna/pictures/sci9.001.5.html|access-date=2023-05-18|website=scarc.library.oregonstate.edu}}</ref>。この写真は、[[モーリス・ウィルキンス]]からワトソンとクリックに渡されたもので、彼らがDNAの正しい構造を得る上で極めて重要なものであった。フランクリンはクリックとワトソンに、主鎖は外側になければならないと語った。それまでは、ライナス・ポーリングや、ワトソンとクリックらは、鎖が内側にあって塩基が外側を向いた誤ったモデルを持っていた。フランクリンがDNA結晶の[[空間群]]を特定したことで、クリックは、DNAの二本鎖が[[逆平行 (生化学)|逆平行]]であることを突き止めた<ref name="ReferenceA">{{cite book| vauthors = Schwartz J |title=In pursuit of the gene: from Darwin to DNA |url= https://archive.org/details/inpursuitofgenef00schw |url-access=registration |year=2008|publisher=Harvard University Press|location=Cambridge, Mass.|isbn=978-0-674-02670-4 }}</ref>。1953年2月、[[ライナス・ポーリング]]と[[ロバート・コリー]]は、リン酸が軸の近くにあり、塩基が外側にある、3本の鎖が絡み合った核酸のモデルを提案した。ワトソンとクリックはそのモデルを完成させ、現在では{{Ill2|核酸の分子構造: デオキシリボース核酸の構造|en|Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid|label=DNA二重らせん}}の最初の正しいモデルとして受け入れられている<ref name="pmid16578429">{{cite journal | vauthors = Pauling L, Corey RB | title = A Proposed Structure For The Nucleic Acids | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 39 | issue = 2 | pages = 84–97 | date = February 1953 | pmid = 16578429 | pmc = 1063734 | doi = 10.1073/pnas.39.2.84| url = http://scarc.library.oregonstate.edu/coll/pauling/dna/papers/1953p.9-084.html | bibcode = 1953PNAS...39...84P | doi-access = free }}</ref>。1953年2月28日、クリックは、英国ケンブリッジの{{Ill2|ザ・イーグル (ケンブリッジ)|en|The Eagle, Cambridge|label=ザ・イーグル}}パブで常連客のランチタイムを中断し、彼とワトソンが「生命の秘密を発見した」と発表した<ref>{{cite book | vauthors = Regis E | date = 2009 | title = What Is Life?: investigating the nature of life in the age of synthetic biology | location = Oxford | publisher = [[:en:Oxford University Press|Oxford University Press]] | isbn = 978-0-19-538341-6 | page = 52 }}</ref>。 |
|||
1953年4月25日、雑誌「[[Nature (journal)|''Nature'']]」は、ワトソンとクリックの二重らせん構造DNAとそれを支持する証拠を示す一連の5本の論文を掲載した<ref name="NatureDNA50">{{cite web |work=Nature Archives |url=http://www.nature.com/nature/dna50/archive.html |title=Double Helix of DNA: 50 Years |archive-url=https://web.archive.org/web/20150405140401/http://www.nature.com/nature/dna50/archive.html |archive-date=5 April 2015 |url-status=dead |access-date=2007-02-15}}</ref>。その構造は、『''MOLECULAR STRUCTURE OF NUCLEIC ACIDS A Structure for Deoxyribose Nucleic Acid''({{Ill2|核酸の分子構造: デオキシリボース核酸の構造|en|Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid}})』と題されたレターで報告され、その中で彼らは次のように述べている。『私たちが仮定した特異的な対形成が、遺伝物質の複製メカニズムである可能性を即座に示唆していることを、私たちは見逃さなかった<ref name="autogenerated2" />』。この後、フランクリンとゴスリングのレターが続き、彼ら自身のX線回折データと独自の解析方法が初めて公表された<ref name="NatFranGos" /><ref>{{cite web | url = http://osulibrary.oregonstate.edu/specialcollections/coll/pauling/dna/pictures/franklin-typeBphoto.html | title = Original X-ray diffraction image | publisher = Oregon State Library | access-date = 6 February 2011 | url-status=live | archive-url = https://web.archive.org/web/20090130111849/http://osulibrary.oregonstate.edu/specialcollections/coll/pauling/dna/pictures/franklin-typeBphoto.html | archive-date=30 January 2009 }}</ref>。さらに、ウィルキンスと彼の同僚2名によるレターが続き、生体内におけるB-DNA X線パターンの解析が報告されており、生体内にワトソンとクリックの構造が存在することを裏付けていた<ref name="NatWilk" />。 |
|||
1962年、フランクリンの死後、ワトソン、クリック、ウィルキンスの3名は[[ノーベル生理学・医学賞]]を共同受賞した<ref>{{cite web |url=http://nobelprize.org/nobel_prizes/medicine/laureates/1962/ |title=The Nobel Prize in Physiology or Medicine 1962 |work=Nobelprize.org |access-date=2006-12-24}}</ref>。ノーベル賞は存命中の受賞者にのみ授与される。2023年4月、科学者たちは新たな証拠に基づき、ロザリンド・フランクリンはDNA発見の過程に貢献しただけでなく「対等な役割」を果たした人物であり、発見後に発表されたような貢献者ではないと結論づけた<ref name="AP-20230425">{{cite news | vauthors = Burakoff M |title=Rosalind Franklin's role in DNA discovery gets a new twist |url=https://apnews.com/article/dna-double-helix-rosalind-franklin-watson-crick-69ec8164c720e0b23374da69a1d3708d |date=25 April 2023 |work=[[:en:AP News|AP News]] |accessdate=25 April 2023 }}</ref><ref name="NYT-20230425">{{cite news | vauthors = Anthes E |title=Untangling Rosalind Franklin's Role in DNA Discovery, 70 Years On – Historians have long debated the role that Dr. Franklin played in identifying the double helix. A new opinion essay argues that she was an "equal contributor." |url=https://www.nytimes.com/2023/04/25/science/rosalind-franklin-dna.html |date=25 April 2023 |work=[[:en:The New York Times|The New York Times]] |url-status=live |archiveurl=https://archive.today/20230425182515/https://www.nytimes.com/2023/04/25/science/rosalind-franklin-dna.html |archivedate=25 April 2023 |accessdate=26 April 2023 }}</ref><ref name="NAT-202304254">{{cite journal | vauthors = Cobb M, Comfort N |title=What Rosalind Franklin truly contributed to the discovery of DNA's structure – Franklin was no victim in how the DNA double helix was solved. An overlooked letter and an unpublished news article, both written in 1953, reveal that she was an equal player. |date=25 April 2023 |journal=[[:en:Nature (journal)|Nature]] |volume=616 |issue=7958 |pages=657–660 |doi=10.1038/d41586-023-01313-5 |pmid=37100935 |s2cid=258314143 |doi-access=free }}</ref>。誰がこの発見の功績を称えられるべきかについては議論が続いている<ref>{{cite journal | vauthors = Maddox B | s2cid = 4428347 | title = The double helix and the 'wronged heroine' | journal = Nature | volume = 421 | issue = 6921 | pages = 407–08 | date = January 2003 | pmid = 12540909 | doi = 10.1038/nature01399 | url = http://www.biomath.nyu.edu/index/course/hw_articles/nature4.pdf | bibcode = 2003Natur.421..407M | url-status=live | archive-url = https://web.archive.org/web/20161017011403/http://www.biomath.nyu.edu/index/course/hw_articles/nature4.pdf | archive-date = 17 October 2016 | df = dmy-all | doi-access = free }}</ref>。 |
|||
1957年に行われた影響力のある講演で、クリックは、[[セントラルドグマ|分子生物学におけるセントラル・ドグマ]]を打ち出し、DNA、RNA、タンパク質の関係を予言し、「{{Ill2|アダプター仮説|en|Adaptor hypothesis}}」を公にした<ref>{{cite speech | vauthors = Crick FH |title=A Note for the RNA Tie Club | date= 1955 | location = Cambridge, England |url= http://genome.wellcome.ac.uk/assets/wtx030893.pdf | archive-url = https://web.archive.org/web/20081001223217/http://genome.wellcome.ac.uk/assets/wtx030893.pdf | url-status=dead | archive-date = 1 October 2008 }}</ref>。二重らせん構造が示唆する複製機構の最終確認は、1958年の[[メセルソン-スタールの実験]]によってなされた<ref>{{cite journal | vauthors = Meselson M, Stahl FW | title = The Replication of DNA in Escherichia Coli | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 44 | issue = 7 | pages = 671–82 | date = July 1958 | pmid = 16590258 | pmc = 528642 | doi = 10.1073/pnas.44.7.671 | bibcode = 1958PNAS...44..671M | doi-access = free }}</ref>。クリックと共同研究者らによる更なる研究によって、遺伝暗号が[[コドン]]と呼ばれる塩基の非重複トリプレット(三連符)<!-- non-overlapping triplets of bases -->に基づいていることが明らかにされ、[[ハー・ゴビンド・コラナ]]、[[ロバート・W・ホリー]]、[[マーシャル・ニーレンバーグ]]によって遺伝暗号の解読が可能となった<ref>{{cite web |url=http://nobelprize.org/nobel_prizes/medicine/laureates/1968/ |title=The Nobel Prize in Physiology or Medicine 1968 |work=Nobelprize.org |access-date=2006-12-24}}</ref>。[[分子生物学]]の誕生は、これらの発見が基礎となった<ref>{{cite journal | vauthors = Pray L | year = 2008 | title = Discovery of DNA structure and function: Watson and Crick. | journal = Nature Education | volume = 1 | issue = 1 | pages = 100 }}</ref>。 |
|||
{{clear}} |
|||
1986年、英国の警察がレスター大学のアレック・ジェフリーズに強姦殺人に関する容疑者の自白の検証または反証を依頼したとき、DNA鑑定は初めて犯罪捜査に利用された。この特別な事件では、容疑者は2件の強姦殺人を自白していたが、後に自白を撤回した。大学の研究所でのDNA鑑定によって、容疑者の当初の「自白」の真実性はすぐに否定され、容疑者は強姦殺人の容疑を晴らすことができた<ref>{{cite journal | pmc=3561883 | year=2003 | vauthors=Panneerchelvam S, Norazmi MN | title=Forensic DNA Profiling and Database | journal=The Malaysian Journal of Medical Sciences | volume=10 | issue=2 | pages=20–26 | pmid=23386793 }}</ref>。 |
|||
== 符号位置 == |
== 符号位置 == |
||
| 137行目: | 382行目: | ||
{{CharCode|129516|1F9EC|-|dna|font=絵文字フォント}} |
{{CharCode|129516|1F9EC|-|dna|font=絵文字フォント}} |
||
|} |
|} |
||
== 取扱 == |
|||
== 参照項目 == |
|||
特定の個人のDNAは一般に[[個人情報]]を構成すると考えられており、[[2022年]]2月には、[[フランス大統領]]の[[エマニュエル・マクロン]]は[[クレムリン]]での[[ウラジーミル・プーチン]][[ロシア大統領]]との会談時、DNA採取を防ぐために、ロシア側による[[COVID-19]]の[[ポリメラーゼ連鎖反応|PCR]]検査を拒んだという<ref>{{cite news|title= Macron refused Russian COVID test in Putin trip over DNA theft fears|url= https://www.reuters.com/world/europe/putin-kept-macron-distance-snubbing-covid-demands-sources-2022-02-10/|author= Michel Rose|publisher=REUTERS|date=2022-02-12}}</ref>。 |
|||
{{Div col}} |
|||
* [[常染色体]] - 性染色体以外の染色体 |
|||
* [[結晶学]] - 結晶性固体中の原子の配列を決定する科学的研究 |
|||
* [[DNAの日|全米DNAの日]] - 4月25日に祝われるアメリカ合衆国の祝日 |
|||
* [[DNAマイクロアレイ]] - 固体表面に付着した微細なDNAスポットの集合体 |
|||
* [[DNAシークエンシング]] - 核酸配列を決定する工程 |
|||
* [[遺伝子疾患]] - ゲノムの1つまたは複数の異常によって引き起こされる健康問題 |
|||
* [[遺伝子系図]] - DNA検査によって推定した個人間の遺伝的関係 |
|||
* [[ハプロタイプ]] - 親から受け継いだ遺伝子の集合 |
|||
* [[減数分裂]] - 単数体の配偶子を作る細胞分裂の様式 |
|||
* {{ill2|核酸表記法|en|Nucleic acid notation}} - DNAの4つのヌクレオチドをA、C、G、Tと呼ぶ国際共通表記法 |
|||
* [[塩基配列]] - 核酸中のヌクレオチドの連続物 |
|||
* [[リボソームDNA]] - リボソームRNAをコーディングするDNAの特定の領域 |
|||
* [[サザンブロット]] - DNA配列の検出と定量に用いられる分析技術 |
|||
* {{ill2|X線回折法|en|X-ray scattering techniques}} - X線ビームの散乱強度に基づく非破壊分析技術の一群 |
|||
* [[ゼノ核酸]] - 合成核酸アナログ |
|||
* {{ill2|三本鎖DNA|en|Triple-stranded DNA}} - 3本のオリゴヌクレオチドが三重らせんを形成するDNA構造 |
|||
{{Div col end}} |
|||
== 脚注 == |
== 脚注 == |
||
{{脚注ヘルプ}} |
{{脚注ヘルプ}} |
||
{{Reflist|33em|refs = |
|||
=== 注釈 === |
|||
{{Reflist|group="†"|}} |
|||
<ref name="Birney_2007">{{cite journal |author-link2=John Stamatoyannopoulos | vauthors = Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, Weng Z, Snyder M, Dermitzakis ET, Thurman RE, Kuehn MS, Taylor CM, Neph S, Koch CM, Asthana S, Malhotra A, Adzhubei I, Greenbaum JA, Andrews RM, Flicek P, Boyle PJ, Cao H, Carter NP, Clelland GK, Davis S, Day N, Dhami P, Dillon SC, Dorschner MO, Fiegler H, Giresi PG, Goldy J, Hawrylycz M, Haydock A, Humbert R, James KD, Johnson BE, Johnson EM, Frum TT, Rosenzweig ER, Karnani N, Lee K, Lefebvre GC, Navas PA, Neri F, Parker SC, Sabo PJ, Sandstrom R, Shafer A, Vetrie D, Weaver M, Wilcox S, Yu M, Collins FS, Dekker J, Lieb JD, Tullius TD, Crawford GE, Sunyaev S, Noble WS, Dunham I, Denoeud F, Reymond A, Kapranov P, Rozowsky J, Zheng D, Castelo R, Frankish A, Harrow J, Ghosh S, Sandelin A, Hofacker IL, Baertsch R, Keefe D, Dike S, Cheng J, Hirsch HA, Sekinger EA, Lagarde J, Abril JF, Shahab A, Flamm C, Fried C, Hackermüller J, Hertel J, Lindemeyer M, Missal K, Tanzer A, Washietl S, Korbel J, Emanuelsson O, Pedersen JS, Holroyd N, Taylor R, Swarbreck D, Matthews N, Dickson MC, Thomas DJ, Weirauch MT, Gilbert J, Drenkow J, Bell I, Zhao X, Srinivasan KG, Sung WK, Ooi HS, Chiu KP, Foissac S, Alioto T, Brent M, Pachter L, Tress ML, Valencia A, Choo SW, Choo CY, Ucla C, Manzano C, Wyss C, Cheung E, Clark TG, Brown JB, Ganesh M, Patel S, Tammana H, Chrast J, Henrichsen CN, Kai C, Kawai J, Nagalakshmi U, Wu J, Lian Z, Lian J, Newburger P, Zhang X, Bickel P, Mattick JS, Carninci P, Hayashizaki Y, Weissman S, Hubbard T, Myers RM, Rogers J, Stadler PF, Lowe TM, Wei CL, Ruan Y, Struhl K, Gerstein M, Antonarakis SE, Fu Y, Green ED, Karaöz U, Siepel A, Taylor J, Liefer LA, Wetterstrand KA, Good PJ, Feingold EA, Guyer MS, Cooper GM, Asimenos G, Dewey CN, Hou M, Nikolaev S, Montoya-Burgos JI, Löytynoja A, Whelan S, Pardi F, Massingham T, Huang H, Zhang NR, Holmes I, Mullikin JC, Ureta-Vidal A, Paten B, Seringhaus M, Church D, Rosenbloom K, Kent WJ, Stone EA, Batzoglou S, Goldman N, Hardison RC, Haussler D, Miller W, Sidow A, Trinklein ND, Zhang ZD, Barrera L, Stuart R, King DC, Ameur A, Enroth S, Bieda MC, Kim J, Bhinge AA, Jiang N, Liu J, Yao F, Vega VB, Lee CW, Ng P, Shahab A, Yang A, Moqtaderi Z, Zhu Z, Xu X, Squazzo S, Oberley MJ, Inman D, Singer MA, Richmond TA, Munn KJ, Rada-Iglesias A, Wallerman O, Komorowski J, Fowler JC, Couttet P, Bruce AW, Dovey OM, Ellis PD, Langford CF, Nix DA, Euskirchen G, Hartman S, Urban AE, Kraus P, Van Calcar S, Heintzman N, Kim TH, Wang K, Qu C, Hon G, Luna R, Glass CK, Rosenfeld MG, Aldred SF, Cooper SJ, Halees A, Lin JM, Shulha HP, Zhang X, Xu M, Haidar JN, Yu Y, Ruan Y, Iyer VR, Green RD, Wadelius C, Farnham PJ, Ren B, Harte RA, Hinrichs AS, Trumbower H, Clawson H, Hillman-Jackson J, Zweig AS, Smith K, Thakkapallayil A, Barber G, Kuhn RM, Karolchik D, Armengol L, Bird CP, de Bakker PI, Kern AD, Lopez-Bigas N, Martin JD, Stranger BE, Woodroffe A, Davydov E, Dimas A, Eyras E, Hallgrímsdóttir IB, Huppert J, Zody MC, Abecasis GR, Estivill X, Bouffard GG, Guan X, Hansen NF, Idol JR, Maduro VV, Maskeri B, McDowell JC, Park M, Thomas PJ, Young AC, Blakesley RW, Muzny DM, Sodergren E, Wheeler DA, Worley KC, Jiang H, Weinstock GM, Gibbs RA, Graves T, Fulton R, Mardis ER, Wilson RK, Clamp M, Cuff J, Gnerre S, Jaffe DB, Chang JL, Lindblad-Toh K, Lander ES, Koriabine M, Nefedov M, Osoegawa K, Yoshinaga Y, Zhu B, de Jong PJ | display-authors = 6 | title = Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project | journal = Nature | volume = 447 | issue = 7146 | pages = 799–816 | date = June 2007 | pmid = 17571346 | pmc = 2212820 | doi = 10.1038/nature05874 | bibcode = 2007Natur.447..799B }}</ref> |
|||
=== 出典 === |
|||
{{Reflist|2}} |
|||
<ref name="Gregory_2006">{{cite journal | vauthors = Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, Scott CE, Howe KL, Woodfine K, Spencer CC, Jones MC, Gillson C, Searle S, Zhou Y, Kokocinski F, McDonald L, Evans R, Phillips K, Atkinson A, Cooper R, Jones C, Hall RE, Andrews TD, Lloyd C, Ainscough R, Almeida JP, Ambrose KD, Anderson F, Andrew RW, Ashwell RI, Aubin K, Babbage AK, Bagguley CL, Bailey J, Beasley H, Bethel G, Bird CP, Bray-Allen S, Brown JY, Brown AJ, Buckley D, Burton J, Bye J, Carder C, Chapman JC, Clark SY, Clarke G, Clee C, Cobley V, Collier RE, Corby N, Coville GJ, Davies J, Deadman R, Dunn M, Earthrowl M, Ellington AG, Errington H, Frankish A, Frankland J, French L, Garner P, Garnett J, Gay L, Ghori MR, Gibson R, Gilby LM, Gillett W, Glithero RJ, Grafham DV, Griffiths C, Griffiths-Jones S, Grocock R, Hammond S, Harrison ES, Hart E, Haugen E, Heath PD, Holmes S, Holt K, Howden PJ, Hunt AR, Hunt SE, Hunter G, Isherwood J, James R, Johnson C, Johnson D, Joy A, Kay M, Kershaw JK, Kibukawa M, Kimberley AM, King A, Knights AJ, Lad H, Laird G, Lawlor S, Leongamornlert DA, Lloyd DM, Loveland J, Lovell J, Lush MJ, Lyne R, Martin S, Mashreghi-Mohammadi M, Matthews L, Matthews NS, McLaren S, Milne S, Mistry S, Moore MJ, Nickerson T, O'Dell CN, Oliver K, Palmeiri A, Palmer SA, Parker A, Patel D, Pearce AV, Peck AI, Pelan S, Phelps K, Phillimore BJ, Plumb R, Rajan J, Raymond C, Rouse G, Saenphimmachak C, Sehra HK, Sheridan E, Shownkeen R, Sims S, Skuce CD, Smith M, Steward C, Subramanian S, Sycamore N, Tracey A, Tromans A, Van Helmond Z, Wall M, Wallis JM, White S, Whitehead SL, Wilkinson JE, Willey DL, Williams H, Wilming L, Wray PW, Wu Z, Coulson A, Vaudin M, Sulston JE, Durbin R, Hubbard T, Wooster R, Dunham I, Carter NP, McVean G, Ross MT, Harrow J, Olson MV, Beck S, Rogers J, Bentley DR, Banerjee R, Bryant SP, Burford DC, Burrill WD, Clegg SM, Dhami P, Dovey O, Faulkner LM, Gribble SM, Langford CF, Pandian RD, Porter KM, Prigmore E | display-authors = 6 | title = The DNA sequence and biological annotation of human chromosome 1 | journal = Nature | volume = 441 | issue = 7091 | pages = 315–21 | date = May 2006 | pmid = 16710414 | doi = 10.1038/nature04727 | bibcode = 2006Natur.441..315G | doi-access = free }}</ref> |
|||
=== 参考文献 === |
|||
<ref name="Venter_2001">{{cite journal | vauthors = Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Huson DH, Wortman JR, Zhang Q, Kodira CD, Zheng XH, Chen L, Skupski M, Subramanian G, Thomas PD, Zhang J, Gabor Miklos GL, Nelson C, Broder S, Clark AG, Nadeau J, McKusick VA, Zinder N, Levine AJ, Roberts RJ, Simon M, Slayman C, Hunkapiller M, Bolanos R, Delcher A, Dew I, Fasulo D, Flanigan M, Florea L, Halpern A, Hannenhalli S, Kravitz S, Levy S, Mobarry C, Reinert K, Remington K, Abu-Threideh J, Beasley E, Biddick K, Bonazzi V, Brandon R, Cargill M, Chandramouliswaran I, Charlab R, Chaturvedi K, Deng Z, Di Francesco V, Dunn P, Eilbeck K, Evangelista C, Gabrielian AE, Gan W, Ge W, Gong F, Gu Z, Guan P, Heiman TJ, Higgins ME, Ji RR, Ke Z, Ketchum KA, Lai Z, Lei Y, Li Z, Li J, Liang Y, Lin X, Lu F, Merkulov GV, Milshina N, Moore HM, Naik AK, Narayan VA, Neelam B, Nusskern D, Rusch DB, Salzberg S, Shao W, Shue B, Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F, Zhong W, Zhu S, Zhao S, Gilbert D, Baumhueter S, Spier G, Carter C, Cravchik A, Woodage T, Ali F, An H, Awe A, Baldwin D, Baden H, Barnstead M, Barrow I, Beeson K, Busam D, Carver A, Center A, Cheng ML, Curry L, Danaher S, Davenport L, Desilets R, Dietz S, Dodson K, Doup L, Ferriera S, Garg N, Gluecksmann A, Hart B, Haynes J, Haynes C, Heiner C, Hladun S, Hostin D, Houck J, Howland T, Ibegwam C, Johnson J, Kalush F, Kline L, Koduru S, Love A, Mann F, May D, McCawley S, McIntosh T, McMullen I, Moy M, Moy L, Murphy B, Nelson K, Pfannkoch C, Pratts E, Puri V, Qureshi H, Reardon M, Rodriguez R, Rogers YH, Romblad D, Ruhfel B, Scott R, Sitter C, Smallwood M, Stewart E, Strong R, Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril JF, Guigó R, Campbell MJ, Sjolander KV, Karlak B, Kejariwal A, Mi H, Lazareva B, Hatton T, Narechania A, Diemer K, Muruganujan A, Guo N, Sato S, Bafna V, Istrail S, Lippert R, Schwartz R, Walenz B, Yooseph S, Allen D, Basu A, Baxendale J, Blick L, Caminha M, Carnes-Stine J, Caulk P, Chiang YH, Coyne M, Dahlke C, Mays A, Dombroski M, Donnelly M, Ely D, Esparham S, Fosler C, Gire H, Glanowski S, Glasser K, Glodek A, Gorokhov M, Graham K, Gropman B, Harris M, Heil J, Henderson S, Hoover J, Jennings D, Jordan C, Jordan J, Kasha J, Kagan L, Kraft C, Levitsky A, Lewis M, Liu X, Lopez J, Ma D, Majoros W, McDaniel J, Murphy S, Newman M, Nguyen T, Nguyen N, Nodell M, Pan S, Peck J, Peterson M, Rowe W, Sanders R, Scott J, Simpson M, Smith T, Sprague A, Stockwell T, Turner R, Venter E, Wang M, Wen M, Wu D, Wu M, Xia A, Zandieh A, Zhu X | display-authors = 6 | title = The sequence of the human genome | journal = Science | volume = 291 | issue = 5507 | pages = 1304–51 | date = February 2001 | pmid = 11181995 | doi = 10.1126/science.1058040 | bibcode = 2001Sci...291.1304V | doi-access = free }}</ref> |
|||
}} |
|||
== 推薦文献 == |
|||
{{refbegin}} |
|||
* {{Cite book|和書|author = 田村隆明|editor = |title = 基礎細胞生物学|edition = 第1版第1刷|year = 2010|publisher = [[東京化学同人]]|isbn = 978-4-8079-0724-3|page = |ref = 田村(2010)}} |
* {{Cite book|和書|author = 田村隆明|editor = |title = 基礎細胞生物学|edition = 第1版第1刷|year = 2010|publisher = [[東京化学同人]]|isbn = 978-4-8079-0724-3|page = |ref = 田村(2010)}} |
||
* {{Cite book|和書|author = 武村政春|editor = |title = DNAを操る分子たち|edition = 初版第1刷|year = 2012|publisher = [[技術評論社]]|isbn = 978-4-7741-4998-1|page = |ref = 武村(2012)}} |
* {{Cite book|和書|author = 武村政春|editor = |title = DNAを操る分子たち|edition = 初版第1刷|year = 2012|publisher = [[技術評論社]]|isbn = 978-4-7741-4998-1|page = |ref = 武村(2012)}} |
||
* {{cite book | vauthors = Berry A, Watson J | author-link2 = James Watson | name-list-style = vanc | title = DNA: the secret of life | publisher = Alfred A. Knopf | location = New York | year = 2003 | isbn = 0-375-41546-7 | url = https://archive.org/details/dnasecretoflife00wats }} |
|||
* {{cite book |title=Understanding DNA: the molecule & how it works | vauthors = Calladine CR, Drew HR, Luisi BF, Travers AA | year = 2003 |publisher=Elsevier Academic Press |location=Amsterdam |isbn=0-12-155089-3}} |
|||
== 関連項目 == |
|||
* {{cite book | vauthors = Carina D, Clayton J | title = 50 years of DNA | publisher = Palgrave Macmillan | location = Basingstoke | year = 2003 | isbn = 1-4039-1479-6 | url = https://archive.org/details/50yearsofdna00clay }} |
|||
{{Div col}} |
|||
* {{cite book | author-link = Horace Freeland Judson | vauthors = Judson HF | date = 1979 | title = The Eighth Day of Creation: Makers of the Revolution in Biology | isbn = 0-671-22540-5 | edition = 2nd | publisher = Cold Spring Harbor Laboratory Press }} |
|||
* [[RNA]] |
|||
* {{cite book | vauthors = Olby RC | author-link = Robert Olby | title = The path to the double helix: the discovery of DNA |publisher=Dover Publications |location=New York |year=1994 |isbn=0-486-68117-3}} First published in October 1974 by MacMillan, with foreword by Francis Crick; the definitive DNA textbook, revised in 1994 with a nine-page postscript. |
|||
* [[DNA複製]] |
|||
* {{cite journal | vauthors = Olby R | title = Quiet debut for the double helix | journal = Nature | volume = 421 | issue = 6921 | pages = 402–05 | date = January 2003 | pmid = 12540907 | doi = 10.1038/nature01397 | author-link = Robert Olby | bibcode = 2003Natur.421..402O | doi-access = free }} |
|||
* [[遺伝子]] |
|||
* {{cite book | vauthors = Olby RC | title=Francis Crick: A Biography |publisher=Cold Spring Harbor Laboratory Press |location=Plainview, N.Y |year=2009 |isbn=978-0-87969-798-3}} |
|||
* [[遺伝子工学]] |
|||
* {{cite book | vauthors = Micklas D | date = 2003 | title = DNA Science: A First Course | publisher = Cold Spring Harbor Press | isbn = 978-0-87969-636-8 }} |
|||
* [[プラスミド]] |
|||
* {{cite book | vauthors = Ridley M | author-link = Matt Ridley |title=Francis Crick: discoverer of the genetic code |publisher=Eminent Lives, Atlas Books |location=Ashland, OH |year=2006 |isbn=0-06-082333-X}} |
|||
* [[ペプチド核酸|PNA]] |
|||
* {{cite book | vauthors = Rosenfeld I | date = 2010 | title = DNA: A Graphic Guide to the Molecule that Shook the World | publisher = Columbia University Press | isbn = 978-0-231-14271-7 }} |
|||
* [[ゲノム]] |
|||
* {{cite book | vauthors = Schultz M, Cannon Z | date = 2009 | title = The Stuff of Life: A Graphic Guide to Genetics and DNA | url = https://archive.org/details/stuffoflifegraph00schu | url-access = registration | publisher = Hill and Wang | isbn = 978-0-8090-8947-5 }} |
|||
* [[アガロースゲル電気泳動]] |
|||
* {{cite book | author-link1 = Gunther Stent | vauthors = Stent GS, Watson J |title=The Double Helix: A Personal Account of the Discovery of the Structure of DNA | url = https://archive.org/details/doublehelixpers00wats_0 | url-access = registration |publisher=Norton |location=New York |year=1980 |isbn=0-393-95075-1}} |
|||
* [[キャリー・マリス]] |
|||
* {{cite book | vauthors = Watson J | date = 2004 | title = DNA: The Secret of Life | publisher = Random House | isbn = 978-0-09-945184-6 }} |
|||
* [[ポリメラーゼ連鎖反応|PCR]] |
|||
* {{cite book | author-link = Maurice Wilkins | vauthors = Wilkins M |title=The third man of the double helix the autobiography of Maurice Wilkins |publisher=University Press |location=Cambridge, England |year=2003 |isbn=0-19-860665-6}} |
|||
* [[相補的塩基対]] |
|||
{{refend}} |
|||
* [[DNA鑑定]] |
|||
* [[DNAマイクロアレイ]] |
|||
* [[DNAシーケンサー]] |
|||
* [[DNAコンピュータ]] |
|||
* [[DNA - DNA分子交雑法]] |
|||
* [[DNAバーコーディング]] |
|||
* [[DNA (小惑星)]] - デオキシリボ核酸に因む[[小惑星]]。 |
|||
* [[DNAの日]] |
|||
* [[ミーム]]:文化における伝統を遺伝子に喩える通俗的な言い方として「何々の'''DNA'''」と用いられることがあるが、これに近い概念として'''ミーム'''がある。学術的な場面以外<!--バラエティ番組とか……-->で「何々の'''DNA'''」という言いまわしが用いられた場合、実質的には「何々の'''ミーム'''」という意味で用いられていることが多い。 |
|||
{{Div col end}} |
|||
== 外部リンク == |
== 外部リンク == |
||
{{Library resources box |
|||
* [http://www.nature.com/nature/dna50/watsoncrick.pdf A Structure for Deoxyribose Nucleic Acid,NATURE No.4356 April 25,1953] |
|||
|onlinebooks=yes |
|||
|by=no |
|||
|lcheading= DNA |
|||
|label=DNA |
|||
}} |
|||
* {{Curlie|Science/Biology/Biochemistry_and_Molecular_Biology/Biomolecules/Nucleic_Acids/DNA|DNA}} |
|||
* [https://web.archive.org/web/20070306082905/http://pipe.scs.fsu.edu/displar.html DNA binding site prediction on protein] |
|||
* [https://web.archive.org/web/20100223035803/http://nobelprize.org/educational_games/medicine/dna_double_helix/ DNA the Double Helix Game] From the official Nobel Prize web site |
|||
* [http://www.fidelitysystems.com/Unlinked_DNA.html DNA under electron microscope] |
|||
* [http://www.dnalc.org/ Dolan DNA Learning Center] |
|||
* [http://www.nature.com/nature/dna50/archive.html Double Helix: 50 years of DNA], ''[[:en:Nature (journal)|Nature]]'' |
|||
* {{Proteopedia|DNA}} |
|||
* {{Proteopedia|Forms_of_DNA}} |
|||
* [http://www.nature.com/encode/ ENCODE threads explorer] ENCODE home page at [[:en:Nature (journal)|Nature]] |
|||
* [https://web.archive.org/web/20070213030135/http://www.ncbe.reading.ac.uk/DNA50/ Double Helix 1953–2003] National Centre for Biotechnology Education |
|||
* [http://www.genome.gov/10506718 Genetic Education Modules for Teachers] – ''DNA from the Beginning'' Study Guide |
|||
* {{PDB Molecule of the Month|23|DNA}} |
|||
* [https://www.nytimes.com/packages/pdf/science/dna-article.pdf "Clue to chemistry of heredity found"]. ''[[:en:The New York Times|The New York Times]]'', June 1953. First American newspaper coverage of the discovery of the DNA structure |
|||
* [http://www.dnaftb.org/ DNA from the Beginning] Another DNA Learning Center site on DNA, genes, and heredity from Mendel to the human genome project. |
|||
* [https://web.archive.org/web/20070825101712/http://orpheus.ucsd.edu/speccoll/testing/html/mss0660a.html#abstract The Register of Francis Crick Personal Papers 1938 – 2007] at Mandeville Special Collections Library, [[:en:University of California, San Diego|University of California, San Diego]] |
|||
* [http://www.nature.com/polopoly_fs/7.9746!/file/Crick%20letter%20to%20Michael.pdf Seven-page, handwritten letter that Crick sent to his 12-year-old son Michael in 1953 describing the structure of DNA.] See [http://www.nature.com/news/crick-s-medal-goes-under-the-hammer-1.12705 Crick's medal goes under the hammer], Nature, 5 April 2013. |
|||
* {{Kotobank}} |
* {{Kotobank}} |
||
| 183行目: | 463行目: | ||
{{Gene expression}} |
{{Gene expression}} |
||
{{Nucleic acids}} |
{{Nucleic acids}} |
||
{{ |
{{Portalbar|生物学}} |
||
{{Normdaten}} |
{{Normdaten}} |
||
{{デフォルトソート:ておきしりほかくさん}} |
|||
[[Category:Helices]] |
|||
[[Category:デオキシリボ核酸|*]] |
[[Category:デオキシリボ核酸|*]] |
||
[[Category:バイオテクノロジー]] |
|||
[[Category:核酸]] |
[[Category:核酸]] |
||
{{デフォルトソート:ておきしりほかくさん}} |
|||
2024年1月28日 (日) 01:33時点における版

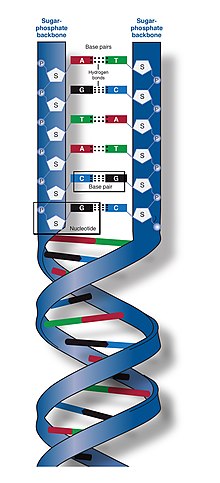
| 遺伝学 |
|---|
 |
デオキシリボ核酸(デオキシリボかくさん、英: deoxyribonucleic acid、DNA[1])は、2本のポリヌクレオチド鎖が互いに巻きついて二重らせんを形成しているポリマーである。このポリマーは、すべての既知の生物と多くのウイルスの発生、機能、成長、および生殖のための遺伝的命令を伝達する。DNAはリボ核酸(英: ribonucleic acid、RNA)とともに核酸と総称される。核酸はタンパク質、脂質、複合多糖と並んで、すべての既知の生命体にとって不可欠な4大生体高分子のひとつである。
DNAの二本鎖は、ヌクレオチドと呼ばれるより単純な単量体単位から構成されていることから、ポリヌクレオチドと呼ばれる[2][3]。各ヌクレオチドは、4つの窒素含有核酸塩基(シトシン: C、グアニン: G、アデニン: A、チミン: T)のうちの1つ、デオキシリボースと呼ばれる糖、およびリン酸基で構成されている。あるヌクレオチドの糖と、次のヌクレオチドのリン酸が共有結合(ホスホジエステル結合と呼ばれる)によって鎖状に結合し、糖-リン酸が交互に繰り返される主鎖が形成される。二本のポリヌクレオチド鎖の窒素塩基は、塩基対合則(AとT、CとG)に従って水素結合で結合し、二本鎖DNAを形成する。窒素塩基は、単環のピリミジンと二重環のプリンという2つのグループに分類される。DNAでは、チミンとシトシンがピリミジン、アデニンとグアニンがプリンである。
二本鎖DNAの両鎖は同一の生物学的情報を保存している。この情報は2本の鎖が分離するときに複製される。DNAの大部分(ヒトでは98%以上)はノンコーディングであり、これらの部分はタンパク質配列のパターンとしては機能しない。DNAの2本の鎖は互いに反対方向に走っているため、逆平行になっている。それぞれの糖には4種類の核酸塩基(または塩基)のうちの1つが結合している。遺伝情報をコード(符号化)するのは、主鎖に沿ったこれら4種類の核酸塩基の配列である。RNA(リボ核酸)鎖はDNA鎖を鋳型として転写と呼ばれる過程で作られ、その際にDNA塩基は対応する塩基と交換されるが、チミン(T)の場合は例外で、RNAはウラシル(U)と交換する[4]。これらのRNA鎖は翻訳と呼ばれる過程で、遺伝暗号に基づいてタンパク質のアミノ酸配列を決定する。
真核細胞では、DNAは染色体と呼ばれる長い構造体に組織化されている。これらの染色体は、通常の細胞分裂の前にDNA複製過程で複製され、それぞれの娘細胞に完全な染色体の集合を提供する。真核生物(動物、植物、真菌類、原生生物)はDNAの大部分を核DNAとして細胞核内に保存し、一部をミトコンドリアDNAとしてミトコンドリア内、あるいは葉緑体DNAとして葉緑体内に保存している[5]。対照的に、原核生物(細菌と古細菌)はDNAを細胞質内の環状染色体にのみ保存している。真核生物の染色体内では、ヒストンなどのクロマチンタンパク質がDNAを小さくまとめて組織化している。これらの緻密な構造は、DNAと他のタンパク質との相互作用を導き、DNAのどの部分が転写されるかを制御するのに役立っている。
特性
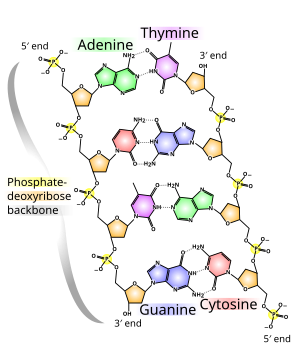
DNAはヌクレオチドと呼ばれる反復単位からなる長いポリマーである[6][7]。DNAの構造はその長さに沿って動的であり、密なループを作ったり、他の形状に巻きつくことができる[8]。どの生物種においても、DNAは水素結合で結合した2本のらせん状の鎖で構成されている。両方の鎖とも、同じ軸にらせん状に巻かれ、ピッチも同じで34オングストローム (3.4 nm)である。一対の鎖の半径は10 Å (1.0 nm)である[9]。別の研究によると、別の溶液中で測定した場合、DNA鎖の幅は22–26 Å (2.2–2.6 nm)、1ヌクレオチド単位の長さは3.3 Å (0.33 nm)であった[10]。ほとんどのDNAの浮力密度は1.7 g/cm3である[11]。
通常、DNAは一本の鎖として存在するのではなく、一対の鎖がしっかりと結合して存在する[9][12]。この2本の長い鎖は互いに巻きついて二重らせんを形成している。ヌクレオチドには、DNA分子の主鎖の一部(鎖を構成する)と核酸塩基(らせん内部でもう一方のDNA鎖と相互作用する)の両方が含まれている。糖と結合した核酸塩基はヌクレオシド(英: nucleoside)と呼ばれ、これに対し糖と1つ以上のリン酸基と結合した塩基はヌクレオチド(英: nucleotide)と呼ばれる。(DNAのように)複数のヌクレオチドが結合した生体高分子をポリヌクレオチドと呼ぶ[13]。
DNA鎖の主鎖はリン酸基と糖基が交互に結合してできている[14]。DNAの糖は2-デオキシリボースで、ペントース(炭素数5、五炭糖)の一種である。糖と糖は、隣接する糖環の3番目と5番目の炭素原子間にホスホジエステル結合を形成するリン酸基によって結合している。これらの炭素はそれぞれ、3'末端(three prime end)、5'末端(five prime end)と呼ばれる。プライム記号(')は、デオキシリボースがグリコシド結合を形成する塩基の炭素原子と区別するために使われる[12]。
このようにDNA鎖には通常、リボースの5'炭素に結合したリン酸基(5'ホスホリル)を持つ末端と、リボースの3'炭素に結合した遊離ヒドロキシ基(3'ヒドロキシ)を持つ末端がある。糖-リン酸骨格に沿った3’と5'炭素の配向は、各DNA鎖に方向性(極性とも呼ばれる)を与える。核酸の二重らせんでは、一方の鎖のヌクレオチドの方向ともう一方の鎖のヌクレオチドの方向は反対で、逆平行になっている。DNA鎖の非対称末端については、5'末端方向と3'末端方向という方向性を有し、5'末端はリン酸基を有し、3'末端はヒドロキシ基を有すると呼ばれる。DNAとRNAの大きな違いの一つは糖で、DNAの2-デオキシリボースがRNAではペントース糖のリボースに置き換えられている[12]。
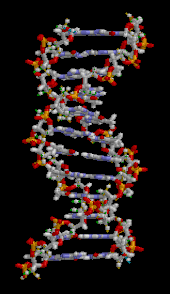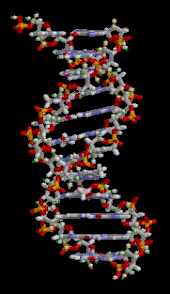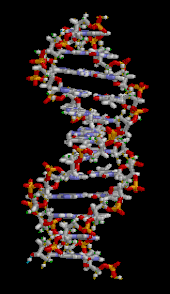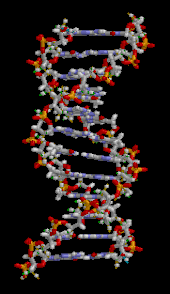
DNA二重らせんは、ヌクレオチド間の水素結合と、芳香族性核酸塩基間の塩基スタッキング相互作用という、主に2つの力によって安定化されている[16]。DNAに含まれる4つの塩基は、アデニン(A)、シトシン(C)、グアニン(G)、チミン(T)である。これらの4つの塩基は、アデノシン一リン酸で示したように、糖-リン酸に結合して完全なヌクレオチドを形成する。アデニンはチミンと対になり、グアニンはシトシンと対になり、それぞれ A-T と G-C の塩基対を形成する[17][18]。
核酸塩基の分類
核酸塩基は、5員および6員の縮合複素環式化合物であるプリン A と G と、6員環のピリミジン C と T の2種類に分類される[12]。第5のピリミジン核酸塩基であるウラシル(U)は通常、RNA内でチミンの代わりを担い、その環上にメチル基を持たない点でチミンと異なる。RNAとDNAに加えて、多くの人工核酸類似体が核酸の特性を研究するため、あるいはバイオテクノロジーで使用するために作成されてきた[19]。
非標準塩基
DNAには修飾塩基が存在する。このうち最初に認識されたのは5-メチルシトシンで、1925年に、結核菌(Mycobacterium tuberculosis)のゲノムから発見された[20]。細菌ウイルス(バクテリオファージ)にこうした非標準塩基(英: noncanonical base)が存在する理由は、細菌に存在する制限酵素を避けるためである。この酵素系は、少なくとも部分的には、細菌をウイルス感染から保護する分子免疫系として働く[21]。より一般的な修飾DNA塩基であるシトシンとアデニンの修飾は、動植物における遺伝子発現のエピジェネティック制御(後成的調整)において、重要な役割を果たしている[22]。
DNAには多くの非標準塩基が存在することが知られている[23]。これらのほとんどは、ウラシルを含む標準塩基(英: canonical base)が修飾されたものである。
- 修飾アデニン
- N6-カルバモイル-メチルアデニン
- N6-メチルアデニン
- 修飾グアニン
- 7-デアザグアニン
- 7-メチルグアニン
- 修飾シトシン
- N4-メチルシトシン
- 5-カルボキシルシトシン
- 5-ホルミルシトシン
- 5-グリコシルヒドロキシメチルシトシン
- 5-ヒドロキシシトシン
- 5-メチルシトシン
- 修飾チミジン
- α-グルタミルチミジン
- α-プトレシニルチミン
- ウラシルおよび修飾物
- 塩基J
- ウラシル
- 5-ジヒドロキシペンタウラシル
- 5-ヒドロキシメチルデオキシウラシル
- その他
- デオキシアルケオシン
- 2,6-ジアミノプリン(2-アミノアデニン)
主溝と副溝

二本のらせん鎖がDNAの主鎖を形成している。もう一つの二重らせんが、その鎖と鎖の間にある空隙、あるいは溝をたどって見いだされる。これらの空隙は塩基対に隣接しており、結合部位となる可能性がある。鎖は互いに対称に配置されていないため、溝の大きさは不均等である。主溝(しゅこう)の幅は22オングストローム (2.2 nm)で、副溝(ふくこう)の幅は12 Å (1.2 nm)である[24]。主溝の方が幅が広いため、塩基の端は副溝よりも主溝の方が近づきやすい。その結果、二本鎖DNAの特異的配列に結合できる転写因子などのタンパク質は、通常、主溝に露出した塩基の側面に接触する傾向がある[25]。このような状況は細胞内のDNAの異常なコンホメーション(立体配座)によって異なるが、主溝と副溝はDNAを通常のB型に巻き戻した場合に見られる幅の違いを反映するよう常に命名されている。
塩基対合
DNAの二重らせんでは、一方の鎖上にあるそれぞれの核酸塩基が、もう一方の鎖上のただ一種類の核酸塩基と結合する。これは相補的塩基対形成(英: complementary base pairing)と呼ばれる。プリンとピリミジンは対合して水素結合を形成し、アデニンとチミンは2本、シトシンとグアニンは3本の水素結合を形成する。このように、二重らせんを挟んで(6炭素環から6炭素環へ)2つのヌクレオチドが結合対を形成する配置は、ワトソン・クリック塩基対と呼ばれる。GC含量の高いDNAはGC含量の低いDNAよりも安定である。フーグスティーン塩基対(英: Hoogsteen base pair、6炭素環と5炭素環の水素結合)は、塩基対形成のまれな変種である[26]。共有結合と異なり、水素結合は比較的簡単に切断したり再結合したりすることができる。そのため二重らせんを構成するDNAの二本鎖は、機械的な力や高温によってファスナーのように引き離すことができる[27]。この塩基対の相補性の結果、DNAらせんの二本鎖配列のすべての情報がそれぞれの鎖に複製され、これはDNA複製に不可欠である。相補的な塩基対間のこの可逆的で特異的な相互作用は、生物におけるDNAのすべての機能にとって重要である[7]。
ssDNAとdsDNA
上述したように、ほとんどのDNA分子は実際には2本のポリマー鎖であり、非共有結合によってらせん状に結合している。この二本鎖DNA構造(英: double-stranded DNA、dsDNA)は、主に鎖内塩基スタッキング相互作用(G,Cスタックが最も強い)によって維持されている。この2本の鎖は、融解(melting)と呼ばれる過程を経て分離し、2本の一本鎖DNA分子(英: single-stranded DNA、ssDNA)を形成することがある。融解は、高温、低塩、高pHの条件下で起こる(低pHもDNAを融解させるが、DNAは酸-脱プリン化により不安定であるため、低pHはほとんど行われない)。
dsDNA型の安定性は、GC含有(G,C塩基対の割合)だけでなく、配列(スタッキングは配列特異的であるため)および長さ(分子が長いほど安定)にも依存する。安定性はさまざまな方法で測定できる。一般的な方法は融解温度(Tm値とも呼ばれる)であり、二本鎖分子の50%が一本鎖分子に変換される温度である。融解温度はDNAのイオン強度と濃度に依存する。したがって、GC塩基対の割合とDNA二重らせんの全長の両方が、DNAの二本鎖間の結合の強さを決定する。GC含量が高く長いDNAらせんは相互作用が強い鎖が多く、AT含量が高く短いDNAらせんは相互作用が弱い鎖が多い[28]。生物学では、DNA二重らせんのうち分離しやすい部分、たとえば一部のプロモーターに含まれる TATAAT プリブノー・ボックスなどは、鎖を引き離しやすくするためにAT含量が高くなる傾向がある[29]。
実験室では、水素結合の半分を切断するのに必要な融解温度Tmを求めることにより、この相互作用の強さを測定することができる。DNA二重らせん内の塩基対がすべて融解すると鎖は分離し、溶液中に完全に独立した2つの分子として存在する。これらの一本鎖DNA分子には単一の共通形状は存在しないが、いくつかのコンホメーションは他のものよりも安定している[30]。
含有量

ヒトの場合、細胞1個あたり、女性の二倍体核ゲノムの総長は6.37ギガ塩基対(Gbp)に及び、長さは208.23 cm、質量は6.51 pgである[31]。男性の値はそれぞれ、6.27 Gbp、205.00 cm、6.41 pgである[31]。各DNAポリマーは、1番染色体のように数億ものヌクレオチドを含むことがある。1番染色体は約2億2千万塩基対からなるヒト最大の染色体で、まっすぐに伸ばすと85 mmの長さになる[32]。
真核生物には、核DNAのほかにミトコンドリアDNA(mtDNA)もあり、ミトコンドリアで使われる特定のタンパク質をコードしている。mtDNAは通常、核DNAに比べて比較的小さい。たとえば、ヒトのミトコンドリアDNAは閉じた環状分子を形成し、それぞれの分子は16,569個[33][34]のDNA塩基対を含み[35]、そうした各分子には通常、ミトコンドリア遺伝子の完全な集合が含まれる。ヒトの各ミトコンドリアには、このようなmtDNA分子が平均して約5個含まれている[35]。各ヒト細胞は約100個のミトコンドリアを含むので、ヒト細胞あたりのmtDNA分子の総数は約500個となる[35]。ただし、細胞あたりのミトコンドリアの量も細胞の種類によって異なり、卵細胞には10万個のミトコンドリアが含まれることがあり、ミトコンドリアゲノム(細胞のDNAの最大90%を構成する)の最大150万コピーに相当する[36]。
センスとアンチセンス
あるDNA配列が、タンパク質に翻訳されるメッセンジャーRNAのコピーと同じである場合、「センス配列」(英: sense sequence)と呼ばれる[37]。反対側の鎖の配列は「アンチセンス配列」(英: antisense sequence)と呼ばれる。センス配列とアンチセンス配列は、同じDNA鎖の異なる部分に存在することがある(すなわち、両方の鎖がセンス配列とアンチセンス配列の両方を含む)。原核生物でも真核生物でもアンチセンスRNA配列が作られるが、これらのRNAの機能は完全には解明されていない[38]。一つの提案は、アンチセンスRNAがRNA-RNA塩基対形成を通じて遺伝子発現の調節に関与しているというものである[39]。
原核生物や真核生物のDNA配列、そしてプラスミドやウイルスではより多くのDNA配列が、オーバーラップ遺伝子(英: overlapping gene)を持つことによってセンス鎖とアンチセンス鎖の区別をあいまいにしている[40]。このような場合、DNA配列の中には、一方の鎖に沿って読まれると一方のタンパク質をコードし、もう一方の鎖に沿って逆方向に読まれるともう一方のタンパク質をコードするという、二重の役割を果たすものがある。細菌では、この重畳が遺伝子転写の調節に関与している可能性がある[41]。一方、ウイルスでは、オーバーラップ遺伝子によって、小さなウイルスゲノム内にコードできる情報量を増加させる[42]。
スーパーコイル
DNAは、DNAスーパーコイル(英: DNA supercoiling、DNA超らせん)と呼ばれる過程で、ロープのようにねじれることがある。DNAが「弛緩した」状態では、鎖は通常10.4塩基対ごとに二重らせんの軸の周りを一周するが、DNAがねじれると鎖はよりきつく、あるいはより緩く巻かれる[43]。DNAがらせんの方向にねじれている場合、これは正のスーパーコイルと呼ばれ、塩基同士はより近くに配置される。もし反対方向にねじれているなら、これは負のスーパーコイルと呼ばれ、塩基同士はより離れやすくなる。自然界では、ほとんどのDNAは、トポイソメラーゼと呼ばれる酵素によって導入される、わずかに負のスーパーコイルを持っている[44]。これらの酵素は、転写やDNA複製などの過程でDNA鎖に生じるねじれ応力を緩和するためにも必要である[45]。
代替DNA構造

DNAは、A-DNA(A型DNA)、B-DNA(B型DNA)、Z-DNA(Z型DNA)などの多くの起こりうるコンホメーションで存在するが、機能的な生物で直接観察されているのはB-DNAとZ-DNAに限られる[14]。DNAが取るコンホメーションは、水和レベル、DNA配列、スーパーコイルの量と方向、塩基の化学修飾、金属イオンの種類と濃度、溶液中のポリアミンの有無に依存する[46]。
A-DNA、およびB-DNAのX線回折パターンについて最初に発表された報告では、パターソン関数に基づく解析が使用され、DNAの配向繊維に限られた構造情報しか得られなかった[47][48]。1953年、ウィルキンスらによって、高水和DNA繊維の in vivo(生体内)B-DNA X線回折散乱パターンについて、ベッセル関数の2乗という観点から別の解析法が提案された[49]。同じジャーナルで、ジェームズ・ワトソンとフランシス・クリックが、DNAのX線回折パターンの分子モデリング解析を発表し、その構造が二重らせんであることを提案した[9]。
B-DNAは細胞内で見られる条件下で最もありふれているが[50]、これは明確に定義されたコンホメーションではなく、細胞内で見られる高水和レベルで生じる関連するDNAコンホメーションの一群である[51]。それらに対応するX線回折とX線散乱のパターンは、かなりの程度の無秩序を伴う分子準結晶に特徴的である[52][53]。
B-DNAと比較すると、A-DNAは浅く広い副溝と狭く深い主溝を持つ、より幅の広い右巻きらせんである。A型は、非生理学的条件下では、部分的に脱水したDNA試料中に生じるが、細胞内ではDNA鎖とRNA鎖の混成ペアリングや、酵素-DNA複合体に生じることがある[54][55]。塩基がメチル化で化学修飾されたDNAセグメントは、より大きなコンホメーション変化を起こし、Z-DNAを取ることがある。この場合、鎖はらせん軸を中心に左巻きのらせんを描き、より一般的なB型とは正反対となる[56]。このような特異な構造は、特異的なZ-DNA結合タンパク質によって認識され、転写制御に関与している可能性がある[57]。
代替DNA化学
宇宙生物学者たちは長年にわたり、現在知られている生命とは根本的に異なる生化学的および分子学的プロセスを用いる、地球上の微生物生物圏(影の生物圏)の存在を提案してきた。その提案の一つは、DNA中のリンの代わりにヒ素を使用する生命体の存在であった。2010年、GFAJ-1という細菌におけるその可能性が報告されたが[58][59]、この研究は論争を呼び[59][60]、細菌がDNA骨格や他の生体分子へのヒ素の取り込みを積極的に妨げていることを示唆する証拠が示された[61]。
四重鎖構造
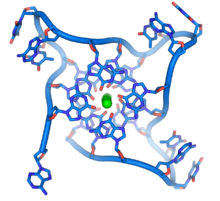
線状染色体の末端には、テロメアと呼ばれる特殊なDNA領域がある。テロメアの主な役割は、通常DNAを複製する酵素は染色体の3'末端の端部をコピーできないため、細胞がテロメラーゼという酵素を使用して染色体末端を複製できるようにすることである[63]。これらの特殊な染色体キャップはDNA末端を保護し、細胞のDNA修復系がそれらを修正すべき損傷として扱うことを防ぐのにも役立つ[64]。ヒト細胞ではテロメアは通常、単純な TTAGGG 配列が数千回繰り返された一本鎖DNAである[65]。
これらのグアニンに富んだ配列は他のDNA分子に見られる通常の塩基対ではなく、4塩基単位が積み重なった構造を形成することによって染色体末端を安定化させる可能性がある。ここでは4つのグアニン塩基が、グアニンテトラッド(英: guanine tetrad)と呼ばれる平面を形成している。そして、これらの4塩基単位の平面が積み重なり、安定したグアニン四重鎖構造を形成する[66]。これらの構造は、塩基の端同士の水素結合と、各4塩基単位の中心にある金属イオンのキレート化によって安定化している[67]。他の構造を形成することも可能で、中央にある4塩基の集まりは、塩基の周囲に折りたたまれた単鎖か、それぞれが中央の構造に1塩基ずつ寄与するいくつかの異なる平行鎖のいずれかから形成される。
このような積層構造に加えて、テロメアはテロメアループ(Tループ)と呼ばれる大きなループ構造も形成する。ここでは、一本鎖DNAがテロメア結合タンパク質によって安定化された大きな円を描くように巻きついている[68]。Tループの最先端では一本鎖テロメアDNAがテロメア鎖によって二本鎖DNAの領域に保持され、二重らせんDNAを分離し、二本鎖の一方と塩基対を形成する。この三重鎖構造は、置換ループあるいはDループと呼ばれる[66]。
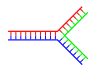
|

|
| 単一分岐 | 多重分岐 |
分岐DNA
DNAでは、相補的であるべき二本鎖DNAの末端部に相補的でない領域が存在すると「ほつれ」を生じる。しかし第三のDNA鎖が導入され、既存の二本鎖のほつれ領域と混成できる隣接領域を含む場合、分岐DNA(英: branched DNA)が生じる可能性がある。分岐DNAの最も単純な例は3本のDNA鎖のみであるが、さらなる鎖と複数の分岐を含む複合体も可能である[69]。分岐DNAは、幾何学的形状を構築するためにナノテクノロジーで使用することができる。以下の技術における用途の節も参照のこと。
人工塩基
いくつかの人工塩基が合成され、ハチモジDNA(英: Hachimoji DNA)と呼ばれる8塩基の核酸アナログに組み込むことに成功した。S、B、P、Zと命名されたこれらの人工塩基は、予測可能な方法で互いに結合し(S-BとP-Z)、DNAの二重らせん構造を維持し、RNAに転写することができる。これらの人工塩基の存在は、地球上で進化してきた4つの天然の核酸塩基には特別なものは何もないことを示すものと考えられる[70][71]。一方、DNAはRNAと密接な関係にあり、RNAはDNAの転写産物としてだけではなく、細胞内で多くの仕事をこなす分子機械でもある。そのためには、RNAは適切な構造に折り畳まれなければならない。すべての可能な立体構造を作るためには、対応するRNAに少なくとも4つの塩基が必要であることが示されている[72]。一方、それ以上の数も可能であるが、これは最小努力の自然原理に反することになる。
酸性度
DNAのリン酸基はリン酸と同様の酸性特性を与えることから、強酸とみなすことができる。DNAは、通常の細胞内pHでは完全にイオン化し、陽子を放出してリン酸基は負電荷を帯びる。これらの負電荷は、DNAを加水分解しうる求核物質をはねつけて、加水分解による分解からDNAを保護する[73]。

巨視的外観
細胞から抽出された純粋なDNAは、白い糸状の凝集塊を形成する[74]。
化学修飾とDNAパッケージングの変化
塩基修飾とDNAパッケージング
遺伝子の発現は、DNAが染色体の中でクロマチンと呼ばれる階層的な構造にどのようにパッケージングされているかに影響される。塩基修飾はパッケージングに関与する可能性があり、遺伝子発現が低いかまったくない領域は通常、シトシン塩基のメチル化が高レベルで見られる。DNAパッケージングとその遺伝子発現への影響は、クロマチン構造においてDNAが巻きついているヒストンタンパク質コアの共有結合修飾や、クロマチン・リモデリング複合体によるリモデリングでも起こりうる。さらに、DNAメチル化とヒストン修飾の間にはクロストークがあるため、クロマチンと遺伝子発現に協調的に影響を与える可能性がある[75]。
たとえば、シトシンのメチル化は5-メチルシトシンを生成し、これはX染色体の不活性化に重要である[76]。メチル化の平均レベルは生物によって異なり、カエノラブディティス・エレガンス(Caenorhabditis elegans)という線虫はシトシンのメチル化を欠くが、脊椎動物はメチル化のレベルが高く、DNAの最大1%が5-メチルシトシンを含む[77]。5-メチルシトシンは重要であるにもかかわらず、脱アミノ化してチミン塩基に変換されることがあるため、メチル化シトシンは特に変異を起こしやすい[78]。その他の塩基修飾としては、細菌におけるアデニンのメチル化、脳における5-ヒドロキシメチルシトシンの存在[79]、およびキネトプラスト類における塩基Jを生成するためのウラシルのグリコシル化などがある[80][81]。
損傷

DNAは、DNA配列を変化させるさまざまな種類の変異原によって損傷を受ける可能性がある。変異原には、酸化剤やアルキル化剤などの化学物質のほか、紫外線やX線などの高エネルギー電磁放射線も含まれる。どのようなDNA損傷が生じるかは変異原の種類によって異なる。たとえば、紫外線はピリミジン塩基間の架橋であるチミン二量体を生成することによって、DNAに損傷を与える可能性がある[83]。一方、フリーラジカルや過酸化水素のような酸化剤は、塩基修飾、特にグアノシンの修飾や、二本鎖切断など、さまざまな形の損傷を引き起こす[84]。典型的なヒト細胞には、酸化的損傷を受けた塩基が約15万個所ある[85]。これらの酸化的損傷のうち最も危険なのは修復が困難な二本鎖切断であり、点変異、DNA配列からの挿入や欠失、あるいは染色体転座を引き起こす可能性がある[86]。これらの変異は癌(がん)を引き起こす可能性がある。DNA修復機構には本質的な限界があるため、人間が長生きすれば、いずれは誰も癌を発症することになる[87][88]。活性酸素種や細胞水の加水分解活性などを産生する正常な細胞プロセスに起因する、自然発生的なDNA損傷も頻繁に起こる。これらの損傷の大部分は修復されるが、どの細胞においても、修復過程の作用にもかかわらず、DNA損傷の一部が残ることがある。これらの残存DNA損傷は、哺乳類の有糸分裂後組織において加齢とともに蓄積する。この蓄積は老化の重要な根本原因であると考えられている[89][90][91]。
変異原の多くは隣接する2つの塩基対の間に侵入し、これはインターカレーション(英: intercalation)と呼ばれる過程である。ほとんどのインターカレーター(侵入物質)は芳香族の平面分子であり、たとえば臭化エチジウム、アクリジン、ダウノルビシン、ドキソルビシンなどである。インターカレーターが塩基対の間に侵入するためには、塩基が離れなければならず、二重らせんがほどけることでDNA鎖に歪みが生じる。これは転写とDNA複製の両方を阻害し、毒性と変異を引き起こす[92]。その結果、DNAインターカレーターは発癌性を生じ、またサリドマイドの場合は催奇形性を生じる可能性がある[93]。また、ベンゾ[a]ピレンジオールエポキシドやアフラトキシンのように、DNA付加体を形成し、複製誤りを引き起こすものもある[94]。それにもかかわらず、DNAの転写や複製を阻害する能力があるため、他の類似毒素も、急速に増殖する癌細胞を阻害する化学療法に使用されている[95]。
生物学的機能
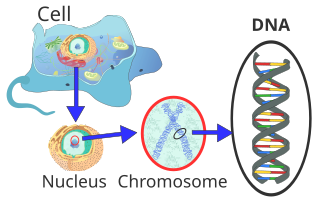
DNAは通常、真核生物では線状染色体として存在し、原核生物では環状染色体として存在する。細胞内の染色体の集合がゲノムを構成し、ヒトゲノムでは46本の染色体に約30億塩基対のDNAが配置されている[96]。DNAが伝達する情報は、遺伝子と呼ばれるDNA断片の配列に含まれている。遺伝子による遺伝情報の伝達すなわち遺伝は、相補的な塩基対形成によって達成される。たとえば、転写において細胞が遺伝子の情報を使用する際、DNAと正しいRNAヌクレオチドとの間に引力が作用することで、DNA配列が相補的なRNA配列に複製される。通常、翻訳と呼ばれる過程で、このRNAコピーは一致するタンパク質配列を作るために使用されるが、これもRNAヌクレオチド間の同様な相互作用に依存している。あるいは、細胞はDNA複製と呼ばれる過程で、その遺伝情報を複製することができる。これらの機能の詳細については他の記事で取り上げており、ここではゲノムの機能を仲介するDNAと他の分子との相互作用に焦点を当てる。
遺伝子とゲノム
ゲノムDNAは、DNA凝縮と呼ばれる過程を通じて、細胞の小さな体積に収まるようにきつく整然と詰め込まれている。真核生物の場合、DNAは細胞核に存在し、ミトコンドリアや葉緑体にも少量が存在する。原核生物では、DNAは核様体(ヌクレオイド)と呼ばれる細胞質内の不規則な形をした構造体に保持されている[97]。ゲノムの遺伝情報は遺伝子内に保持されており、生物におけるこの情報の完全な集合をその遺伝型(英: genotype)と呼ぶ。遺伝子は遺伝の単位であり、生物の特定の形質に影響を与えるDNAの領域である。遺伝子には、転写可能なオープンリーディングフレームと、オープンリーディングフレームの転写を制御するプロモーターやエンハンサーなどの制御配列が含まれている。
多くの生物種では、ゲノム配列全体のごく一部のみタンパク質をコードしている。たとえば、ヒトゲノムのうちタンパク質をコードするエクソンはわずか約1.5%しかなく、ヒトDNAの50%以上は非コード反復配列で構成されている[98]。真核生物のゲノムに非常に多くの非コードDNAが存在する理由と、ゲノムの大きさ(C値)が生物種によって著しく異なる理由は、「C値の謎」として知られる長年の難問である[99]。しかし、タンパク質をコードしないDNA配列の中には、遺伝子発現の調節に関与する機能的な非コードRNA分子をコードしているものもある[100]。

非コードDNA配列の中には染色体の構造的役割を果たすものがある。テロメアとセントロメアには通常、ほとんど遺伝子が存在しないが、染色体の機能と安定性にとって重要である[64][102]。ヒトに多く存在する非コードDNAは偽遺伝子であり、変異によって機能しなくなった遺伝子の複製である[103]。これらの配列は、遺伝子の重複や分岐の過程を通じて、新しい遺伝子を生み出すための遺伝物質の原料として役に立つこともあるが、通常は単なる分子の遺物である[104]。
転写と翻訳
遺伝子は遺伝情報を含むDNA配列で、生物の表現型に影響を与えることがある。遺伝子内では、DNA鎖に沿った塩基配列がメッセンジャーRNA配列を規定し、それが1つか複数のタンパク質配列を規定する。遺伝子のヌクレオチド配列とタンパク質のアミノ酸配列との関係は、遺伝暗号と総称される翻訳規則によって決定される。遺伝暗号は、コドン(codon)と呼ばれる3文字の「単語」からなり(例:ACT、CAG、TTT)、ヌクレオチドが3個連続した配列に基づいている。
転写の際、遺伝子のコドンがRNAポリメラーゼによってメッセンジャーRNAにコピーされる。次に、このRNAコピーはリボソームによって解読され、リボソームはメッセンジャーRNAをアミノ酸を運ぶトランスファーRNAに塩基対合させることによってRNA配列を読み取る。4種類の塩基を表す3文字が組み合わさって、64通りのコドンの可能性が存在する(43 通りの組み合わせ)。これらのコドンは20種類の標準アミノ酸をコードしており、ほとんどのアミノ酸は複数のコドンに対応付けられる。また、コード領域の終わりを示す3つの「終止コドン」(ナンセンスコドンとも呼ばれる)もある。これらは、TAG、TAA、TGAコドンである(mRNAではUAG、UAA、UGA)。

複製
細胞分裂は生物が成長するために不可欠であるが、細胞が分裂する際には、2つの娘細胞が親と同じ遺伝情報を持つように、ゲノム中のDNAを複製しなければならない。DNAの二本鎖構造はDNA複製の単純な機構を提供する。ここでは二本鎖が分離され、次にDNAポリメラーゼと呼ばれる酵素によってそれぞれの鎖の相補的DNA配列が再作成される。この酵素は、相補的塩基対の形成を通じて正しい塩基を見つけ、それを元の鎖に結合させることで相補鎖を作成する。DNAポリメラーゼはDNA鎖を5'から3'の方向にしか伸長できないため、二重らせんの逆平行鎖を複製するために異なる機構が使われる[105]。このようにして、古い鎖の塩基が新しい鎖の塩基を決定し、細胞はそのDNAの完全な複製を得ることができる。
細胞外核酸
裸の細胞外DNA(英: extracellular DNA、eDNA)は、そのほとんどが細胞死の際に放出されたもので、環境中にほぼ遍在している。土壌中の濃度は2 μg/Lと高く、自然の水性環境中では88 μg/Lに達することもある[106]。eDNAの働きとして、遺伝子の水平伝播への関与[107]、栄養素の供給[108]、あるいはイオンや抗生物質を取り込んだり用量を調整するための緩衝剤としての機能など、さまざまな可能性が提案されている[109]。eDNAは、いくつかの細菌種のバイオフィルムにおいて、機能的な細胞外マトリックス成分として機能する。eDNAの働きには、バイオフィルム内の特定の細胞型の付着と分散を制御する認識因子として働く可能性や[110]、バイオフィルム形成に寄与する可能性[111]、あるいはバイオフィルムの物理的強度と生物学的ストレスに対する抵抗性に寄与する可能性がある[112]。
無細胞胎児DNAは母体の血液中に存在し、その塩基配列を決定することで発達中の胎児に関する多くの情報を得ることができる[113]。
環境DNAとして知られるeDNAは、水中、大気中、陸上における生物種の動きと存在を監視し、その地域の生物多様性を評価する生態学の調査ツールとして、自然科学の分野で利用が拡大している[114][115]。
好中球細胞外トラップ
好中球細胞外トラップ(英: neutrophil extracellular trap、NET)は、主にDNAから構成される細胞外繊維のネットワークであり、白血球の一種である好中球が宿主細胞への損傷を最小限に抑えながら細胞外の病原体を殺滅することを可能にする。
タンパク質との相互作用
DNAの機能はすべてタンパク質との相互作用に依存している。これらのタンパク質相互作用は非特異的であることもあれば、タンパク質が単一のDNA配列に特異的に結合することもある。酵素もDNAに結合することができ、その中でも特に重要なものは、転写とDNA複製の際にDNA塩基配列をコピーするポリメラーゼである。
DNA結合タンパク質

DNAと結合する構造タンパク質は、非特異的DNA-タンパク質相互作用の例としてよく理解されている。染色体内でDNAは構造タンパク質と複合体を形成して保持されている。これらのタンパク質はDNAをクロマチン(染色質)と呼ばれる緻密な構造に組織化する。真核生物では、この構造はヒストンという小さな塩基性タンパク質の複合体にDNAが結合したものであるが、原核生物では複数種類のタンパク質が関与している[116][117]。ヒストンはヌクレオソームと呼ばれる円盤状の複合体を形成し、その表面には二本鎖DNAが2周完全に巻きついている。これらの非特異的相互作用は、ヒストンの塩基性残基がDNAの酸性糖-リン酸骨格とイオン結合を形成することによって生じるもので、したがって、塩基配列とはほとんど無関係である[118]。これらの塩基性アミノ酸残基の化学修飾には、メチル化、リン酸化、アセチル化などがある[119]。これらの化学的変化はDNAとヒストン間の相互作用の強度を変化させ、DNAを転写因子に近づきやすくしたり、あるいは近づきにくくし、転写速度を変化させる[120]。クロマチン内の他の非特異的DNA結合タンパク質には、曲がったDNAや歪んだDNAに結合する高移動度郡タンパク質がある[121]。これらのタンパク質は、ヌクレオソームの配列を曲げたり、染色体を構成する大きな構造体を組み立てる際に重要である[122]。
DNA結合タンパク質のもう一つのグループとして、一本鎖DNAと特異的に結合するDNA結合タンパク質がある。ヒトの場合、複製タンパク質Aがこの一群の中で最もよく理解されており、DNA複製、組換え、DNA修復など、二重らせんが分離するプロセスに関与している[123]。これらの結合タンパク質は一本鎖DNAを安定化させ、ステムループを形成したり、ヌクレアーゼによる分解からDNAを保護していると考えられている。

対照的に、他のタンパク質は特定のDNA配列に結合するような進化をしてきた。最も研究が進んでいるのは、転写を制御するタンパク質であるさまざまな転写因子である。各転写因子はプロモーター近くの特定のDNA配列に結合し、遺伝子の転写を活性化または阻害する。転写因子は2つの方法でこれを行う。一つは、転写を担うRNAポリメラーゼに直接、あるいは他の媒介タンパク質を介して結合することである。これによって、ポリメラーゼはプロモーターに位置し、転写を開始することができる[125]。あるいは、転写因子はプロモーターのヒストンを修飾する酵素と結合することができる。これによってDNA鋳型に対するポリメラーゼの近づきやすさを変化させる[126]。
これらのDNA標的は生物のゲノム全体に存在する可能性があるため、一種類の転写因子の活性が変化すると、何千もの遺伝子に影響を及ぼす可能性がある[127]。その結果、これらのタンパク質はしばしば、環境変化への応答や細胞の分化・発達を制御するシグナル伝達プロセスの標的となる。これらの転写因子のDNAとの相互作用の特異性は、タンパク質がDNA塩基の端と何度も接触して、DNA配列を「読み取る」ことを可能にすることで生じる。これらの塩基相互作用のほとんどは塩基が最も接近しやすい主溝で起こる[25]。
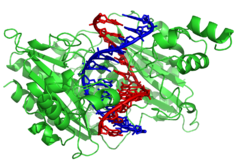
DNA修飾酵素
ヌクレアーゼとリガーゼ
ヌクレアーゼは、ホスホジエステル結合の加水分解を触媒することによってDNA鎖を切断する酵素である。DNA鎖の末端からヌクレオチドを加水分解するヌクレアーゼはエキソヌクレアーゼと呼ばれ、一方エンドヌクレアーゼは鎖内で切断する。分子生物学で最もよく使用されるヌクレアーゼは、特異的配列でDNAを切断する制限エンドヌクレアーゼである。たとえば、上図に示したEcoRV酵素は、DNA鎖の6塩基配列 5′-GATATC-3′ を認識し、水平線で切断する。自然界でこれらの酵素は、制限修飾系の一部として細菌の細胞内に侵入したファージDNAを消化することにより、細菌をファージ感染から保護している[129]。技術分野では、これらの配列特異的ヌクレアーゼは分子クローニングやDNAプロファイリングに使用されている。
DNAリガーゼと呼ばれる酵素は、切断または破損したDNA鎖を再結合させることができる[130]。リガーゼは、ラギング鎖DNA複製において特に重要で、複製フォークで作られた短いDNAセグメントをDNA鋳型の完全なコピーに結合する働きをする。これらはまたDNA修復や遺伝的組換えにも使用される[130]。
トポイソメラーゼとヘリカーゼ
トポイソメラーゼはヌクレアーゼとリガーゼの両方の活性を持つ酵素である。これらのタンパク質はDNAスーパーコイルの量を変化させる。これらの酵素の中には、DNAらせんを切断し、その一部分を回転させることでスーパーコイルのひずみを低減させ、その後DNAの切断部を封着するものもある[44]。別の種類の酵素は、DNAらせんを切断し、その切断部分に2本目のDNAを通過させてから、らせんを再結合することができる[131]。このようにトポイソメラーゼは、DNA複製や転写など、DNAが関与する多くの過程に必要な酵素である[45]。
ヘリカーゼは分子モーターとして働くタンパク質である。これらは、ヌクレオシド三リン酸、主にアデノシン三リン酸(ATP)の化学エネルギーを利用して、塩基間の水素結合を切断し、DNA二重らせんをほどいて一本鎖にする[132]。これらの酵素は、酵素がDNA塩基に近接する必要があるほとんどの過程にとって不可欠である。
ポリメラーゼ
ポリメラーゼはヌクレオシド三リン酸からポリヌクレオチド鎖を合成する酵素である。その生成物の配列は、鋳型(英: template)と呼ばれる既存のポリヌクレオチド鎖に基づいて作られる。これらの酵素は、伸長するポリヌクレオチド鎖末端の3'ヒドロキシ基に繰り返しヌクレオチドを付加する機能を持つ。結果としてすべてのポリメラーゼは5'から3'の方向に働く[133]。これらの酵素の活性部位では、入ってきたヌクレオシド三リン酸が鋳型と塩基対を形成する。これにより、ポリメラーゼは鋳型の相補鎖を正確に合成することができる。ポリメラーゼは、使用する鋳型の種類によって分類される。
DNA複製は、DNA依存性DNAポリメラーゼがDNAポリヌクレオチド鎖のコピーを作る。生物学的情報を保存するためには、各コピーの塩基配列が鋳型鎖の塩基配列と正確に相補的であることが不可欠である。多くのDNAポリメラーゼは校正活性を持っている。これによりポリメラーゼは、ミスマッチしたヌクレオチド間での塩基対形成の欠如によって、合成反応の際にときおり起こる誤りを検出することができる。ミスマッチが検出されると、3'→5'エキソヌクレアーゼ活性が活性化され、誤った塩基が除去される[134]。ほとんどの生物でDNAポリメラーゼは、DNAクランプやヘリカーゼなどの複数のアクセサリー・サブユニットを含む、レプリソームと呼ばれる大きな複合体の中で機能する[135]。
RNA依存性DNAポリメラーゼは、RNA鎖の塩基配列をDNAにコピーする特殊なポリメラーゼである。これらには、レトロウイルスによる細胞感染に関与するウイルス性酵素である逆転写酵素や、テロメアの複製に必要なテロメラーゼが含まれる[63][136]。たとえば、HIV逆転写酵素は、エイズウイルスの複製に関与する酵素である[136]。テロメラーゼは、その構造の一部として自身のRNA鋳型を含むという珍しいポリメラーゼである。これは染色体の末端にテロメアを合成する。テロメアは隣接する染色体末端が融合するのを防ぎ、染色体末端を損傷から保護する[64]。
転写は、DNA鎖の配列をRNAにコピーするDNA依存性RNAポリメラーゼによって行われる。遺伝子の転写を開始するために、RNAポリメラーゼはプロモーターと呼ばれるDNA配列に結合し、DNA鎖を分離する。その後、ターミネーターと呼ばれるDNAの領域に到達するまで、遺伝子配列をメッセンジャーRNA転写物にコピーし、そこで停止してDNAから分離する。ヒトのDNA依存性DNAポリメラーゼと同様に、ヒトゲノムのほとんどの遺伝子を転写する酵素であるRNAポリメラーゼIIは、いくつか調節サブユニットとアクセサリーサブユニットを持つ大きなタンパク質複合体の一部として働いている[137]。
遺伝子組換え

|

|
| 遺伝的組換えにおけるホリデイジャンクション中間体の構造。4本のDNA鎖は、赤、青、緑、黄に色分けされている[138] | |
DNAらせんは通常、他のDNAセグメントと相互作用することはなく、ヒトの細胞では、異なる染色体は染色体テリトリー(英: chromosome territories)と呼ばれる核内の別々の領域を占めることさえある[139]。このように異なる染色体が物理的に分離していることは、DNAが安定した情報保管場所として機能するために重要である。なぜなら、染色体が相互作用する数少ない機会のひとつが、有性生殖の際に起こる染色体交差(英: chromosomal crossover)であり、その際に遺伝的組換えが起こるからである。染色体交差とは、DNAの2本のらせんが切断され、一部が入れ替わり、再び結合することである。
組換えは、染色体が遺伝情報を交換して遺伝子の新しい組み合わせを作り出すことを可能にし、これにより自然選択の効率を高め、新しいタンパク質の急速な進化において重要である[140]。遺伝的組換えはDNA修復、特に二本鎖切断に対する細胞の反応にも関与している可能性がある[141]。
染色体交差の最も一般的な形態は相同組換えで、関与する2つの染色体の配列は非常によく似ている。非相同組換えは、染色体転座や遺伝的異常を生じさせるため、細胞に損傷を与える可能性がある。組換え反応は、RAD51のようなリコンビナーゼとして知られる酵素によって触媒される[142]。組換えの最初の段階は、エンドヌクレアーゼかDNAの損傷によって引き起こされる二本鎖切断である[143]。その後、リコンビナーゼによって部分的に触媒される一連の段階によって、2つのらせんは少なくとも1つのホリデイジャンクションによって結合され、次に、各らせん中の一本鎖セグメントが他方のらせんの相補鎖と二本鎖を形成する。ホリデイジャンクションは四面体の接合構造で、染色体対に沿って移動することができ、一方の鎖をもう一方の鎖と交換することができる。組換え反応は、結合部の切断と遊離したDNAの再結合によって停止する[144]。組換えの際に同じ方向性(極性)の鎖だけがDNAを交換する。切断には東西切断(east-west cleavage)と南北切断(north–south cleavage)の2種類がある。南北切断はDNAの両鎖を切断するが、東西切断はDNAの片鎖をそのまま残す。組換えの際にホリデイジャンクションが形成されることで、遺伝的多様性、染色体上での遺伝子の交換、および野生型ウイルスゲノムの発現が可能になる。
進化
DNAにはあらゆる生命体が機能し、成長し、生殖するための遺伝情報が含まれている。しかし40億年の生命の歴史の中で、DNAがいつからこの機能を果たしてきたかは不明である。最も初期の生命体はRNAを遺伝物質として使っていたのではないかという提案もある[145][146]。RNAは遺伝情報の伝達とリボザイムの一部としての触媒作用の両方を行うことができるため、初期の細胞代謝において中心的な役割を果たしていた可能性がある[147]。核酸が触媒作用と遺伝学の両方に使われていたとする、この古代のRNAワールドは、4塩基に基づく現在の遺伝暗号の進化に影響を与えたかもしれない。このような生物における異なる塩基の数は、少ない塩基数による複製精度の向上と、多数の塩基によるリボザイムの触媒効率の向上との釣り合い関係によってきまった可能性もある[148]。しかしDNAは環境中で100万年未満しか存在できず、溶液中でゆっくりと短い断片に分解されるため、ほとんどの化石からDNAを回収することは不可能で、古代の遺伝子系の直接的な証拠はない[149]。より古いDNAが存在するという主張もなされており、特に2億5千万年前の塩の結晶から生存可能な細菌が分離されたという報告があるが[150]、これらの主張には賛否がある[151][152]。
DNAの構成要素(アデニン、グアニン、および関連する有機分子)は、地球外の宇宙空間で形成された可能性もある[153][154][155]。ウラシル、シトシン、チミンを含む、生命の複雑なDNAやRNAの有機化合物もまた、隕石から発見されたピリミジンのような化学物質を出発点として、宇宙空間の模倣した条件下の実験室で合成されている。ピリミジンは、宇宙で発見された最も炭素を多く含む化学物質である多環芳香族炭化水素(PAH)と同様、赤色巨星や星間宇宙塵やガス雲で形成された可能性がある[156]。
2021年2月、科学者たちは初めて、100万年以上前のマンモス象の遺体からDNA配列を決定したことを報告した。これまでに塩基配列が決定された最古のDNAである[157][158]。
技術における用途
遺伝子工学
フェノール・クロロホルム抽出法のように、生物からDNAを精製する方法や、制限消化やポリメラーゼ連鎖反応のように実験室でDNAを操作する方法が開発された。現代の生物学や生化学では、組換えDNAの分野でこれらの技術を活用している。組換えDNAとは、他のDNA配列から組み立てられた人工のDNA配列である。これらはウイルスベクターを利用して、プラスミドあるいは他の適切な型式で、生物に形質転換することができる[159]。生産された遺伝子組換え生物は、組換えタンパク質のような製品を製造したり、医学研究で使用したり[160]、農業で繁殖したりする[161][162]。
DNAプロファイリング
法科学者は、犯罪現場で発見された血液、精液、皮膚、唾液、または毛髪に含まれるDNAを利用して、加害者などの個人と一致するDNAを特定することができる[163]。この手法は正式にはDNAプロファイリング(英: DNA profiling)と呼ばれ、DNA指紋法(英: DNA fingerprinting)とも呼ばれる。DNAプロファイリングでは、ショートタンデムリピート(縦列型反復配列)やミニサテライトなど、反復DNAの可変部分の長さを個人間で比較する。この方法は通常、一致するDNAを同定するための非常に信頼性の高い技術である[164]。ただし、現場が複数名のDNAで汚染されている場合、同定が複雑になることがある[165]。DNAプロファイリングは1984年にイギリスの遺伝学者アレック・ジェフリーズによって開発され[166]、1988年のエンダービー殺人事件でコリン・ピッチフォークを有罪にするために法科学で初めて使用された[167]。
法科学が発達し、血液、皮膚、唾液、毛髪などの微量サンプルで遺伝子照合ができるようになったことで、多くの事件が再調査されるようになった。当初の調査時には科学的に不可能であった証拠も、現在では発見されることがある。一部の地域において二重の危険の原則(英: double jeopardy law)が撤廃されたこともあいまって、これまでの裁判で陪審を納得させるに十分な証拠が得られなかった事件でも再審が可能になることがある。重大犯罪で起訴された人々は、照合目的でDNAサンプルの提出を求められることがある。法科学的に得られたDNA照合に対する最も明白な抗弁は、証拠の相互汚染が起こったと主張することである。このため、重大犯罪の新事例に対し、細心の注意を払った厳格な取り扱い手順が導入されるようになった。
DNAプロファイリングはまた、集団死傷事件の犠牲者[168]、重大事故の遺体やその一部、集団戦没者墓地における犠牲者個人の身元を、家族との照合によって確認するためにも使用され、成功を収めている。
DNAプロファイリングは、誰かが子供の生みの親または祖父母であるかどうかを判定するためのDNA親子鑑定にも使用され、親とされる人物が子供と生物学的に血縁関係がある場合、親である確率は通常99.99%である。通常のDNA配列決定法は出生後に行われるが、母親がまだ妊娠している間に親子関係を検査する新しい方法がある[169]。
DNA酵素または触媒DNA
デオキシリボザイム(英: deoxyribozyme)は、DNA酵素(DNAzymes)または触媒DNA(catalytic DNA)とも呼ばれ、1994年に初めて発見された[170]。これらの大部分は、in vitro選択法または試験管内進化法(英: Systematic Evolution of Ligands by Exponential Enrichment、SELEX)と呼ばれる組み合わせアプローチを使用して、ランダムなDNA配列の大規模プールから単離された一本鎖DNA配列である。DNA酵素は、RNA-DNA切断、RNA-DNAライゲーション、アミノ酸のリン酸化-脱リン酸化、炭素-炭素結合形成など、さまざまな化学反応を触媒する。DNA酵素は、触媒反応の化学反応速度を、無触媒反応の最大1千億倍に向上させることができる[171]。DNA酵素の中でもっとも広く研究されているのはRNA切断型で、さまざまな金属イオンの検出や治療薬の設計に使用されている。GR-5 DNA酵素(鉛特異的)[170]、CA1-3 DNA酵素(銅特異的)[172]、39E DNA酵素(ウラニル特異的)、NaA43 DNA酵素(ナトリウム特異的)[173]など、いくつかの金属特異的DNA酵素が報告されている。NaA43 DNA酵素は、ナトリウムに対して他の金属イオンよりも10,000倍以上選択的であると報告されており、細胞内でリアルタイムのナトリウムセンサーを作成するために使用された。
バイオインフォマティクス
バイオインフォマティクスは、DNA核酸配列データを含む生物学的データの保存、データマイニング、検索、操作のための技術開発を含む学問分野である。これらの技術は、コンピュータサイエンス、特に文字列検索アルゴリズム、機械学習、データベース理論に広く応用されるようになった[174]。文字列検索またはマッチングアルゴリズムは、より大きな文字列の中にある文字列の出現を検出する手法で、ヌクレオチドの特異的配列を検索するために開発された[175]。DNA配列を他のDNA配列と整列させることで、相同配列を同定し、それらを区別する特異的変異を突き止めることができる。これらの技術、特に多重配列アラインメントは、系統的関係やタンパク質機能を研究する際に使用される[176]。ヒトゲノムプロジェクトで作成されたような全ゲノムDNA配列の大規模なデータセットは、各染色体上の遺伝子や調節エレメントの位置を特定するアノテーションがなくては利用が困難である。タンパク質やRNAをコードする遺伝子に関連する特徴的なパターンを持つDNA配列領域は、遺伝子探索アルゴリズムによって同定することができ、これにより研究者は、特定の遺伝子産物が実験的に単離される前であっても、生物内での存在と可能性のある機能を予測することができる[177]。また、ゲノム全体を比較することで、生物の進化の歴史に焦点を当てたり、複雑な進化の過程を研究することもできる。
DNAナノテクノロジー

DNAナノテクノロジーは、DNAや他の核酸に特有の分子認識特性を利用して、有用な特性を備えた自己集合化能・分岐DNA複合体を作り出す技術領域である[179]。DNAは生物学的情報の伝達手段としてではなく、構造材料として使用することもできる。その結果、2次元周期格子(タイルベースとDNAオリガミ法の両方)や、多面体形状を持つ3次元構造の創造につながった[180]。ナノメカニカルデバイスやアルゴリズム的自己集合化も実証されており[181]、これらのDNA構造は、金ナノ粒子やストレプトアビジンタンパク質など、他の分子集合体の鋳型とするために使用されている[182]。DNAや他の核酸は、アプタマー(さまざまなバイオテクノロジーや生物医学の用途に使われる、特定の標的分子に対する合成オリゴヌクレオチドリガンド)の基礎となっている[183]。
系統学と人類学
DNAは時間の経過とともに変異を蓄積し、遺伝によって歴史的な情報を含んでおり、DNAの塩基配列を比較することで、遺伝学者は生物の進化の歴史、系統発生を推定することができる[184]。系統発生学は進化生物学における強力な道具である。生物種内のDNA配列を比較することで、集団遺伝学者は特定の集団の歴史を知ることができる。これは、生態遺伝学から人類学に至るまで、さまざまな研究に利用できる。
情報ストレージ
情報記録媒体としてのDNAは、電子機器に比べて記録密度がはるかに高いため、非常に大きな可能性を秘めている。しかしコストが高く、読み書きに時間がかかり(メモリレイテンシ)、信頼性が十分でないことなどから、実用化には至っていない[185][186]。
歴史

DNAが最初に単離されたのは、1869年、スイスの医師フリードリッヒ・ミーシェルによって、廃棄された手術用包帯の膿(うみ)の中から微小な物質を発見した時にさかのぼる。細胞核に存在することから、彼はこれを「ヌクレイン(英: nuclein)」と命名した[187][188]。1878年、アルブレヒト・コッセルが「ヌクレイン」の非タンパク質成分である核酸を単離し、その後、5つの標準核酸塩基を単離した[189][190]。
1909年、フィーバス・レヴィーンはRNA(当時は「酵母核酸(英: yeast nucleic acid)」と呼んだ)の塩基、糖、リン酸のヌクレオチド単位を同定した[191][192][193]。1929年、レヴィーンはDNA(当時は「胸腺核酸(英: thymus nucleic acid)」)内のデオキシリボース糖を同定した[194]。レヴィーンは、DNAはリン酸基によって結合された4つのヌクレオチド単位からなる紐で構成されていることを提案した(テトラヌクレオチド仮説)。レヴィーンは、この鎖は短く、塩基が一定の順序で繰り返されていると考えた。1927年、ニコライ・コルツォフは、遺伝形質は「それぞれの鎖を鋳型として半保存的に複製される2本の鏡像鎖」からなる「巨大な遺伝分子」を介して遺伝すると提案した[195][196]。1928年、フレデリック・グリフィスは実験によって、肺炎球菌(Pneumococcus)のS型菌の形質が、死滅したS型菌と生きたR型菌とを混合することによって、R型菌に転換できることを発見した(グリフィスの実験)[197][198]。この実験系は、DNAが遺伝情報を伝達していることを初めて明確に示唆した。
1933年、ウニの未受精卵を研究していたジャン・ブラッシェ(Jean Brachet)は、DNAは細胞核に存在し、RNAは細胞質にのみ存在することを提案した。当時は、酵母核酸(RNA)は植物だけに、胸腺核酸(DNA)は動物だけに存在すると考えられていた。後者は細胞内pHを緩衝する機能を持つ四量体であると考えられていた[199][200]。
1937年、ウィリアム・アストベリーは、DNAが規則正しい構造を持っていることを示すX線回折パターンを初めて作成した[201]。
1943年、オズワルド・アベリーは、共同研究者であるコリン・マクロード、マクリン・マッカーティとともに、DNAが形質転換原理であることを突き止め、グリフィスの提案を支持した(アベリー-マクロード-マッカーティの実験)[202]。エルヴィン・シャルガフは、現在「シャルガフの法則」として知られる見解を発表し、どの生物種のDNAにおいても、グアニンの量はシトシンと等しく、アデニンの量はチミンと等しくなければならないと述べた[203][204]。

1951年末、フランシス・クリックは、英国ケンブリッジ大学のキャヴェンディッシュ研究所でジェームズ・ワトソンとともに研究を始めた。遺伝におけるDNAの役割は、1952年にアルフレッド・ハーシーとマーサ・チェイスが行った一連の実験(ハーシー-チェイス実験)で、DNAが腸内細菌ファージT2の遺伝物質であることを示して確認された[205]。
1952年5月、ロザリンド・フランクリンの指導下で研究をしていた大学院生、レイモンド・ゴスリングは、高水和レベルでのDNA X線回折像を撮影し、「Photo 51」とラベルを付けた[206]。この写真は、モーリス・ウィルキンスからワトソンとクリックに渡されたもので、彼らがDNAの正しい構造を得る上で極めて重要なものであった。フランクリンはクリックとワトソンに、主鎖は外側になければならないと語った。それまでは、ライナス・ポーリングや、ワトソンとクリックらは、鎖が内側にあって塩基が外側を向いた誤ったモデルを持っていた。フランクリンがDNA結晶の空間群を特定したことで、クリックは、DNAの二本鎖が逆平行であることを突き止めた[207]。1953年2月、ライナス・ポーリングとロバート・コリーは、リン酸が軸の近くにあり、塩基が外側にある、3本の鎖が絡み合った核酸のモデルを提案した。ワトソンとクリックはそのモデルを完成させ、現在ではDNA二重らせんの最初の正しいモデルとして受け入れられている[208]。1953年2月28日、クリックは、英国ケンブリッジのザ・イーグルパブで常連客のランチタイムを中断し、彼とワトソンが「生命の秘密を発見した」と発表した[209]。
1953年4月25日、雑誌「Nature」は、ワトソンとクリックの二重らせん構造DNAとそれを支持する証拠を示す一連の5本の論文を掲載した[210]。その構造は、『MOLECULAR STRUCTURE OF NUCLEIC ACIDS A Structure for Deoxyribose Nucleic Acid(核酸の分子構造: デオキシリボース核酸の構造)』と題されたレターで報告され、その中で彼らは次のように述べている。『私たちが仮定した特異的な対形成が、遺伝物質の複製メカニズムである可能性を即座に示唆していることを、私たちは見逃さなかった[9]』。この後、フランクリンとゴスリングのレターが続き、彼ら自身のX線回折データと独自の解析方法が初めて公表された[48][211]。さらに、ウィルキンスと彼の同僚2名によるレターが続き、生体内におけるB-DNA X線パターンの解析が報告されており、生体内にワトソンとクリックの構造が存在することを裏付けていた[49]。
1962年、フランクリンの死後、ワトソン、クリック、ウィルキンスの3名はノーベル生理学・医学賞を共同受賞した[212]。ノーベル賞は存命中の受賞者にのみ授与される。2023年4月、科学者たちは新たな証拠に基づき、ロザリンド・フランクリンはDNA発見の過程に貢献しただけでなく「対等な役割」を果たした人物であり、発見後に発表されたような貢献者ではないと結論づけた[213][214][215]。誰がこの発見の功績を称えられるべきかについては議論が続いている[216]。
1957年に行われた影響力のある講演で、クリックは、分子生物学におけるセントラル・ドグマを打ち出し、DNA、RNA、タンパク質の関係を予言し、「アダプター仮説」を公にした[217]。二重らせん構造が示唆する複製機構の最終確認は、1958年のメセルソン-スタールの実験によってなされた[218]。クリックと共同研究者らによる更なる研究によって、遺伝暗号がコドンと呼ばれる塩基の非重複トリプレット(三連符)に基づいていることが明らかにされ、ハー・ゴビンド・コラナ、ロバート・W・ホリー、マーシャル・ニーレンバーグによって遺伝暗号の解読が可能となった[219]。分子生物学の誕生は、これらの発見が基礎となった[220]。
1986年、英国の警察がレスター大学のアレック・ジェフリーズに強姦殺人に関する容疑者の自白の検証または反証を依頼したとき、DNA鑑定は初めて犯罪捜査に利用された。この特別な事件では、容疑者は2件の強姦殺人を自白していたが、後に自白を撤回した。大学の研究所でのDNA鑑定によって、容疑者の当初の「自白」の真実性はすぐに否定され、容疑者は強姦殺人の容疑を晴らすことができた[221]。
符号位置
| 記号 | Unicode | JIS X 0213 | 文字参照 | 名称 |
|---|---|---|---|---|
| 🧬 | U+1F9EC |
- |
🧬🧬 |
dna |
参照項目
- 常染色体 - 性染色体以外の染色体
- 結晶学 - 結晶性固体中の原子の配列を決定する科学的研究
- 全米DNAの日 - 4月25日に祝われるアメリカ合衆国の祝日
- DNAマイクロアレイ - 固体表面に付着した微細なDNAスポットの集合体
- DNAシークエンシング - 核酸配列を決定する工程
- 遺伝子疾患 - ゲノムの1つまたは複数の異常によって引き起こされる健康問題
- 遺伝子系図 - DNA検査によって推定した個人間の遺伝的関係
- ハプロタイプ - 親から受け継いだ遺伝子の集合
- 減数分裂 - 単数体の配偶子を作る細胞分裂の様式
- 核酸表記法 - DNAの4つのヌクレオチドをA、C、G、Tと呼ぶ国際共通表記法
- 塩基配列 - 核酸中のヌクレオチドの連続物
- リボソームDNA - リボソームRNAをコーディングするDNAの特定の領域
- サザンブロット - DNA配列の検出と定量に用いられる分析技術
- X線回折法 - X線ビームの散乱強度に基づく非破壊分析技術の一群
- ゼノ核酸 - 合成核酸アナログ
- 三本鎖DNA - 3本のオリゴヌクレオチドが三重らせんを形成するDNA構造
脚注
- ^ "deoxyribonucleic acid". Merriam-Webster Dictionary. 2023年12月13日閲覧。
- ^ Molecular Biology of the Cell (6th ed.). Garland. (2014). p. Chapter 4: DNA, Chromosomes and Genomes. ISBN 978-0-8153-4432-2. オリジナルの14 July 2014時点におけるアーカイブ。
- ^ “DNA”. Basic Biology. 5 January 2017時点のオリジナルよりアーカイブ。2015年7月28日閲覧。
- ^ “Uracil” (英語). Genome.gov. 21 November 2019閲覧。
- ^ iGenetics. New York: Benjamin Cummings. (2001). ISBN 0-8053-4553-1
- ^ Principles of Nucleic Acid Structure. New York: Springer-Verlag. (1984). ISBN 0-387-90762-9
- ^ a b Molecular Biology of the Cell (Fourth ed.). New York and London: Garland Science. (2002). ISBN 0-8153-3218-1. OCLC 145080076. オリジナルの1 November 2016時点におけるアーカイブ。
- ^ “Structural diversity of supercoiled DNA”. Nature Communications 6: 8440. (October 2015). Bibcode: 2015NatCo...6.8440I. doi:10.1038/ncomms9440. ISSN 2041-1723. PMC 4608029. PMID 26455586.
- ^ a b c d “Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid”. Nature 171 (4356): 737–38. (April 1953). Bibcode: 1953Natur.171..737W. doi:10.1038/171737a0. ISSN 0028-0836. PMID 13054692. オリジナルの4 February 2007時点におけるアーカイブ。.
- ^ “The dimensions of DNA in solution”. Journal of Molecular Biology 152 (1): 153–61. (October 1981). doi:10.1016/0022-2836(81)90099-1. ISSN 0022-2836. PMID 7338906.
- ^ Arrighi, Frances E.; Mandel, Manley; Bergendahl, Janet; Hsu, T. C. (June 1970). “Buoyant densities of DNA of mammals”. Biochemical Genetics 4 (3): 367–376. doi:10.1007/BF00485753.
- ^ a b c d Biochemistry. W.H. Freeman and Company. (2002). ISBN 0-7167-4955-6
- ^ IUPAC-IUB Commission on Biochemical Nomenclature (CBN) (December 1970). “Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents. Recommendations 1970”. The Biochemical Journal 120 (3): 449–54. doi:10.1042/bj1200449. ISSN 0306-3283. PMC 1179624. PMID 5499957. オリジナルの5 February 2007時点におけるアーカイブ。.
- ^ a b “A glossary of DNA structures from A to Z”. Acta Crystallographica Section D 59 (Pt 4): 620–26. (April 2003). doi:10.1107/S0907444903003251. ISSN 0907-4449. PMID 12657780.
- ^ “RCSB PDB – 1D65: Molecular structure of the B-DNA dodecamer d(CGCAAATTTGCG)2. An examination of propeller twist and minor-groove water structure at 2.2 A resolution.” (英語). www.rcsb.org. 2023年3月27日閲覧。
- ^ “Base-stacking and base-pairing contributions into thermal stability of the DNA double helix”. Nucleic Acids Research 34 (2): 564–74. (2006). doi:10.1093/nar/gkj454. ISSN 0305-1048. PMC 1360284. PMID 16449200.
- ^ Molecular Biology (4th ed.). Sudbury, Mass.: Jones and Barlett Learning. (2012). ISBN 978-0-7637-8663-2
- ^ “Watson-Crick Structure of DNA”. Memorial University of Newfoundland (1953年). 19 July 2016時点のオリジナルよりアーカイブ。13 July 2016閲覧。
- ^ “Modified oligonucleotides: synthesis and strategy for users”. Annual Review of Biochemistry 67: 99–134. (1998). doi:10.1146/annurev.biochem.67.1.99. ISSN 0066-4154. PMID 9759484.
- ^ “Pyrimidines. CIII. The discovery of 5-methylcytosine in tuberculinic acid, the nucleic acid of the tubercle bacillus.”. Journal of the American Chemical Society 47: 2838–44. (1925). doi:10.1021/ja01688a030. ISSN 0002-7863.
- ^ “Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses”. Chemical Reviews 116 (20): 12655–12687. (October 2016). doi:10.1021/acs.chemrev.6b00114. ISSN 0009-2665. PMID 27319741.
- ^ “Epigenetics of Modified DNA Bases: 5-Methylcytosine and Beyond”. Frontiers in Genetics 9: 640. (2018). doi:10.3389/fgene.2018.00640. ISSN 1664-8021. PMC 6305559. PMID 30619465.
- ^ “Non-canonical Bases in the Genome: The Regulatory Information Layer in DNA”. Angewandte Chemie 57 (16): 4296–4312. (April 2018). doi:10.1002/anie.201708228. PMID 28941008.
- ^ “Crystal structure analysis of a complete turn of B-DNA”. Nature 287 (5784): 755–58. (October 1980). Bibcode: 1980Natur.287..755W. doi:10.1038/287755a0. PMID 7432492.
- ^ a b “Protein-DNA recognition”. Annual Review of Biochemistry 53: 293–321. (1984). doi:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ^ “A historical account of Hoogsteen base-pairs in duplex DNA”. Biopolymers 99 (12): 955–68. (2013). doi:10.1002/bip.22334. PMC 3844552. PMID 23818176.
- ^ “Mechanical stability of single DNA molecules”. Biophysical Journal 78 (4): 1997–2007. (April 2000). Bibcode: 2000BpJ....78.1997C. doi:10.1016/S0006-3495(00)76747-6. PMC 1300792. PMID 10733978.
- ^ “A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques”. Proceedings of the National Academy of Sciences of the United States of America 96 (14): 7853–58. (July 1999). Bibcode: 1999PNAS...96.7853C. doi:10.1073/pnas.96.14.7853. PMC 22151. PMID 10393911.
- ^ “Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA”. Molecular Microbiology 16 (5): 817–24. (June 1995). doi:10.1111/j.1365-2958.1995.tb02309.x. PMID 7476180.
- ^ “Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern”. Biochemistry 43 (51): 15996–6010. (December 2004). doi:10.1021/bi048221v. PMID 15609994. オリジナルの10 June 2007時点におけるアーカイブ。.
- ^ a b “On the length, weight and GC content of the human genome.”. BMC Res Notes 12 (1): 106. (2019). doi:10.1186/s13104-019-4137-z. PMC 6391780. PMID 30813969.
- ^ “The DNA sequence and biological annotation of human chromosome 1”. Nature 441 (7091): 315–21. (May 2006). Bibcode: 2006Natur.441..315G. doi:10.1038/nature04727. PMID 16710414.
- ^ “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–465. (April 1981). Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
- ^ “Untitled”. 2011年8月13日時点のオリジナルよりアーカイブ。2012年6月13日閲覧。
- ^ a b c “Organization of multiple nucleoids and DNA molecules in mitochondria of a human cell”. Experimental Cell Research 196 (1): 137–140. (September 1991). doi:10.1016/0014-4827(91)90467-9. PMID 1715276.
- ^ “Mitochondria in oocyte aging: current understanding”. Facts, Views & Vision in ObGyn 9 (1): 29–38. (March 2017). PMC 5506767. PMID 28721182.
- ^ Designation of the two strands of DNA Archived 24 April 2008 at the Wayback Machine. JCBN/NC-IUB Newsletter 1989. Retrieved 7 May 2008
- ^ “Non-coding RNAs: hope or hype?”. Trends in Genetics 21 (5): 289–97. (May 2005). doi:10.1016/j.tig.2005.03.007. PMID 15851066.
- ^ “Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns”. Journal of Cellular Biochemistry 93 (4): 664–71. (November 2004). doi:10.1002/jcb.20252. PMID 15389973.
- ^ “Overlapping genes in vertebrate genomes”. Computational Biology and Chemistry 29 (1): 1–12. (February 2005). doi:10.1016/j.compbiolchem.2004.12.006. PMID 15680581.
- ^ “Properties of overlapping genes are conserved across microbial genomes”. Genome Research 14 (11): 2268–72. (November 2004). doi:10.1101/gr.2433104. PMC 525685. PMID 15520290.
- ^ “Diversity of coding strategies in influenza viruses”. Trends in Genetics 7 (8): 261–66. (August 1991). doi:10.1016/0168-9525(91)90326-L. PMC 7173306. PMID 1771674.
- ^ “DNA mechanics”. Annual Review of Biomedical Engineering 7: 21–53. (2005). doi:10.1146/annurev.bioeng.6.062403.132016. PMID 16004565. オリジナルの1 March 2019時点におけるアーカイブ。.
- ^ a b “DNA topoisomerases: structure, function, and mechanism”. Annual Review of Biochemistry 70: 369–413. (2001). doi:10.1146/annurev.biochem.70.1.369. PMID 11395412.
- ^ a b “Cellular roles of DNA topoisomerases: a molecular perspective”. Nature Reviews Molecular Cell Biology 3 (6): 430–40. (June 2002). doi:10.1038/nrm831. PMID 12042765.
- ^ “Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies”. Journal of Biomolecular Structure & Dynamics 6 (2): 299–309. (October 1988). doi:10.1080/07391102.1988.10507714. PMID 2482766.
- ^ *“The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content”. Acta Crystallogr 6 (8–9): 673–77. (6 March 1953). doi:10.1107/S0365110X53001939. オリジナルの9 January 2016時点におけるアーカイブ。.
- “The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function”. Acta Crystallogr 6 (8–9): 678–85. (1953). doi:10.1107/S0365110X53001940. オリジナルの2017-06-29時点におけるアーカイブ。.
- ^ a b “Molecular configuration in sodium thymonucleate”. Nature 171 (4356): 740–41. (April 1953). Bibcode: 1953Natur.171..740F. doi:10.1038/171740a0. PMID 13054694. オリジナルの3 January 2011時点におけるアーカイブ。.
- ^ a b “Molecular structure of deoxypentose nucleic acids”. Nature 171 (4356): 738–40. (April 1953). Bibcode: 1953Natur.171..738W. doi:10.1038/171738a0. PMID 13054693. オリジナルの13 May 2011時点におけるアーカイブ。.
- ^ “Polymorphism of DNA double helices”. Journal of Molecular Biology 143 (1): 49–72. (October 1980). doi:10.1016/0022-2836(80)90124-2. PMID 7441761.
- ^ “Structural Order and Partial Disorder in Biological systems”. Bull. Math. Biol. 42 (4): 137–41. (1980). doi:10.1007/BF02462372.
- ^ Direct analysis of diffraction by matter. Amsterdam – New York: North-Holland Publishers. (1962)
- ^ “X-ray scattering by partially disordered membrane systems”. Acta Crystallogr A 34 (5): 751–53. (1978). Bibcode: 1978AcCrA..34..751B. doi:10.1107/S0567739478001540. オリジナルの14 March 2020時点におけるアーカイブ。 29 August 2019閲覧。.
- ^ “Crystal structures of A-DNA duplexes”. Biopolymers 44 (1): 45–63. (1997). doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#. PMID 9097733.
- ^ “A-form conformational motifs in ligand-bound DNA structures”. Journal of Molecular Biology 300 (4): 819–40. (July 2000). doi:10.1006/jmbi.2000.3690. PMID 10891271.
- ^ “DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles”. Immunological Reviews 184: 286–98. (December 2001). doi:10.1034/j.1600-065x.2001.1840125.x. PMID 12086319.
- ^ “Z-DNA-binding proteins can act as potent effectors of gene expression in vivo”. Proceedings of the National Academy of Sciences of the United States of America 99 (26): 16666–71. (December 2002). Bibcode: 2002PNAS...9916666O. doi:10.1073/pnas.262672699. PMC 139201. PMID 12486233.
- ^ “Arsenic-loving bacteria may help in hunt for alien life”. BBC News. (2 December 2010). オリジナルの3 December 2010時点におけるアーカイブ。 2 December 2010閲覧。
- ^ a b “Arsenic-Eating Bacteria Opens New Possibilities for Alien Life”. (2 December 2010). オリジナルの4 December 2010時点におけるアーカイブ。 2 December 2010閲覧。
- ^ “Arsenic-eating microbe may redefine chemistry of life”. Nature News. (2 December 2010). doi:10.1038/news.2010.645. オリジナルの12 February 2012時点におけるアーカイブ。.
- ^ “'Arsenic-life' Bacterium Prefers Phosphorus after all”. Nature News. (3 October 2012). doi:10.1038/nature.2012.11520.
- ^ “Structure and packing of human telomeric DNA”. ndbserver.rutgers.edu. 2023年5月18日閲覧。
- ^ a b “Identification of a specific telomere terminal transferase activity in Tetrahymena extracts”. Cell 43 (2 Pt 1): 405–13. (December 1985). doi:10.1016/0092-8674(85)90170-9. PMID 3907856.
- ^ a b c “The telomerase reverse transcriptase: components and regulation”. Genes & Development 12 (8): 1073–85. (April 1998). doi:10.1101/gad.12.8.1073. PMID 9553037.
- ^ “Normal human chromosomes have long G-rich telomeric overhangs at one end”. Genes & Development 11 (21): 2801–09. (November 1997). doi:10.1101/gad.11.21.2801. PMC 316649. PMID 9353250.
- ^ a b “Quadruplex DNA: sequence, topology and structure”. Nucleic Acids Research 34 (19): 5402–15. (2006). doi:10.1093/nar/gkl655. PMC 1636468. PMID 17012276.
- ^ “Crystal structure of parallel quadruplexes from human telomeric DNA”. Nature 417 (6891): 876–80. (June 2002). Bibcode: 2002Natur.417..876P. doi:10.1038/nature755. PMID 12050675.
- ^ “Mammalian telomeres end in a large duplex loop”. Cell 97 (4): 503–14. (May 1999). doi:10.1016/S0092-8674(00)80760-6. PMID 10338214.
- ^ “DNA enables nanoscale control of the structure of matter”. Quarterly Reviews of Biophysics 38 (4): 363–71. (November 2005). doi:10.1017/S0033583505004087. PMC 3478329. PMID 16515737.
- ^ “Four new DNA letters double life's alphabet”. Nature 566 (7745): 436. (21 February 2019). Bibcode: 2019Natur.566..436W. doi:10.1038/d41586-019-00650-8. PMID 30809059.
- ^ “Hachimoji DNA and RNA: A genetic system with eight building blocks (paywall)”. Science 363 (6429): 884–887. (22 February 2019). Bibcode: 2019Sci...363..884H. doi:10.1126/science.aat0971. PMC 6413494. PMID 30792304.
- ^ “RNA secondary structure design”. Physical Review E 75 (2): 021920. (February 2007). arXiv:physics/0609135. Bibcode: 2007PhRvE..75b1920B. doi:10.1103/PhysRevE.75.021920. PMID 17358380.
- ^ “Nucleic Acids”. Michigan State University. 30 June 2022閲覧。
- ^ “How To Extract DNA From Anything Living”. University of Utah. 30 June 2022閲覧。
- ^ “Epigenetic regulation of human embryonic stem cells”. Frontiers in Genetics 3: 238. (2012). doi:10.3389/fgene.2012.00238. PMC 3488762. PMID 23133442.
- ^ “Genomic DNA methylation: the mark and its mediators”. Trends in Biochemical Sciences 31 (2): 89–97. (February 2006). doi:10.1016/j.tibs.2005.12.008. PMID 16403636.
- ^ “DNA methylation patterns and epigenetic memory”. Genes & Development 16 (1): 6–21. (January 2002). doi:10.1101/gad.947102. PMID 11782440.
- ^ “Cytosine methylation and DNA repair”. Current Topics in Microbiology and Immunology 301: 283–315. (2006). doi:10.1007/3-540-31390-7_11. ISBN 3-540-29114-8. PMID 16570853.
- ^ “The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain”. Science 324 (5929): 929–30. (May 2009). Bibcode: 2009Sci...324..929K. doi:10.1126/science.1169786. PMC 3263819. PMID 19372393.
- ^ “N6-methyladenine: the other methylated base of DNA”. BioEssays 28 (3): 309–15. (March 2006). doi:10.1002/bies.20342. PMC 2754416. PMID 16479578.
- ^ “beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei”. Cell 75 (6): 1129–36. (December 1993). doi:10.1016/0092-8674(93)90322-H. hdl:1874/5219. PMID 8261512.
- ^ Created from PDB 1JDG Archived 22 September 2008 at the Wayback Machine.
- ^ “Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation”. Biochemistry 42 (30): 9221–26. (August 2003). doi:10.1021/bi034593c. PMID 12885257.
- ^ “Hydroxyl radicals and DNA base damage”. Mutation Research 424 (1–2): 9–21. (March 1999). doi:10.1016/S0027-5107(99)00004-4. PMID 10064846.
- ^ “Oxidative decay of DNA”. The Journal of Biological Chemistry 272 (32): 19633–36. (August 1997). doi:10.1074/jbc.272.32.19633. PMID 9289489.
- ^ “Regulation and mechanisms of mammalian double-strand break repair”. Oncogene 22 (37): 5792–812. (September 2003). doi:10.1038/sj.onc.1206679. PMID 12947387.
- ^ “Unearthing Prehistoric Tumors, and Debate”. The New York Times. (28 December 2010). オリジナルの24 June 2017時点におけるアーカイブ。. "If we lived long enough, sooner or later we all would get cancer."
- ^ “The Preventable Causes of Cancer”. Molecular biology of the cell (4th ed.). New York: Garland Science. (2002). ISBN 0-8153-4072-9. オリジナルの2 January 2016時点におけるアーカイブ。. "A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop."
- ^ “Cancer and aging as consequences of un-repaired DNA damage”. New Research on DNA Damage. New York: Nova Science Publishers. (2008). pp. 1–47. ISBN 978-1-60456-581-2. オリジナルの25 October 2014時点におけるアーカイブ。
- ^ “DNA damage, aging, and cancer”. The New England Journal of Medicine 361 (15): 1475–85. (October 2009). doi:10.1056/NEJMra0804615. PMID 19812404.
- ^ “A review and appraisal of the DNA damage theory of ageing”. Mutation Research 728 (1–2): 12–22. (2011). doi:10.1016/j.mrrev.2011.05.001. PMID 21600302.
- ^ “The genetic toxicology of acridines”. Mutation Research 258 (2): 123–60. (September 1991). doi:10.1016/0165-1110(91)90006-H. PMID 1881402.
- ^ “Mechanism of action in thalidomide teratogenesis”. Biochemical Pharmacology 59 (12): 1489–99. (June 2000). doi:10.1016/S0006-2952(99)00388-3. PMID 10799645.
- ^ “DNA modification by chemical carcinogens”. Pharmacology & Therapeutics 28 (2): 237–72. (1985). doi:10.1016/0163-7258(85)90013-0. PMID 3936066.
- ^ “Intercalators as anticancer drugs”. Current Pharmaceutical Design 7 (17): 1745–80. (November 2001). doi:10.2174/1381612013397113. PMID 11562309.
- ^ “The sequence of the human genome”. Science 291 (5507): 1304–51. (February 2001). Bibcode: 2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ “The bacterial nucleoid: a highly organized and dynamic structure”. Journal of Cellular Biochemistry 96 (3): 506–21. (October 2005). doi:10.1002/jcb.20519. PMID 15988757.
- ^ “Guide to the draft human genome”. Nature 409 (6822): 824–26. (February 2001). Bibcode: 2001Natur.409..824W. doi:10.1038/35057000. PMID 11236998.
- ^ “The C-value enigma in plants and animals: a review of parallels and an appeal for partnership”. Annals of Botany 95 (1): 133–46. (January 2005). doi:10.1093/aob/mci009. PMC 4246714. PMID 15596463.
- ^ “Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project”. Nature 447 (7146): 799–816. (June 2007). Bibcode: 2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346.
- ^ “RCSB PDB – 1MSW: Structural basis for the transition from initiation to elongation transcription in T7 RNA polymerase” (英語). www.rcsb.org. 2023年3月27日閲覧。
- ^ “The role of heterochromatin in centromere function”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 360 (1455): 569–79. (March 2005). doi:10.1098/rstb.2004.1611. PMC 1569473. PMID 15905142.
- ^ “Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22”. Genome Research 12 (2): 272–80. (February 2002). doi:10.1101/gr.207102. PMC 155275. PMID 11827946.
- ^ “Studying genomes through the aeons: protein families, pseudogenes and proteome evolution”. Journal of Molecular Biology 318 (5): 1155–74. (May 2002). doi:10.1016/S0022-2836(02)00109-2. PMID 12083509.
- ^ “Replicative DNA polymerases”. Genome Biology 2 (1): REVIEWS3002. (2001). doi:10.1186/gb-2001-2-1-reviews3002. PMC 150442. PMID 11178285.
- ^ “Roles of Extracellular DNA in Bacterial Ecosystems”. Extracellular Nucleic Acids. Springer. (2010). pp. 25–38. ISBN 978-3-642-12616-1
- ^ “Extracellular nucleic acids”. BioEssays 29 (7): 654–67. (July 2007). doi:10.1002/bies.20604. PMID 17563084.
- ^ “DNA as a nutrient: novel role for bacterial competence gene homologs”. Journal of Bacteriology 183 (21): 6288–93. (November 2001). doi:10.1128/JB.183.21.6288-6293.2001. PMC 100116. PMID 11591672.
- ^ “Extracellular DNA chelates cations and induces antibiotic resistance in Pseudomonas aeruginosa biofilms”. PLOS Pathogens 4 (11): e1000213. (November 2008). doi:10.1371/journal.ppat.1000213. PMC 2581603. PMID 19023416.
- ^ “A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm”. Molecular Microbiology 77 (4): 815–29. (August 2010). doi:10.1111/j.1365-2958.2010.07267.x. PMC 2962764. PMID 20598083.
- ^ “Extracellular DNA required for bacterial biofilm formation”. Science 295 (5559): 1487. (February 2002). doi:10.1126/science.295.5559.1487. PMID 11859186.
- ^ “DNA builds and strengthens the extracellular matrix in Myxococcus xanthus biofilms by interacting with exopolysaccharides”. PLOS ONE 7 (12): e51905. (2012). Bibcode: 2012PLoSO...751905H. doi:10.1371/journal.pone.0051905. PMC 3530553. PMID 23300576.
- ^ “Recent advances in the prenatal interrogation of the human fetal genome”. Trends in Genetics 29 (2): 84–91. (February 2013). doi:10.1016/j.tig.2012.10.013. PMC 4378900. PMID 23158400.
- ^ “Investigating the potential use of environmental DNA (eDNA) for genetic monitoring of marine mammals”. PLOS ONE 7 (8): e41781. (2012). Bibcode: 2012PLoSO...741781F. doi:10.1371/journal.pone.0041781. PMC 3430683. PMID 22952587.
- ^ “Researchers Detect Land Animals Using DNA in Nearby Water Bodies”. 2020年5月24日閲覧。
- ^ “Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome”. Cellular and Molecular Life Sciences 54 (12): 1350–64. (December 1998). doi:10.1007/s000180050259. PMID 9893710.
- ^ “The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin”. Molecular Microbiology 56 (4): 858–70. (May 2005). doi:10.1111/j.1365-2958.2005.04598.x. PMID 15853876.
- ^ “Crystal structure of the nucleosome core particle at 2.8 A resolution”. Nature 389 (6648): 251–60. (September 1997). Bibcode: 1997Natur.389..251L. doi:10.1038/38444. PMID 9305837.
- ^ “Translating the histone code”. Science 293 (5532): 1074–80. (August 2001). doi:10.1126/science.1063127. PMID 11498575. オリジナルの8 August 2017時点におけるアーカイブ。.
- ^ “Nucleosome Assembly and Remodeling”. Protein Complexes that Modify Chromatin. Current Topics in Microbiology and Immunology. 274. (2003). pp. 1–22. doi:10.1007/978-3-642-55747-7_1. ISBN 978-3-540-44208-0. PMID 12596902
- ^ “HMG1 and 2: architectural DNA-binding proteins”. Biochemical Society Transactions 29 (Pt 4): 395–401. (August 2001). doi:10.1042/BST0290395. PMID 11497996.
- ^ “HMG domain proteins: architectural elements in the assembly of nucleoprotein structures”. Trends in Genetics 10 (3): 94–100. (March 1994). doi:10.1016/0168-9525(94)90232-1. PMID 8178371.
- ^ “Replication protein A (RPA): the eukaryotic SSB”. Critical Reviews in Biochemistry and Molecular Biology 34 (3): 141–80. (1999). doi:10.1080/10409239991209255. PMID 10473346.
- ^ “RCSB PDB – 1LMB: Refined 1.8 Å crystal structure of the lambda repressor-operator complex” (英語). www.rcsb.org. 2023年3月27日閲覧。
- ^ “Mediator of transcriptional regulation”. Annual Review of Biochemistry 69: 729–49. (2000). doi:10.1146/annurev.biochem.69.1.729. PMID 10966474.
- ^ “Biological control through regulated transcriptional coactivators”. Cell 119 (2): 157–67. (October 2004). doi:10.1016/j.cell.2004.09.037. PMID 15479634.
- ^ “A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells”. Proceedings of the National Academy of Sciences of the United States of America 100 (14): 8164–69. (July 2003). Bibcode: 2003PNAS..100.8164L. doi:10.1073/pnas.1332764100. PMC 166200. PMID 12808131.
- ^ “RCSB PDB – 1RVA: Mg2+ binding to the active site of EcoRV endonuclease: a crystallographic study of complexes with substrate and product DNA at 2 Å resolution” (英語). www.rcsb.org. 2023年3月27日閲覧。
- ^ “Biology of DNA restriction”. Microbiological Reviews 57 (2): 434–50. (June 1993). doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ a b “Structural and mechanistic conservation in DNA ligases”. Nucleic Acids Research 28 (21): 4051–58. (November 2000). doi:10.1093/nar/28.21.4051. PMC 113121. PMID 11058099.
- ^ “Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism”. Biochemical Society Transactions 33 (Pt 6): 1465–70. (December 2005). doi:10.1042/BST20051465. PMID 16246147.
- ^ “Unraveling DNA helicases. Motif, structure, mechanism and function”. European Journal of Biochemistry 271 (10): 1849–63. (May 2004). doi:10.1111/j.1432-1033.2004.04094.x. PMID 15128295.
- ^ “Polymerase structures and function: variations on a theme?”. Journal of Bacteriology 177 (22): 6321–29. (November 1995). doi:10.1128/jb.177.22.6321-6329.1995. PMC 177480. PMID 7592405.
- ^ “Eukaryotic DNA polymerases”. Annual Review of Biochemistry 71: 133–63. (2002). doi:10.1146/annurev.biochem.71.090501.150041. PMID 12045093. オリジナルの26 January 2021時点におけるアーカイブ。.
- ^ “Cellular DNA replicases: components and dynamics at the replication fork”. Annual Review of Biochemistry 74: 283–315. (2005). doi:10.1146/annurev.biochem.73.011303.073859. PMID 15952889.
- ^ a b “The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention”. FASEB Journal 8 (8): 497–503. (May 1994). doi:10.1096/fasebj.8.8.7514143. PMID 7514143.
- ^ “Multi-protein complexes in eukaryotic gene transcription”. Plant Molecular Biology 50 (6): 925–47. (December 2002). doi:10.1023/A:1021258713850. PMID 12516863.
- ^ “RCSB PDB – 1M6G: Structural Characterisation of the Holliday Junction TCGGTACCGA” (英語). www.rcsb.org. 2023年3月27日閲覧。
- ^ “Chromosome territories, nuclear architecture and gene regulation in mammalian cells”. Nature Reviews Genetics 2 (4): 292–301. (April 2001). doi:10.1038/35066075. PMID 11283701.
- ^ “An integrated view of protein evolution”. Nature Reviews Genetics 7 (5): 337–48. (May 2006). doi:10.1038/nrg1838. PMID 16619049.
- ^ “The role of double-strand break repair – insights from human genetics”. Nature Reviews Genetics 7 (1): 45–54. (January 2006). doi:10.1038/nrg1746. PMID 16369571.
- ^ “Mammalian Rad51 protein: a RecA homologue with pleiotropic functions”. Biochimie 79 (9–10): 587–92. (October 1997). doi:10.1016/S0300-9084(97)82007-X. PMID 9466696.
- ^ “Clarifying the mechanics of DNA strand exchange in meiotic recombination”. Nature 442 (7099): 153–58. (July 2006). Bibcode: 2006Natur.442..153N. doi:10.1038/nature04885. PMC 5607947. PMID 16838012.
- ^ “The RuvABC resolvasome”. European Journal of Biochemistry 269 (22): 5492–501. (November 2002). doi:10.1046/j.1432-1033.2002.03250.x. PMID 12423347.
- ^ “The antiquity of RNA-based evolution”. Nature 418 (6894): 214–21. (July 2002). Bibcode: 2002Natur.418..214J. doi:10.1038/418214a. PMID 12110897.
- ^ “Prebiotic chemistry and the origin of the RNA world”. Critical Reviews in Biochemistry and Molecular Biology 39 (2): 99–123. (2004). doi:10.1080/10409230490460765. PMID 15217990.
- ^ “Ribozymes. Making copies in the RNA world”. Science 292 (5520): 1278a–1278. (May 2001). doi:10.1126/science.292.5520.1278a. PMID 11360970.
- ^ “What is the optimum size for the genetic alphabet?”. Proceedings of the National Academy of Sciences of the United States of America 89 (7): 2614–18. (April 1992). Bibcode: 1992PNAS...89.2614S. doi:10.1073/pnas.89.7.2614. PMC 48712. PMID 1372984.
- ^ “Instability and decay of the primary structure of DNA”. Nature 362 (6422): 709–15. (April 1993). Bibcode: 1993Natur.362..709L. doi:10.1038/362709a0. PMID 8469282.
- ^ “Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal”. Nature 407 (6806): 897–900. (October 2000). Bibcode: 2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666.
- ^ “Geologically ancient DNA: fact or artefact?”. Trends in Microbiology 13 (5): 212–20. (May 2005). doi:10.1016/j.tim.2005.03.010. PMID 15866038.
- ^ “Curiously modern DNA for a "250 million-year-old" bacterium”. Journal of Molecular Evolution 54 (1): 134–37. (January 2002). Bibcode: 2002JMolE..54..134N. doi:10.1007/s00239-001-0025-x. PMID 11734907.
- ^ “Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases”. Proceedings of the National Academy of Sciences of the United States of America 108 (34): 13995–98. (August 2011). Bibcode: 2011PNAS..10813995C. doi:10.1073/pnas.1106493108. PMC 3161613. PMID 21836052.
- ^ “NASA Researchers: DNA Building Blocks Can Be Made in Space”. NASA (8 August 2011). 23 June 2015時点のオリジナルよりアーカイブ。10 August 2011閲覧。
- ^ ScienceDaily Staff (9 August 2011). “DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests”. ScienceDaily. 5 September 2011時点のオリジナルよりアーカイブ。9 August 2011閲覧。
- ^ “NASA Ames Reproduces the Building Blocks of Life in Laboratory”. NASA (3 March 2015). 5 March 2015時点のオリジナルよりアーカイブ。5 March 2015閲覧。
- ^ “World's oldest DNA sequenced from a mammoth that lived more than a million years ago”. CNN News. (17 February 2021) 17 February 2021閲覧。
- ^ “Million-year-old mammoth genomes shatter record for oldest ancient DNA – Permafrost-preserved teeth, up to 1.6 million years old, identify a new kind of mammoth in Siberia.”. Nature 590 (7847): 537–538. (17 February 2021). Bibcode: 2021Natur.590..537C. doi:10.1038/d41586-021-00436-x. ISSN 0028-0836. PMID 33597786.
- ^ “Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells”. Cell 9 (4 PT 2): 695–705. (December 1976). doi:10.1016/0092-8674(76)90133-1. PMID 189942.
- ^ “Transgenic animal models in biomedical research”. Target Discovery and Validation Reviews and Protocols. Methods in Molecular Biology. 360. (2007). pp. 163–202. doi:10.1385/1-59745-165-7:163. ISBN 978-1-59745-165-9. PMID 17172731
- ^ “Multigene engineering: dawn of an exciting new era in biotechnology”. Current Opinion in Biotechnology 13 (2): 136–41. (April 2002). doi:10.1016/S0958-1669(02)00297-5. PMC 3481857. PMID 11950565.
- ^ “Plant biotechnology in agriculture”. Biochimie 84 (11): 1105–10. (November 2002). doi:10.1016/S0300-9084(02)00013-5. PMID 12595138.
- ^ “From the crime scene to the courtroom: the journey of a DNA sample”. The Conversation. (29 August 2017). オリジナルの22 October 2017時点におけるアーカイブ。 22 October 2017閲覧。
- ^ “Likelihood ratios for DNA identification”. Proceedings of the National Academy of Sciences of the United States of America 91 (13): 6007–11. (June 1994). Bibcode: 1994PNAS...91.6007C. doi:10.1073/pnas.91.13.6007. PMC 44126. PMID 8016106.
- ^ “Interpreting DNA mixtures”. Journal of Forensic Sciences 42 (2): 213–22. (March 1997). doi:10.1520/JFS14100J. PMID 9068179.
- ^ “Individual-specific 'fingerprints' of human DNA”. Nature 316 (6023): 76–79. (1985). Bibcode: 1985Natur.316...76J. doi:10.1038/316076a0. PMID 2989708.
- ^ “Colin Pitchfork” (2006年12月14日). 14 December 2006時点のオリジナルよりアーカイブ。2023年3月27日閲覧。
- ^ “DNA Identification in Mass Fatality Incidents”. National Institute of Justice (September 2006). 12 November 2006時点のオリジナルよりアーカイブ。2007年1月1日閲覧。
- ^ “Before Birth, Dad's ID” (英語). The New York Times. (2012年6月19日). ISSN 0362-4331. オリジナルの2017年6月24日時点におけるアーカイブ。 2023年3月27日閲覧。
- ^ a b “A DNA enzyme that cleaves RNA”. Chemistry & Biology 1 (4): 223–29. (December 1994). doi:10.1016/1074-5521(94)90014-0. PMID 9383394.
- ^ “DNA-catalyzed sequence-specific hydrolysis of DNA”. Nature Chemical Biology 5 (10): 718–20. (October 2009). doi:10.1038/nchembio.201. PMC 2746877. PMID 19684594.
- ^ “In vitro selection of self-cleaving DNAs”. Chemistry & Biology 3 (12): 1039–46. (December 1996). doi:10.1016/S1074-5521(96)90170-2. PMID 9000012.
- ^ “In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing”. Proceedings of the National Academy of Sciences of the United States of America 112 (19): 5903–08. (May 2015). Bibcode: 2015PNAS..112.5903T. doi:10.1073/pnas.1420361112. PMC 4434688. PMID 25918425.
- ^ Bioinformatics: The Machine Learning Approach. MIT Press. (2001). ISBN 978-0-262-02506-5. OCLC 45951728
- ^ Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press. (15 January 1997). ISBN 978-0-521-58519-4
- ^ “Phylogenomic inference of protein molecular function: advances and challenges”. Bioinformatics 20 (2): 170–79. (January 2004). doi:10.1093/bioinformatics/bth021. PMID 14734307.
- ^ Bioinformatics: Sequence and Genome Analysis (2nd ed.). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. (2004). ISBN 0-87969-712-1. OCLC 55106399
- ^ “Protein nanomachines”. PLOS Biology 2 (3): E73. (March 2004). doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
- ^ “Folding DNA to create nanoscale shapes and patterns”. Nature 440 (7082): 297–302. (March 2006). Bibcode: 2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064.
- ^ “Self-assembly of a nanoscale DNA box with a controllable lid”. Nature 459 (7243): 73–76. (May 2009). Bibcode: 2009Natur.459...73A. doi:10.1038/nature07971. hdl:11858/00-001M-0000-0010-9362-B. PMID 19424153.
- ^ “DNA nanotechnology: a nanomachine goes live”. Nature Nanotechnology 4 (5): 281–82. (May 2009). Bibcode: 2009NatNa...4..281I. doi:10.1038/nnano.2009.101. PMID 19421208.
- ^ “Assembling materials with DNA as the guide”. Science 321 (5897): 1795–99. (September 2008). Bibcode: 2008Sci...321.1795A. doi:10.1126/science.1154533. PMID 18818351.
- ^ “Analysis of aptamer discovery and technology”. Nature Reviews Chemistry 1 (10). (2017). doi:10.1038/s41570-017-0076 30 June 2022閲覧。.
- ^ “Dating branches on the tree of life using DNA”. Genome Biology 3 (1): REVIEWS0001. (2002). doi:10.1186/gb-2001-3-1-reviews0001. PMC 150454. PMID 11806830.
- ^ “DNA as a digital information storage device: hope or hype?”. 3 Biotech 8 (5): 239. (May 2018). doi:10.1007/s13205-018-1246-7. PMC 5935598. PMID 29744271.
- ^ “Trends to store digital data in DNA: an overview”. Molecular Biology Reports 45 (5): 1479–1490. (October 2018). doi:10.1007/s11033-018-4280-y. PMID 30073589.
- ^ “Ueber die chemische Zusammensetzung der Eiterzellen [On the chemical composition of pus cells]” (ドイツ語). Medicinisch-chemische Untersuchungen 4: 441–60. (1871). "[p. 456] Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend. (Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ("Nuclein"))"
- ^ “Discovering DNA: Friedrich Miescher and the early years of nucleic acid research”. Human Genetics 122 (6): 565–81. (January 2008). doi:10.1007/s00439-007-0433-0. PMID 17901982.
- ^ See:
- “Ueber Nucleïn der Hefe [On nuclein in yeast]” (ドイツ語). Zeitschrift für physiologische Chemie 3: 284–91. (1879).
- “Ueber Nucleïn der Hefe II [On nuclein in yeast, Part 2]” (ドイツ語). Zeitschrift für physiologische Chemie 4: 290–95. (1880).
- “Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich [On the distribution of hypoxanthins in the animal and plant kingdoms]” (ドイツ語). Zeitschrift für physiologische Chemie 5: 267–71. (1881).
- (ドイツ語) Untersuchungen über die Nucleine und ihre Spaltungsprodukte [Investigations into nuclein and its cleavage products]. Strassburg, Germany. (1881). pp. 19
- “Ueber Xanthin und Hypoxanthin [On xanthin and hypoxanthin]”. Zeitschrift für physiologische Chemie 6: 422–31. (1882).
- Albrect Kossel (1883) "Zur Chemie des Zellkerns" Archived 17 November 2017 at the Wayback Machine. (On the chemistry of the cell nucleus), Zeitschrift für physiologische Chemie, 7: 7–22.
- “Weitere Beiträge zur Chemie des Zellkerns [Further contributions to the chemistry of the cell nucleus]” (ドイツ語). Zeitschrift für Physiologische Chemie 10: 248–64. (1886). "On p. 264, Kossel remarked presciently: Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge. (The study of the quantitative relations of the four nitrogenous bases—[and] of the dependence of their quantity on the physiological states of the cell—promises important insights into the fundamental physiological-chemical processes.)"
- ^ “Albrecht Kossel, a biographical sketch”. The Yale Journal of Biology and Medicine 26 (1): 80–97. (September 1953). PMC 2599350. PMID 13103145.
- ^ “Über Inosinsäure” (ドイツ語). Berichte der Deutschen Chemischen Gesellschaft 42: 1198–203. (1909). doi:10.1002/cber.190904201196.
- ^ “Über die Hefe-Nucleinsäure” (ドイツ語). Berichte der Deutschen Chemischen Gesellschaft 42 (2): 2474–78. (1909). doi:10.1002/cber.190904202148.
- ^ “The structure of yeast nucleic acid”. J Biol Chem 40 (2): 415–24. (1919). doi:10.1016/S0021-9258(18)87254-4.
- ^ “The search for the chemical structure of DNA”. Connecticut Medicine 38 (10): 551–52, 554–57. (1974). PMID 4609088.
- ^ Koltsov proposed that a cell's genetic information was encoded in a long chain of amino acids. See:
- Koltsov HK (12 December 1927). Физико-химические основы морфологии [The physical-chemical basis of morphology] (Speech). 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists (ロシア語). Leningrad, U.S.S.R.
- Reprinted in: “Физико-химические основы морфологии [The physical-chemical basis of morphology]” (ロシア語). Успехи экспериментальной биологии (Advances in Experimental Biology) series B 7 (1): ?. (1928).
- Reprinted in German as: “Physikalisch-chemische Grundlagen der Morphologie [The physical-chemical basis of morphology]” (ドイツ語). Biologisches Zentralblatt 48 (6): 345–69. (1928).
- In 1934, Koltsov contended that the proteins that contain a cell's genetic information replicate. See: “The structure of the chromosomes in the salivary glands of Drosophila”. Science 80 (2075): 312–13. (October 1934). Bibcode: 1934Sci....80..312K. doi:10.1126/science.80.2075.312. PMID 17769043. "From page 313: "I think that the size of the chromosomes in the salivary glands [of Drosophila] is determined through the multiplication of genonemes. By this term I designate the axial thread of the chromosome, in which the geneticists locate the linear combination of genes; … In the normal chromosome there is usually only one genoneme; before cell-division this genoneme has become divided into two strands.""
- ^ “The consequences of political dictatorship for Russian science”. Nature Reviews Genetics 2 (9): 723–29. (September 2001). doi:10.1038/35088598. PMID 11533721.
- ^ “The Significance of Pneumococcal Types”. The Journal of Hygiene 27 (2): 113–59. (January 1928). doi:10.1017/S0022172400031879. PMC 2167760. PMID 20474956.
- ^ “Bacterial gene transfer by natural genetic transformation in the environment”. Microbiological Reviews 58 (3): 563–602. (September 1994). doi:10.1128/MMBR.58.3.563-602.1994. PMC 372978. PMID 7968924.
- ^ “Recherches sur la synthese de l'acide thymonucleique pendant le developpement de l'oeuf d'Oursin” (イタリア語). Archives de Biologie 44: 519–76. (1933).
- ^ “Jean Brachet's Cytochemical Embryology: Connections with the Renovation of Biology in France?”. Les sciences biologiques et médicales en France 1920–1950. Cahiers pour I'histoire de la recherche. 2. Paris: CNRS Editions. (1994). pp. 207–20
- ^ See:
- “Some recent developments in the X-ray study of proteins and related structures”. Cold Spring Harbor Symposia on Quantitative Biology 6: 109–21. (1938). doi:10.1101/sqb.1938.006.01.013. オリジナルの14 July 2014時点におけるアーカイブ。.
- “X-ray studies of nucleic acids”. Symposia of the Society for Experimental Biology (1): 66–76. (1947). PMID 20257017. オリジナルの5 July 2014時点におけるアーカイブ。.
- ^ “Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III”. The Journal of Experimental Medicine 79 (2): 137–158. (February 1944). doi:10.1084/jem.79.2.137. PMC 2135445. PMID 19871359.
- ^ “Chemical specificity of nucleic acids and mechanism of their enzymatic degradation”. Experientia 6 (6): 201–209. (June 1950). doi:10.1007/BF02173653. PMID 15421335.
- ^ “Chargaff's Rules: the Work of Erwin Chargaff”. Journal of Biological Chemistry 280 (24): 172–174. (June 2005). doi:10.1016/S0021-9258(20)61522-8.
- ^ “Independent functions of viral protein and nucleic acid in growth of bacteriophage”. The Journal of General Physiology 36 (1): 39–56. (May 1952). doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
- ^ “Pictures and Illustrations: Crystallographic photo of Sodium Thymonucleate, Type B. "Photo 51." May 1952”. scarc.library.oregonstate.edu. 2023年5月18日閲覧。
- ^ In pursuit of the gene: from Darwin to DNA. Cambridge, Mass.: Harvard University Press. (2008). ISBN 978-0-674-02670-4
- ^ “A Proposed Structure For The Nucleic Acids”. Proceedings of the National Academy of Sciences of the United States of America 39 (2): 84–97. (February 1953). Bibcode: 1953PNAS...39...84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ What Is Life?: investigating the nature of life in the age of synthetic biology. Oxford: Oxford University Press. (2009). p. 52. ISBN 978-0-19-538341-6
- ^ “Double Helix of DNA: 50 Years”. Nature Archives. 5 April 2015時点のオリジナルよりアーカイブ。2007年2月15日閲覧。
- ^ “Original X-ray diffraction image”. Oregon State Library. 30 January 2009時点のオリジナルよりアーカイブ。6 February 2011閲覧。
- ^ “The Nobel Prize in Physiology or Medicine 1962”. Nobelprize.org. 2006年12月24日閲覧。
- ^ “Rosalind Franklin's role in DNA discovery gets a new twist”. AP News. (25 April 2023) 25 April 2023閲覧。
- ^ “Untangling Rosalind Franklin's Role in DNA Discovery, 70 Years On – Historians have long debated the role that Dr. Franklin played in identifying the double helix. A new opinion essay argues that she was an "equal contributor."”. The New York Times. (25 April 2023). オリジナルの25 April 2023時点におけるアーカイブ。 26 April 2023閲覧。
- ^ “What Rosalind Franklin truly contributed to the discovery of DNA's structure – Franklin was no victim in how the DNA double helix was solved. An overlooked letter and an unpublished news article, both written in 1953, reveal that she was an equal player.”. Nature 616 (7958): 657–660. (25 April 2023). doi:10.1038/d41586-023-01313-5. PMID 37100935.
- ^ “The double helix and the 'wronged heroine'”. Nature 421 (6921): 407–08. (January 2003). Bibcode: 2003Natur.421..407M. doi:10.1038/nature01399. PMID 12540909. オリジナルの17 October 2016時点におけるアーカイブ。.
- ^ Crick FH (1955). A Note for the RNA Tie Club (PDF) (Speech). Cambridge, England. 2008年10月1日時点のオリジナル (PDF)よりアーカイブ。
- ^ “The Replication of DNA in Escherichia Coli”. Proceedings of the National Academy of Sciences of the United States of America 44 (7): 671–82. (July 1958). Bibcode: 1958PNAS...44..671M. doi:10.1073/pnas.44.7.671. PMC 528642. PMID 16590258.
- ^ “The Nobel Prize in Physiology or Medicine 1968”. Nobelprize.org. 2006年12月24日閲覧。
- ^ “Discovery of DNA structure and function: Watson and Crick.”. Nature Education 1 (1): 100. (2008).
- ^ “Forensic DNA Profiling and Database”. The Malaysian Journal of Medical Sciences 10 (2): 20–26. (2003). PMC 3561883. PMID 23386793.
推薦文献
- 田村隆明『基礎細胞生物学』(第1版第1刷)東京化学同人、2010年。ISBN 978-4-8079-0724-3。
- 武村政春『DNAを操る分子たち』(初版第1刷)技術評論社、2012年。ISBN 978-4-7741-4998-1。
- DNA: the secret of life. New York: Alfred A. Knopf. (2003). ISBN 0-375-41546-7
- Understanding DNA: the molecule & how it works. Amsterdam: Elsevier Academic Press. (2003). ISBN 0-12-155089-3
- 50 years of DNA. Basingstoke: Palgrave Macmillan. (2003). ISBN 1-4039-1479-6
- The Eighth Day of Creation: Makers of the Revolution in Biology (2nd ed.). Cold Spring Harbor Laboratory Press. (1979). ISBN 0-671-22540-5
- The path to the double helix: the discovery of DNA. New York: Dover Publications. (1994). ISBN 0-486-68117-3 First published in October 1974 by MacMillan, with foreword by Francis Crick; the definitive DNA textbook, revised in 1994 with a nine-page postscript.
- “Quiet debut for the double helix”. Nature 421 (6921): 402–05. (January 2003). Bibcode: 2003Natur.421..402O. doi:10.1038/nature01397. PMID 12540907.
- Francis Crick: A Biography. Plainview, N.Y: Cold Spring Harbor Laboratory Press. (2009). ISBN 978-0-87969-798-3
- DNA Science: A First Course. Cold Spring Harbor Press. (2003). ISBN 978-0-87969-636-8
- Francis Crick: discoverer of the genetic code. Ashland, OH: Eminent Lives, Atlas Books. (2006). ISBN 0-06-082333-X
- DNA: A Graphic Guide to the Molecule that Shook the World. Columbia University Press. (2010). ISBN 978-0-231-14271-7
- The Stuff of Life: A Graphic Guide to Genetics and DNA. Hill and Wang. (2009). ISBN 978-0-8090-8947-5
- The Double Helix: A Personal Account of the Discovery of the Structure of DNA. New York: Norton. (1980). ISBN 0-393-95075-1
- DNA: The Secret of Life. Random House. (2004). ISBN 978-0-09-945184-6
- The third man of the double helix the autobiography of Maurice Wilkins. Cambridge, England: University Press. (2003). ISBN 0-19-860665-6
外部リンク
| DNAに関する 図書館収蔵著作物 |
- DNA - Curlie
- DNA binding site prediction on protein
- DNA the Double Helix Game From the official Nobel Prize web site
- DNA under electron microscope
- Dolan DNA Learning Center
- Double Helix: 50 years of DNA, Nature
- Proteopedia DNA
- Proteopedia Forms_of_DNA
- ENCODE threads explorer ENCODE home page at Nature
- Double Helix 1953–2003 National Centre for Biotechnology Education
- Genetic Education Modules for Teachers – DNA from the Beginning Study Guide
- 蛋白質構造データバンク 今月の分子23:DNA(DNA)
- "Clue to chemistry of heredity found". The New York Times, June 1953. First American newspaper coverage of the discovery of the DNA structure
- DNA from the Beginning Another DNA Learning Center site on DNA, genes, and heredity from Mendel to the human genome project.
- The Register of Francis Crick Personal Papers 1938 – 2007 at Mandeville Special Collections Library, University of California, San Diego
- Seven-page, handwritten letter that Crick sent to his 12-year-old son Michael in 1953 describing the structure of DNA. See Crick's medal goes under the hammer, Nature, 5 April 2013.
- 『デオキシリボ核酸』 - コトバンク





